کاربردهای میکرواری (قسمت دوم)
دکتر رضا میرنژاد (دانشیار دانشگاه)
دکتر مهدی فصیحی رامندی (استادیار دانشگاه)
زهرا کریمی (مرکز تحقیقات زیست سلول پژوهان گستر)
از مهمترین کاربردهای این فناوری، تجزیه بیان هزاران ژن در شرایط فیزیولوژیکی و محیطی متفاوت است. کاربرد دیگر این روش، مطالعه چندشکلی است که در آن نواحی چند شکل ژنوم پیمایش میشوند تا همبستگی آنها با صفت خاص (مثل بیماری) پیدا شود. بدین ترتیب میتوان ژنهای عامل مستعد کننده ابتلا به بیماری را شناسایی کرد. از این فناوری همچنین برای تعیین و بررسی تنوع ژنتیکی نیز استفاده شده است.
ریزآرایهها معمولاً از قرار دادن نقاط DNA بر روی یک صفحه جامد مانند یک سطح شیشهای ایجاد میشوند. برخلاف روشهای مرسوم مبتنی بر فیلترها یا غشاهای نایلونی که در آن DNA هدف بر روی فیلتر یا غشاء قرار گرفته و سپس با کاوشگر دورگه میشود، در سیستم ریزآرایه کاوشگر بر روی یک صفحه جامد قرار میگیرد و سپس با DNA هدف دورگه میشود. استفاده از سطوح شیشهای صاف برای قرار دادن DNA امکانات زیر را فراهم میسازد:
- قرار دادن مولکولها بهطور موازی در کنار هم
- کوچک کردن اندازه کل فرایند
- استفاده از رنگهای فلورسانس برای شناسایی
- امکان استفاده از اسکنرهای لیزری به دلیل اینکه موادی که بر روی سطح قرار میگیرند به داخل نفوذ نکنند.
با توجه به مطالب فوق هدف در ریزآرایهها میتواند اولیگوها (در فتولیتوگرافی) یا cDNA (در روش شبکهسازی مکانیکی) باشد.
پس از ایجاد اسلایدها، عمل دورگهسازی بین کاوشگر و هدف صورت میگیرد. شرایط واکنش در دورگهسازی در دو روش کاملاً متفاوت است و آمادهسازی نمونهها نیز باید بهطور خاص برای آنها صورت گیرد. نوع اسلاید که برای قرارگیری هدف استفاده میشود، مواردی که روی آنها را میپوشاند، سیستم برچسبزنی نمونهها و نشاندار سازی آنها با فلورسانس متفاوت بوده و باید برای هر سیستمی بهینه شود.
پس از انجام دورگهسازی، عمل شناسایی و سپس تجزیهوتحلیل دادهها انجام میشود. در این مرحله حساس از فرایند، مولکولهای کاوشگر که با مولکولهای هدف پیوند شدهاند، باید شناسایی و بهصورت کمّی ارزیابی شوند. برای شناسایی نقاط، اسلایدها معمولاً توسط اسکنرهای دو کاناله که بتواند در 543 و 633 نانومتر کار کرده و به ترتیب CY5 و CY3 را برانگیخته کنند، اسکن میشوند. اسلایدها ابتدا بر روی CY5 و سپس CY3 اسکن میشوند؛ زیرا CY5 به شکستگی در مقابل نور حساستر است. دادههای حاصل از این اسکنها بهطور جداگانه ضبط میشوند و سپس ترکیب آنها برای محاسبه میزان نسبی بیان هر ژن و بیان ژنهایی که بهطور متفاوت بیان میگردند، مورداستفاده قرار میگیرد.
برای مقایسه دادههای حاصل از آرایههای مستقل در آزمایشگاههای متفاوت، نیاز به استانداردسازی است. آرایههایی که حاوی هدفهای 50-25 نوکلئوتیدی هستند، یا بهوسیله سطح بیان ژنهای خاص (مثل اکتین که بیان آن با شرایط محیطی تغییر نمیکند) و یا با استفاده از شدت بیان کل در تمام نقاط اسلاید استاندارد میشوند. آرایههایی که محصول PCR هستند نیز نیاز به استانداردسازی دارند، اما روش مورداستفاده کاملاً متفاوت و پیچیده است. بهطورکلی روش استانداردسازی بهکاربرده شده، منعکسکننده هدفی است که بر اساس آن آرایه ایجاد شده است. برای مثال برای بررسی بیان ژنها در سلولهای سرطانی سینه، ترکیبی از ردههای سرطانی میتواند استاندارد خوبی باشد. به نظر میرسد که روشی کاملاً مناسب برای استانداردسازی در موارد مختلف وجود ندارد و در نتیجه مقایسه دادههای حاصل از آزمایشگاههای مختلف مشکل است و باید از فناوریهای دیگر مانند Real time PCR برای تائید دادهها استفاده کرد. استفاده از روشهای مختلف طبقهبندی برای تجزیه دادهها کاملاً متداول است و برای مطالعه بیشتر آنها میتوان به منابع مختلف در زمینه تجزیه دادههای ریزآرایهها مراجعه کرد.
آنالیز ژنوتیپی
تغییر در ردیف DNA دربرگیرنده بیشترین تفاوتهای موجود در داخل و بین گونهها است. مکانیابی و شناسایی این تفاوتهای ژنوتیپی، نخستین مرحله در ایجاد ارتباط بین تفاوت ژنتیکی و فنوتیپی است. طراحی آرایهها برای این منظور آسان است. با در نظر گرفتن یک ردیف مرجع در یک ناحیه، چهار ردیف هدف طراحی میشوند که انواع احتمالات موجود در یک نوکلئوتید را در بر میگیرند. یکی از ردیفها کاملاً مکمل ردیف مرجع است و سه ردیف دیگر در تمام نوکلئوتیدها بهجز یکی شبیه به ردیف مرجع هستند. پس از دورگهسازی با ردیف مرجع کاوشگر مکمل با ردیف مرجع بیشترین شدت فلورسانس را خواهد داشت. در صورت حضور نمونهای حاوی یک جایگزین در ناحیه موردنظر، کاوشگری که حاوی آن باز جانشین شده باشد، بیشترین شدت فلورسانس را خواهد داشت؛ بنابراین چهار هزار دسته چهارتایی از ردیفها برای یافتن تغییرات تک نوکلئوتیدی در هزار باز موردنیاز است. این راهکار بهخوبی میتواند تغییرات کوچک اتفاق افتاده در ردیف مرجع و حتی مکان و نوع جانشینی صورت گرفته را شناسایی کند. البته درصورتیکه چندشکلی به علت تغییرات طول و حذف یا اضافه شدن قطعه بزرگ باشد، این روش مناسب نیست. کاربرد این روش برای شناسایی چندشکلیهای تک نوکلئوتیدی نیز بهخوبی تعریف شده است. تراشههای SNP هماکنون بهصورت تجاری در دسترساند. این آرایهها را همچنین میتوان برای یافتن SNPهای جدید مورداستفاده قرار داد. این راهکارها میتوانند خیلی راحتتر برای ژنوم های کوچک به کار برده شوند.
تكنيكهاي مينياتوري شده، بهشدت در بيوتكنولوژي در حال گسترش ميباشند. تراشههاي زيستي مانند DNA Chips كاربردهاي نوين و فراواني در بيوتكنولوژي پيدا كردهاند. در يكي از اين كاربردها، دانشمندان توانستهاند با استفاده از رشتههاي DNA به توليد تراشههايي دست بزنند كه سرعت پردازش اطلاعات در آنها در مقايسه با حجم كوچك آنها بسيار بيش از تراشههاي معمولي ميباشد. از كاربردهاي ديگر و اصلي تراشههاي زيستي در مورد DNA Chips و DNA Microarray ميباشد.
امروزه تكنيكهاي دورگهسازی بين كاوشگر و توالي هدف (ساترن، نورترن، دات بلات و دات بلات معكوس)، به كمك فناوري نانو در حجم وسيعي تحت عنوان ريزآرايه قابل انجام است.
در اين تكنيك كه در دانشگاه استنفورد ابداع شده است، از يك سطح شيشهاي يا سليكوني به مساحت cm2 6/1 كه به مربعهاي بسيار زيادي تقسيم شده است، استفاده ميشود. در سطح هر كدام از اين مربعها، كاوشگري وجود دارد كه ميتواند به بخشي از DNA ژنومي و يا cDNA متصل شود. طول كاوشگرها 5000-500 باز ميباشد. هر كدام از اين تراشههاي DNA بهصورت يك دات بلات معكوس عمل ميكنند، اما در اینجا تراكم كاوشگرها طوري است كه هزاران قطعه و مولكول مختلف DNA بهطور همزمان بررسي شده و حتي ميتواند بهصورت كمّي اندازهگيري شوند. اندازهگيري كمّي نمونهها در اين روش بهشدت پيغام دريافت شده توسط سيستم آشکارکننده بستگي دارد. اين سيستم آشکارکننده شامل يك رايانه ميباشد كه ميكروسكوپ Confocal را كنترل مينمايد.
اين تكنيك در كشف ژنها و جهشها، در تشخيص بيماريها، در علم فارماكوژنوميك و توكسيكوژنوميك و … مورداستفاده قرار ميگيرد؛ اما دو كاربرد اصلي اين تراشههاي DNA، مطالعه ترانسکریپتوم[1] و شناسايي جهش ژنتيكي ميباشد.
مطالعه بيان ژن
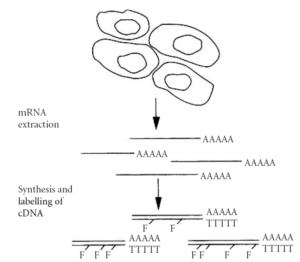
تكنيك ريزآرايه، هنگامیکه براي مطالعه بيان ژن به كار ميرود معادل نورترن بلات معكوس ميباشد. با استفاده از اين تكنيك ميتوان مطالعات كيفي و كمّي روي بيان ژن در يك بافت خاص و در زمان مشخص انجام داد. بدين منظور ريزتراشه به نقاط بسيار زيادي تقسیم شده است كه فاصله بين نقطهها 400 ميكرون ميباشد. در هر كدام از اين نقاط مقدار زيادي كاوشگر اختصاصي يك ژن وجود دارد كه cDNA و يا كاوشگر سنتتيك ميباشد. اين تراشهها با كتابخانه cDNA توسط تكنيك RT-PCR كه بر روي mRNA بافتي انجام شده است، مخلوط شده و دورگه ميشوند. البته شایانذکر است كه اين كتابخانه cDNA با مواد فلورسنته نشاندار شده است (شكل 1).
شكل 1- mRNA از بافت يا سلول كشت دادهشده جدا ميشود. با استفاده از نوكلئوتيد فلورسنته (F)، cDNA نشاندار ساخته ميشود. سپس mRNA حذفشده و cDNA با تراشههاي حاوي آرايههايي از اوليگونوكلئوتيد و يا cDNA مخلوط و دورگه میگردد.
تراكم ريزتراشه طوري است كه 260000 اوليگونوكلئوتيد كه براي مطالعه 6000 ژن مخمر به كار ميرود، بر روي سطحي به مساحت cm21/28 قرار دارند. همچنين 8000 ژن سازنده ژنوم گياه Arabidopsis thaliana بهراحتی بر روي يك تراشه قابلبررسی است. بر روي همين يك تراشه، ميزان بيان ژنهاي اين گياه در ساقه، ريشه، برگ و اندامهاي گلزایی بررسیشده و ترانسكريپتوم گياه بهطور كامل تعيين ميشود.
در انسان، از اين تكنيك براي مقايسه سطح بيان ژن در سلولهاي سالم با سلولهاي سرطاني استفاده ميشود. همچنين براي ارزيابي اثرات داروهاي ضد سرطاني نيز ميتوان از اين تكنيك بهره جست.
شناسايي جهش در ژنها
تكنيكهاي اوليه مورداستفاده در تشخيص جهشها، اختصاصي يك جهش بودند، اما شناسايي جهشهاي متعدد موجود در يك ژن، با استفاده از تكنيكهاي اوليه نيازمند صرف هزينه و وقت زيادي است، لذا به يك پروتكل و روشي نياز بود تا چندين جهش موجود در يك ژن بهطور همزمان مورد ارزيابي قرار گيرند.
تكنيكهاي اوليه (مثل ASO[2], ARMS[3], OLA[4]) در بهترين شرايط قادر به شناسايي 30-10 جهش در يك ژن ميباشند. درحالیکه کارایی تراشهها قابلمقایسه با اين تكنيكها نميباشد. ريزتراشهها قادر هستند تا كل ژنوم را اسكن كرده و تمام جهشهاي موجود در ژنها، پروموترها و تواليهاي خاتمه را شناسايي كنند.
كاوشگرهاي اوليگونوكلئوتيدي مورداستفاده در ريزآرايه، فقط براي جهشهاي شناختهشده (نه همه جهشهاي ممكن) طراحي و ساخته شدهاند. بنابراين براي غربالگري ناقلين بيماريهاي مغلوب و تشخيص قبل از تولد اولين حاملگي كاربرد دارد.
[1] Transcriptome
[2] Allele-specific oligonucleotid
[3] Amplification refractory mutation system
[4] Oligonucleotide ligation assay
برای دانلود فایل pdf بر روی لینک زیر کلیک کنید
ورود / ثبت نام