انواع روشهای شناسایی جهشها
دکتر مهدی فصیحی رامندی (عضو هیئت علمی دانشگاه علوم پزشکی بقیها… (عج))
زهرا کریمی (مرکز تحقیقاتی زیست سلول پژوهان تدبیر)
دکتر رضا میرنژاد (استاد دانشگاه)
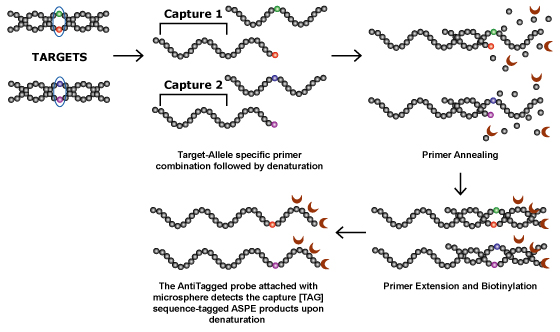
دورگهگيري اختصاصي آلل با استفاده از اوليگونوكلئوتيد
تغييرات آللي (بهعنوان مثال، جهش در يك ژن) اگر باعث تغيير در اندازه يك ژن شوند (از قبيل حذف، اضافه و تكثير تواليهاي تكراري)، بهراحتی با استفاده از الكتروفورز مشخص ميشوند. در اين موارد، محصول PCR بر روي ژل آگاروز يا پلياكريلاميد الكتروفورز شده و اندازه قطعه حاوي جهش تعيين ميشود، همچنين اگر اين تغيير باعث حذف يا ايجاد يك محل شناسايي آنزيم محدودکننده شود، ميتوان محصول PCR را با آنزيم محدودکننده هضم كرد و سپس الكتروفورز نمود. بررسي اندازه اين قطعات بر روي ژل، تغييرات نوكلئوتيدي را نشان خواهد داد.
اگر تغيير بهوجودآمده، هیچکدام از دو حالت فوق نباشد، در نتيجه نميتوان آن را بهطور مستقيم بر روي ژل مشاهده نمود. در اين حالت تكنيك دورگهگيري اختصاصي آلل با استفاده از اوليگونوكلئوتيد[1] (ASO) ميتواند مفيد واقع شود.
اين تكنيك ميتواند بين دو قطعه هدف (با اختلاف حتي يك باز) تمايز قائل شود. با استفاده از PCR قطعه موردنظر تكثير شده و با فلوروفور نشاندار ميشود، از طرف ديگر كاوشگر اوليگونوكلئوتيدي موردنظر بر روي يك سطح، تثبيت ميشود. اين قطعه نشاندار، پس از واسرشت شدن، با كاوشگر مجاور شده و بر اساس تشكيل يا عدم تشكيل دورگه[2] و اندازهگيري شدت فلورسنس، ميتوان به وجود و يا عدم وجود SNP در قطعه موردنظر پي برد.
كاوشگر ASO، تکرشتهای بوده و طول آن بهطور متوسط 20 نوكلئوتيد است. در اين تكنيك، توالي قطعه مورد بررسي و همچنين نوع جهش موجود در آن از قبل شناخته شده و بر اساس نوع اين جهش، كاوشگر طراحي ميشود.
اين كاوشگرها بر اساس اتصال يا عدم اتصال به محصول PCR، وجود حتي يك جهش در ژن را نيز تشخيص ميدهند. در اين تكنيك، شرايط دورگهگيري و شستشو طوري طراحي شده است كه در صورت وجود حتي يك باز ناجور، كاوشگر به محصول PCR متصل نشود.
اين تكنيك محدوديتهايي را نيز به همراه دارد كه شامل موارد زير است:
- اين تكنيك فقط جهشهايي را تشخيص ميدهد كه قبلاً شناخته شدهاند.
- از آنجایی كه كاوشگر براي قطعه حاوي يك جهش خاص طراحي شده است، لذا عدم اتصال كاوشگر به معناي عدم وجود جهش در آن ژن نيست، بلكه فقط بيان ميدارد كه جهش موردنظر در ژن وجود ندارد. كاوشگر فقط 20 نوكلئوتيد از ژن را بررسي ميكند و ممكن است جهش در ديگر نقاط ژن وجود داشته باشد.
در صورتي كه قطعه مورد بررسي فقط يك ژن يا يك توالي بوده و ميزان پليمورفيسم آن كم باشد (يك يا چند جهش)، تكنيك ASO آسان، ارزان، قابل اعتماد و سريع است، اما اگر ميزان تغييرات ژني زياد شود، اين تكنيك گران و ناكارآمد ميشود، زيرا در اين صورت بايد به ازاي هر تغيير نوكلئوتيد احتمالي، يك جفت كاوشگر استاندارد (سالم) و موتانت طراحي و ساخته شود، به همين دليل تكنيك Dot blot معكوس به وجود آمده است. در اين تكنيك، چندين كاوشگر اختصاصي براي چندين جهش يك ژن طراحي و روي يك غشاء تثبيت شدهاند. در اینجا محصول PCR نشاندارشده بر روي اين غشا اضافه ميشود، همچنين ميتوان با استفاده از تكنيك Multiplex PCR چندين قطعه از يك ژن را با هم تكثير نموده و تكنيك ASO را براي همه آنها بهصورت همزمان انجام داد.
شکل 1: تصویر کلی از تکنیک دورگهگيري اختصاصي آلل با استفاده از اوليگونوكلئوتيد (ASO)
(برگرفته از سایت http://premierbiosoft.com/primerplex/allele-specific-primer-extension.html)
تکنیک CFLP
روش CFLP[3] یکی از روشهای شناسایی جهشها است. اساس این روش همانند روش SSCP بر پایه تفاوت در ساختار فضایی سهبعدی توالیهای DNA تکرشتهای مختلف است. در این روش، ابتدا dsDNA بهوسیله حرارت واسرشت شده و سپس سریعاً سرد میشود تا ssDNA تشکیل شود. در ساختار فضایی سهبعدی، برحسب توالی ssDNA، در نقاطی پیوندهای هیدروژنی تشکیل شده و ساختارهای دو رشتهای شبه سنجاقسری ایجاد میکنند. در مرحله بعد، به محلول حاوی توالیهای DNA، آنزیم I Cleavage اضافه میشود. این آنزیم DNA را در نقاطی که ساختار تکرشتهای به دو رشتهای تبدیل شده، برش میدهد. در صورتی که در توالی DNA جهش روی داده باشد، آرایش فضایی پیوندهای هیدروژنی تغییر خواهد کرد و در نتیجه در اثر آنزیم I Cleavage، قطعاتی متفاوت با قطعات حاصل از توالی طبیعی ایجاد خواهد شد.
به این پلیمورفیسم، CFLP گفته میشود؛ بهعنوان مثال از کاربردهاي این روش میتوان به شناسایی جهش R538C (تبدیل کد اسیدآمینه آرژنین به سیستئین) در ژن کدکننده آنزیم بیوتینیداز (ژن BTD واقع در ناحیه 3p25)، اشاره کرد. این جهش، یکی از جهشهای شایع در افراد مبتلا به نقص آنزیم بیوتینیداز است. برای شناسایی این جهش به روش CFLP، ابتدا توالیهای اطراف نقطهای که انتظار جهش در آن وجود دارد (توالی حاوی کدون 450 تا 650)، به کمک PCR تکثیر شده و سپس dsDNAهای تکثیرشده به ssDNA تبدیل میشوند. پس از اضافه کردن آنزیم I Cleavage، قطعات ایجادشده الکتروفورز میشوند.
توالیهایی که دارای جهش R538C باشند، توسط آنزیم Cleavage I به 7 قطعه تبدیل میشوند که هریک دارای وزن مولکولی خاصی است، اما توالیهایی که فاقد جهش فوق باشند، به 11 قطعه با وزن مولکولی متفاوت تبدیل میشوند. به این ترتیب، از روی این نوع پلیمورفیسم یا CFLP میتوان به وجود جهش فوق پی برد.
روش آزمايش كوتاه شدن پروتئين (PTT)
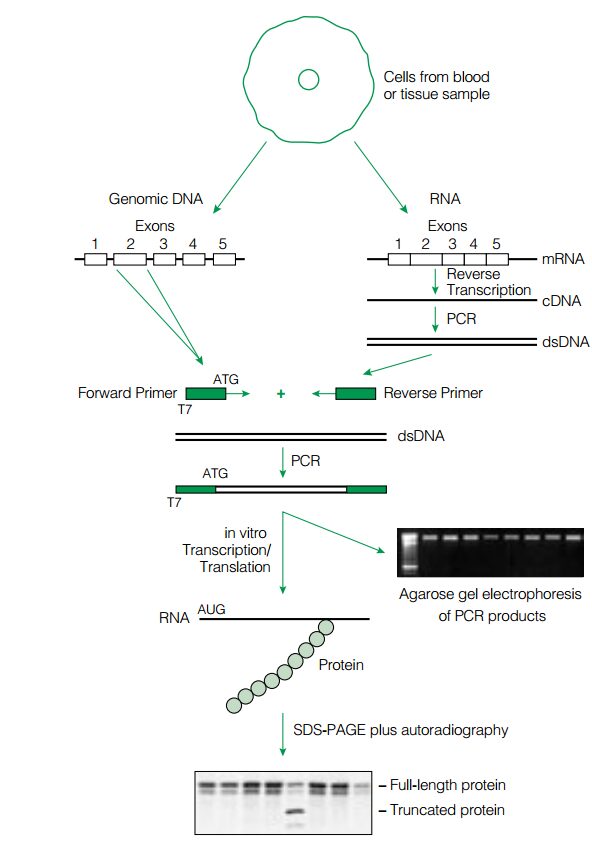
آزمايش [4]PTT براي يافتن جهشهايي طراحي شده است كه طي آن اندازه پروتئين كدشده توسط ژن كوتاه ميشود (جهشهايي مثل ايجاد كدون خاتمه، تغيير چارچوب و حذف قسمتي از ژن بهخصوص در ژنهاي بزرگ).
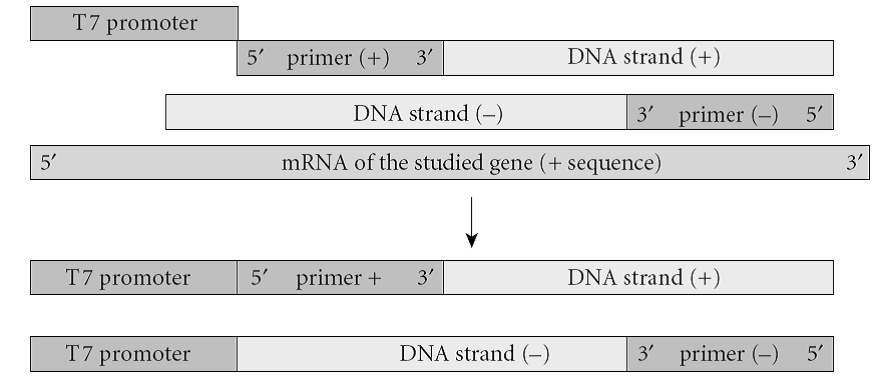
اين روش شامل يك RT-PCR با استفاده از دو پرايمر است؛ پرايمرمنفی موجب ساخته شدن رشته شده و پرايمر مثبت در انتهاي ′5 خود پروموتر فاژ T7 را به همراه دارد. قطعه RNA به روش RT-PCR تكثير شده و بهصورت in vitro ترجمه ميشود. قطعات ساختهشده، به يك سيستم رونويسي/ ترجمه in vitro منتقل ميشوند. اين سيستم كه معمولاً رتيكلوسيت است، حاوي tRNA و اسيدآمينه است، بعضي از اسیدآمینههای اين سيستم توسط راديواكتيو نشاندار شدهاند. بعد از ساخت پپتيد، محلول موجود در لوله بر روي ژل پلياكريلاميد حاوي SDS قرار داده ميشود. الكتروفورز انجام شده و پپتيد بر روي غشاي نيتروسلولزي منتقل ميشود. در اين مرحله وسترنبلات و اتوراديوگرافي انجام شده و اندازه پپتيد حاصل با پپتيد استاندارد و سالم مقايسه ميشود. اين تكنيك بهطور گسترده براي بررسي جهش در ژنهاي (Adenomatous Polyposis Coli )APC، MSH2، MLH1 و ديستروفين بهكار ميرود (شکل 2 و 3).
شكل 2- اساس تكنيك PTT
mRNA ژن موردنظر با كمك پرايمرهاي (+) و (-) و استفاده از تكنيك RT-PCR تكثير میشوند. قطعه بين دو پرايمر كه در اين مرحله حاوي پروموتر T7 است، وارد سيستم رونويسي/ ترجمه ميشود. در اين سيستم، از tRNA حاوي آمينواسيد نشاندار استفاده ميشود. پپتيدهاي ساختهشده، با استفاده از SDS-PAGE، جدا شده و به روي غشاء منتقل ميشوند. با استفاده از وسترن بلات و اتوراديوگرافي، اندازه قطعات تعيين و با حالت استاندارد مقايسه ميشود.
شکل 3: دیاگرام شماتیک از روش PTT
[1]Allele-specific oligonucleotide
[2] Hybrid
[3]Cleavage Fragment Length Polymorphism
[4] Protein truncation test
استخراج و خالصسازی DNA پلاسمیدی از سودوموناس آئروجینوزا
برای دانلود فایل pdf بر روی لینک زیر کلیک کنید
ورود / ثبت نام