تکنیکهای مولکولی بر پایه لیگاز
دکتر مهدی فصیحی رامندی (عضو هیئت علمی دانشگاه علوم پزشکی بقیه ا… (عج))
زهرا کریمی (مرکز تحقیقاتی زیست سلول پژوهان تدبیر)
دکتر رضا میرنژاد (دانشیار دانشگاه)
تکنیکهای لیگازی موجب اتصال دو اولیگونوکلئوتید 20-15 بازی به یکدیگر میشود، در این تکنیکها دو کاوشگر (پرایمر) به مولکول هدف، شامل محصول PCR و یا DNA ژنومی متصل شده و توسط آنزیم DNA لیگاز به هم جوش میخورند. اتصال دو کاوشگر به مولکول هدف در محل جوش[1]، شرط اصلی این واکنش لیگازی است، بهطوری که حتی اگر یک باز در این محل آزاد باشد، این واکنش رخ نخواهد داد؛ به عبارت دیگر برای انجام واکنش، دو کاوشگر باید کاملاً در کنار هم قرار بگیرند.
از این تکنیک برای شناسایی و تشخیص نوکلئوتید جهشیافته در محل جوش و همچنین بهمنظور مطالعه و شناسایی حذف و اضافهها استفاده میگردد. برای تعیین ژنوتیپ DNA به روش لیگاز، مجموعهای از کاوشگرهای اولیگونوکلئوتیدی ساخته میشود. کاوشگرها طوری طراحی میشوند که در محل جایگزینی باز و یا محل جهش در کنار هم قرار گیرند؛ به عنوان مثال برای تعیین ژنوتیپ یک جایگزینی دو آللی، دو کاوشگر اختصاصی آلل (که در نوکلئوتید انتهای ‘3 با هم متفاوت باشند) و یک کاوشگر عمومی طراحی میشود. این کاوشگر عمومی مکمل قطعهای از DNA هدف است که دقیقاً در کنار نوکلئوتید جهشیافته قرار میگیرد. علاوه بر این سه نوع کاوشگر، DNA لیگاز و DNA هدف نیز باید به واکنش اضافه شوند. اگر هنگام هیبریداسیون، اتصال کامل بین بازها رخ دهد، آنزیم لیگاز، دو قطعه کاوشگر را به هم متصل میکند. اگر یک باز متصلنشده وجود داشته باشد، واکنش صورت نمیپذیرد. نتیجه واکنش با الکتروفورز محصولات بر روی ژل مشاهده میشود. روش دیگر برای قرائت نتیجه واکنش بهصورت زیر امکانپذیر است؛ در این سیستم انتهای کاوشگر را با مادهای نشاندار ساخته و با روش ELISA و یا تکنیکهای فلورسنت نتیجه را بررسی میکنند. در این بخش به دو تکنیک تعیین ژنوتیپ DNA اشاره میشود که توسط آنها میتوان جهشهای شناختهشده را تعیین و مطالعه نمود. تکنیک اول روش اتصال اولیگونوکلئوتید[2](OLA) نام داشته و بر پایه رنگسنجی است و تکنیک بعدی واکنش زنجیرهای لیگاز[3](LCR) نام داشته و تکثیر و تعیین ژنوتیپ DNA را امکانپذیر مینماید.
طبقهبندی ليگازها
DNA ليگازها از موجودات مختلفي جدا شدهاند. بر اساس نوع كوفاكتور موردنیاز، اين آنزيمها به دو دستۀ عمده تقسيم ميشوند؛ ليگازهاي جداشده از يوباكترها (باکتریهای حقیقی) به +NAD به عنوان كوفاكتور نياز دارند. درحالیکه ليگازهاي حاصل از يوكاريوتها، آركیباكترها و ویروسها از ATP به عنوان كوفاكتور استفاده ميكنند. در سالهاي اخير اساس مولكولي شناسايي سوبسترا توسط آنزيم ليگاز مشخص شده است. ساختار كريستالي آنزيم DNA ليگاز T7 حاوي ATP، شباهت زيادي با ساختار آنزيم سازنده كلاهك mRNA در ويروس كلورلا[4] دارد. اين امر احتمال تشابه مكانيسم فعاليت اين دو آنزيم را قوت ميبخشد. اين اطلاعات به عنوان پايهاي براي مطالعۀ دقيق و جزئيتر فعاليت و نقش بخشهاي مختلف مولكول آنزيم تلقي ميشود.
مكانيسم اتصال و جوش دادن DNA
واكنش جوش دادن DNA به چهار مرحله تقسيم ميشود كه براي همه ليگازهاي شناختهشده يكسان است؛ ابتدا آنزيم بهطور كووالانت به AMP متصل شده و در اصطلاح شارژ ميشود. اين AMP را از NAD يا ATP گرفته و مولكول NMN و يا پيروفسفات آزاد ميشود. مولكول AMP به گروه آمين اسیدآمینۀ ليزين در موتيف حفاظتشده KXDG آنزيم ليگاز متصل ميگردد. آنزيم آدنيله به محلهاي شكاف در DNA دورشتهاي متصل شده و مولكول AMP را به انتهاي ‘5 فسفريله شكاف منتقل ميكند. در مرحلۀ آخر آنزيم، تشكيل پيوند فسفودياستر بين انتهاي ‘5 شارژ شده و انتهاي ‘OH3 را كاتاليز كرده و AMP آزاد ميشود.
تكنيك اتصال اوليگونوكلئوتيد (OLA)
توانايي آنزيم DNA ليگاز در اتصال اسيد نوكلئيك، توسط بازهاي ناجور موجود در سوبستراها، بهشدت تحت تأثیر قرار ميگيرد. اين خاصيت موجب بوجود آمدن تكنيكهايي شده است كه در آنها، توانايي اتصال و جوش خوردن اوليگونوكلئوتيد، ژنوتيپ نمونۀ مورد آزمايش را تعيين ميكند. مهمترین مزيت اين تكنيك تعيين دقيق و قابل اعتماد آللها و تشخيص SNPها توسط آنزيم ليگاز ميباشد. اين واكنش تحت شرايط استاندارد و با استفاده از دو كاوشگر صورت ميپذيرد. استفاده از دو كاوشگر اختصاصيت واكنش را بالا ميبرد. همچنين اين روش نيازي به PCR ندارد.
در اين تكنيك از دو كاوشگر استفاده ميشود كه اين دو كاوشگر، طي آزمايش بهصورت كووالانت به يكديگر متصل شده و ميتوان نتيجۀ آزمايش را پيگيري نمود. يكي از اين كاوشگرها حاوي بيوتين ميباشد كه از آن براي اتصال و نگه داشتن محصول جوشخورده استفاده ميشود. كاوشگر بعدي نيز داراي فعاليت قابل تشخيص ميباشد. اين تركيب كاوشگرها، براي تشخيصهاي بر پايۀ فاز جامد مناسب هستند. نتيجه آزمايش با چندين روش قابل بررسي است؛ به عنوان مثال ميتوان از تفاوت طول قطعات كاوشگر پس از واكنش ليگاز استفاده كرد. روش متداول براي بررسي نتيجۀ واكنش ليگاز، استفاده از رنگ فلورسنت ميباشد. با استفاده از تكنيك فلورسانس، ميتوان چندين واكنش را بهطور همزمان مورد ارزيابي قرار داد. همچنين ميتوان به يكي از كاوشگرها تواليهاي اختصاصی و بینظیری به عنوان برچسب[5] اضافه نمود و پس از اتمام واكنش، با استفاده از هیبریداسیون و ريزآرايه[6] محصول واكنش جوشخورده را جدا و شناسايي نمود. در حالت فوق غير از ريزآرايه، ميتوان از ذرات[7] نشاندار شده با فلورسانس حاوي توالي مكمل توالي برچسب استفاده نمود. روش ديگر بررسي نتيجۀ واكنش، اندازهگيري FRET بين دو كاوشگر متفاوت ميباشد. در این سیستم میتوان با استفاده از آنتیدیگوکسیژنین و پلیت 96 خانهای تست ELISA طراحی و نتیجه واکنش را با آن مشاهده کرد. در نتیجه، علاوه بر سرعت و حساسیت، سهولت نیز به مزایای این تکنیک اضافه شده و نتایج توسط رایانه قرائت و تفسیر میشود. جهشهای نقطهای جایگزینی، حذف و اضافههای دو آللی و همچنین سیستمهای چند آللی از قبیل HLA را میتوان با این تکنیک تعیین ژنوتیپ نمود.
اصول طراحي تكنيك
بيشترين آنزيمهايي كه در تكنيكهاي بر پايۀ جوش و اتصال به كار گرفته ميشوند آنزيمهاي DNA ليگاز T4 و DNA ليگاز Tth ميباشند. ليگاز T4 از باكتريوفاژ T4 بدست آمده و وابسته به ATP ميباشد. در صورتي كه ليگاز Tth را از يوباكتر ترموس ترموفيلوس[8] جدا میکنند و به +NAD به عنوان كوفاكتور نيازمند است. چند نكتۀ مهم در راهاندازی تكنيكهاي بر پايۀ ليگاز وجود دارد كه بايد موردتوجه قرار گيرد؛ هیبرید كاوشگر و DNA هدف بايد آن قدر بزرگ باشند كه آنزيم ليگاز بتواند بر روي آنها قرار گيرد. آنزيم ليگاز T7 كه شباهت زيادي با ليگاز T4 دارد، براي اتصال به 9-7 نوكلئوتيد از سمت ‘5 فسفات و 5-3 نوكلئوتيد از سمت ‘OH3 نياز دارد. مطالعات نشان داده است كه آنزيم ليگاز Tth به دورگههای شش نوكلئوتيدي و كمتر از آن متصل نميشود، اما ليگاز T4 به اوليگونوكلئوتيدهاي شش نوكلئوتيدي نيز متصل ميگردد. در كل اگر حدود 20 يا بيشتر از 20 نوكلئوتيد در دو طرف شكاف باشد، اين DNA دورگه، سوبستراي مناسبي براي آنزيم ليگاز محسوب ميشود. واكنش اتصال و جوش در دمايي صورت ميگيرد كه همه اوليگونوكلئوتيدها بهطور پايدار به تواليهاي مكمل خود متصل شوند. آنزيم T4 و Tth بازهاي ناجور انتهاي ‘3 را بيشتر از بازهاي ناجور در انتهاي ‘5 شناسايي كرده و افتراق ميدهند، در نتيجه اگر باز ناجور در انتهاي ‘3 شكاف قرار داشته باشد، ميزان اتصال و جوش خوردن دو كاوشگر پايين خواهد آمد. ليگاز Tth بازهاي ناجوري كه چند نوكلئوتيد از شكاف فاصله دارند را نيز شناسايي كرده و افتراق ميدهد.
از تكنيك OLA براي تشخيص واريانتهاي RNA نيز ميتوان بهره جست. البته شرايط بهينۀ اين آزمايش کاملاً متفاوت از شرايط حاكم بر آزمايش بر روي DNA ميباشد.
بهينهسازي شرايط آزمايش
بهمنظور به حداقل رساندن اتصال بازهاي ناجور، بايد از كمترين مقدار آنزيم ليگاز موردنیاز براي اتصال سوبستراي کاملاً متصل استفاده نمود. ليگاز T4 و Tth در نوع كوفاكتور، ميزان دما و pH با همديگر متفاوت هستند. ليگاز T4 در دماي 37 سلسیوس فعال است درحالیکه بهترين دمــا براي فعاليـــــت ليگاز Tth دمای 72-65 درجه سلسیوس ميباشد. هر دو آنزيم به كاتيونهاي دو ظرفيتي نياز دارند. عموماً هر دو نوع آنزيم وابسته به +NAD و ATP از +Mg2 استفاده ميكنند. كاتيونهاي يك ظرفيتي نيز بر روي واكنش تأثیر ميگذارند. افزودن NaCl تا غلظت mM200، موجب حداقل دو برابر شدن قدرت افتراق بازهاي ناجور توسط ليگاز T4 ميشود. افزودن كوفاكتور بيش از مقدار Km، ممكن است كمك كند تا اتصال سوبستراهاي حاوي باز ناجور به حداقل برسد. غلظت بالاي كوفاكتور، مهار آنزيم ليگاز را موجب ميشود، زيرا ATP زودتر از موعد مقرر بر روي آنزيم قرار ميگيرد درحالیکه آنزيم ليگاز به بلوغ مدنظر نرسيده است. در اين حالت آنزيم پس از آدنيلاسيون انتهاي ‘5 از سوبسترا جدا ميشود، در نتيجه آنزيم آدنيله بهسختی ميتواند انتهاي ‘5 آدنيله را جوش دهد. Km ليگاز T4 براي ATP معادل µM14 و Km ليگاز Tth براي كوفاكتور +NAD برابر با nM18/5 ميباشد. در واكنشهاي اتصال و جوش حدود mM1 كوفاكتور اضافه ميكنند. دو روش براي انجام اين تكنيك وجود دارد؛ در يكي از اين روشها از فلورسانس و در ديگري از آنزيم براي بررسي نتيجه استفاده ميشود.
در اين تكنيك يكي از كاوشگرها داراي فعاليت قابل تشخيص بوده و كاوشگر ديگر بهطور كوالان به سطح جامد متصل ميشود، در نتيجه ميتوان نتيجۀ آزمايش را بررسي نمود. واكنشهاي انجامشده با كاوشگرهاي حاوي آنزيم را ميتوان با سيستم ELISA plate readers آناليز نمود. همچنين در مورد فلورسانس، از سه كاوشگر استفاده ميشود. دو تا از كاوشگرها اختصاصي آلل ميباشند. اين دو كاوشگر هرکدام با يك رنگ فلوروفور متفاوت نشاندار شدهاند. كاوشگر سوم نيز يك كاوشگر عمومي و مشترك[9] براي لوكوس مدنظر ميباشد (شکل 1). در نتيجه طي يك واكنش، واريانتهاي مختلف مشخص شده و دقت آزمايش بالا ميرود.
محصول واكنش ليگاز به يك سطح جامد متصل ميشود تا بتوان آن را شستشو داد. چندين نوع سطح جامد حاوي استرپتوآويدين بدين منظور به وجود آمده است.
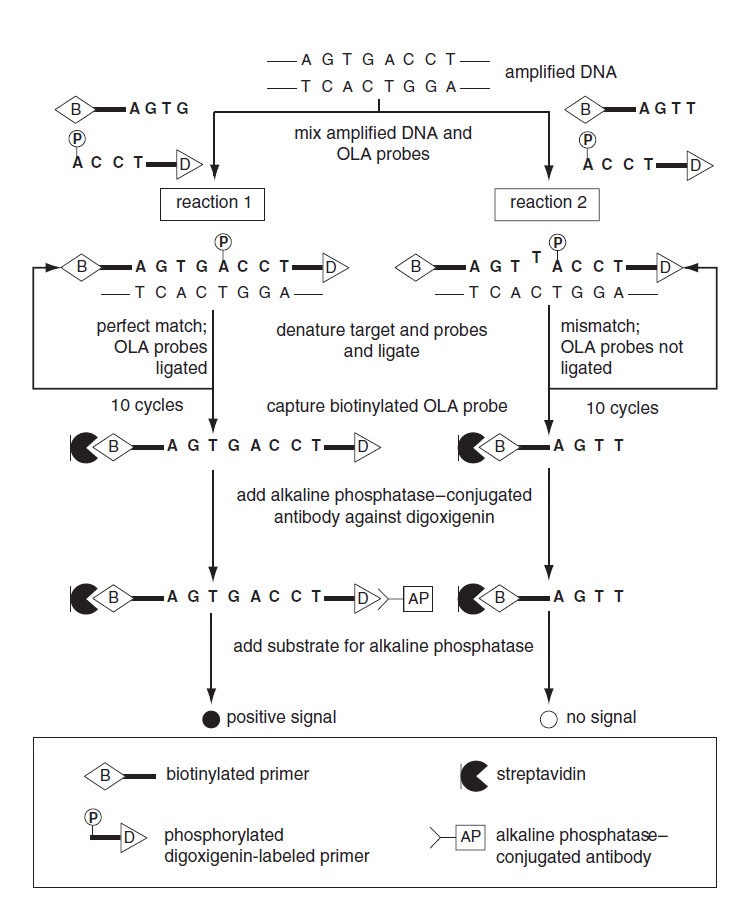
شکل 1: تكنيك OLA توانايي تشخيص واريانتهاي مختلف محصول PCR را داراست. در اين تكنيك از سه كاوشگر و آنزيم ليگاز براي شناسايي SNP استفاده ميشود. در تصوير بالا اوليگونوكلئوتيد شماره سه براي هر دو واريانت استفاده ميگردد (اوليگونوكلئوتيد مشترك). اين اوليگونوكلئوتيد داراي قدرت اتصال به يك سطح جامد ميباشد. اوليگونوكلئوتيدهاي شماره 1 و 2 هرکدام با يك رنگ فلورسنت متفاوت نشاندار شده و اختصاصي واريانتهاي مختلف توالي مورد بررسي ميباشند. البته اگر هرکدام از اين دو اوليگونوكلئوتيدها بهطور جداگانه و در دو واكنش مجزا استفاده شوند ميتوان از يك نوع رنگ فلورسانس استفاده كرد.
مواد موردنیاز:
واكنش زنجيرهاي پليمراز:
- دستگاه ترموسايكلر براي پليت ميكروتيتر
- بافر X1:
50 mM Tris-HCl, pH 8.3
50 mM KCl
12.5µg/mL BSA
1.5 mM MgCl2
200 µM dNTP
Double-distilled water
*براي بهينهسازي واكنش، غلظت MgCl2 را ميتوان در هر مرحله به ميزان mM0/5 تغيير داد.
- اوليگونوكلئوتيد پرايمر
- Taq پليمراز
واكنش اتصال و جوش اوليگونوكلئوتيد:
- محلول اتصال و جوش X2:
18 mM Tris-acetate, pH 7.5
20 mM magnesium acetate
100 mM potassium acetate
400 mM NaCl
2 mM ATP
0.4 µM of T4 DNA ligase
Double-distilled water
2: سه نوع اوليگونوكلئوتيد نشاندار به مقدار fmol600
در مورد اوليگونوكلئوتيدهاي فلورسنته، يكي از آنها بيوتينه و دو اوليگونوكلئوتيد اختصاصي آلل ديگر، بهطور جداگانه با شلاتهاي[10] يوروپيوم[11] و يا تربيوم[12] نشاندار ميشوند.
در سيستمهاي تشخيص بر پايۀ ELISA احتياج به يك كاوشگر بيوتينه و دو كاوشگر اختصاصي آلل كه با دیگوکسیژنین نشاندار شدهاند، ميباشد.
اتصال محصول واكنش ليگاز به سطح جامد:
- شيكر پليت
- پليتهاي كوت شده با استرپتواويدين كه بهصورت زير آماده ميشود:
lµ60 از استرپتواويدين µl100 بر ml در بافر فسفات[13](PBS) را درون چاهكهاي پليت ريخته و به مدت 2 ساعت در 37 درجه سلسیوس قرار دهيد. چاهكها را به مدت حداقل نيم ساعت در دماي اتاق با well /µl200 بر بافر بلوكان بلوكه كنيد.
بافر بلوكان را بهصورت زير تهيه نماييد:
0.5% fat-free dry milk
100 µg/mL denatured salmon sperm DNA
0.02% (w/v) NaN3 in solution B
پليتهاي حاوي بافر بلوكان را ميتوان در دماي ْ 4 سلسیوس نگهداري كرد.
3: محلول A:
1 M NaCl
100 mM Tris-HCl, pH 7.5
0.1% Triton X-100
4: محلول B:
150 mM NaCl
100 mM Tris-HCl, pH 7.5
0.1% Triton X-100
5: محلول دناچوره کننده:
0.1 M NaOH
1 M NaCl
0.1% Triton X-100
شناسايي محصول اتصال در سيستم فلورسنت:
- دستگاه فلورومتر كه توانايي خواندن پليت را دارد.
- شيكر پليت
- محلول تقويت فلورسنت يوروپيوم يا ساماريوم[14]:
0.1 M acetatphthalate, pH 3.2
15 mM 2-naphtoyl trifluoroacetone
50 mM tri-N-octylphosphine oxide
0.1% Triton X-100
4: محلول تقويت تربيوم
100 mM 4-(2,4,6-trimethoxyphenyl)-pyridine-2,6-dicarboxylic acid
1% cetyl trimethyl ammonium bromide in 1.1 M NaHCO3
5: دو برچسب لانتانيد براي حذف يونهاي يوروپيوم و تربيوم. اين برچسبها به اندازهگيري حساس و دقيق مقدار كمي از محلول واكنش (در حد µl0/1) كه حاوي دو رنگ فلورسانس ميباشد، كمك ميكنند.
از فلورفور ساماريوم بهجای تربيوم ميتوان استفاده كرد. فلورسانس حاصل از يون ساماريوم را ميتوان با استفاده از محلول تقويت يوروپيوم تعيين و ثبت كرد، اما فلورسانس حاصل از ساماريوم 10 برابر حساسيت كمتري نسبت به يون يوروپيوم دارد.
شناسايي محصول اتصال با استفاده از سيستم ELISA:
- دستگاه خوانش پليت ELISA[15]
- آنتیبادی ضد دیگوکسیژنین كه با آنزيم آلكالين فسفاتاز كونژوگه شده است. اين آنزيم به نسبت 1:1000 در محلول B كه قبلاً عنوان شد، رقيق ميشود.
- سوبستراي تازۀ آنزيم آلكالين فسفاتاز: يك قرص 5 ميليگرمي پارا نيتروفنيل فسفات را در محلول زير با 9/5 pH حل كنيد:
100 mM diethanolamin
0.5 mM MgCl2
روش كار:
ابتدا قطعۀ موردنظر از DNA ژنومي با PCR تكثير شده، سپس واكنش ليگاز صورت ميپذيرد.
- به هر چاهك پليت ميكروتيتر µl5 از نمونه DNA با غلظت ng/µl 2 در بافر PCR 1X اضافه كنيد.
- µl5 آنزيم Taq پليمراز (U/µl0/1) و µM2 از هر پرايمر را به چاهك اضافه كنيد.
- دستگاه ترموسايكلر را براي 30 سيكل با برنامۀ ̊94، ̊55 و̊ 72 هرکدام براي 30 ثانيه، تنظيم نمایید.
واكنش اتصال و جوش اوليگونوكلئوتيد
بعد از تكثير، محصول واكنش در دستگاه ترموسايكلر دناچوره ميشود. در همين حال، محلول ليگاز حاوي سه كاوشگر و آنزيم ليگاز را درون چاهكهاي پليت ميكروتيتر جديد اضافه ميشود (اين پليتها يا با استرپتواويدين پوشيده شدهاند يا اينكه از ذرات مغناطيسي استفاده ميشود). هنگامي كه دماي محصولات PCR به̊ 37 رسيد، اين محصولات به درون چاهكهاي پليت جديد افزوده ميشوند. در سيستمهاي بر پايۀ ELISA، آللهاي مختلف در دو چاهك مجزا شناسايي ميشوند، اما در سيستم فلورسانس، هر دو آلل در يك چاهك شناسايي ميگردند (شکل 2).
- در سيستم ELISA، محصول PCR را با µl60 آب مقطر رقيق كرده و در دماي ̊ 96 به مدت 5 دقيقه حرارت دهيد تا دناچوره شوند. در سيستم فلورسانس روش كار از مرحلۀ 2 شروع ميشود.
- محصول PCR را در دماي ̊ 96 را به مدت 5 دقيقه حرارت داده تا دناچوره شود. سپس دما را بهسرعت تا ̊ 37كاهش دهيد.
- سریعاً µl10 از محلول ليگاز را به چاهكهاي يك پليت جديد اضافه كنيد.
- µl10 از محصول PCR دناچوره شده را به اين چاهكها منتقل كرده تا حجم نهايي به µl20 برسد.
- پليت را به مدت نيم ساعت در دماي اتاق و يا 15 دقيقه در دماي̊ 37 قرار دهيد تا واكنش اتصال و جوش صورت پذيرد.
اتصال محصول ليگاز به سطح جامد
a) پليتهاي پوشيده با استرپتواويدين
- پليت را دو بار با محلول B شستشو داده تا استرپتواويدينهاي آزاد حذف شوند.
- محصول واكنش ليگاز به اضافۀ µl20 از محلول A را درون پليت بريزيد.
- پليت را به مدت 15 دقيقه در دماي اتاق انكوبه كنيد.
- پليت را دو بار با محلول A و بعد دو بار با محلول دناچوران شستشو دهيد. نهایتاً يك بار ديگر پليت را با محلول A بشویید.
b) ذرات پارامغناطيسي پوشيدهشده با استرپتواويدين
- بعد از واكنش ليگاز، µl20 از محلول A را به واكنش ليگاز اضافه نماييد.
- µl2 از ذرات پارامغناطيسي پوشيدهشده با استرپتواويدين را به محلول واكنش فوق اضافه كنيد.
- پليت واكنش را در دماي اتاق به مدت حداقل 15 دقيقه و بر روي شيكر انكوبه كنيد.
- ذرات را دو بار با محلول A شستشو دهيد. بدين منظور بايد پليت حاوي ذرات را در كنار آهنربا نگاه داريد.
تشخيص و آشکارسازی محصول ليگاز
a) آشکارسازی بر پايۀ ELISA
- µl30 از آنتیبادی آنتيدیگوکسیژنین همراه با بافر بلوكان را به درون چاهك ريخته و نيم ساعت در دماي̊ 37 سلسیوس انكوبه كنيد. پس از انكوباسيون شش بار پليت را با محلول B شستشو دهيد.
- µl50 از محلول سوبسترا را به هر چاهك اضافه كرده و در دماي اتاق انكوبه نماييد تا جذب نوري آن در طولموج nm405 به 2 برسد.
b) آشکارسازی بر پايۀ فلورسانس
- ذرات پارامغناطيسي شستهشده را در چاهكهاي حاوي µl180 از محلول تقويت فلورسانس يوروپيوم بريزيد.
- پليت را به مدت 10 دقيقه بر روي شيكر قرار دهيد.
- پيغام فلورسانس ناشي از يوروپيوم را با فلورومتر تعيين و ثبت نماييد.
- µl20 از محلول تقويت تربيوم را اضافه نماييد.
- پليت را به مدت 10 دقيقه بر روي شيكر قرار دهيد.
- پيغام حاصل از تربيوم را نيز ثبت نماييد.
نكات قابلتوجه:
- تكنيك OLA تحت شرايط استاندارد، توانايي تشخيص و افتراق واريانتهاي مختلف توالي موردنظر را دارد. محققين مختلف ممكن است مقادير متفاوتي از ليگاز را بهكار گيرند تا ميزان بهينۀ آن را بيابند.
- يكي از اوليگونوكلئوتيدها، مكمل توالي مشترك بين دو واريانت ميباشد. اين اوليگونوكلئوتيد دو نوع تغيير را به همراه دارد؛ اين دو تغيير شامل فسفات ‘5 براي اتصال و جوش و بيوتين ‘3 براي اتصال به سطح جامد ميباشد. دو اوليگونوكلئوتيد ديگر هرکدام مكمل يك واريانت از توالي ميباشند. براي تشخيص SNP، توالي اين دو كاوشگر فقط در يك باز ‘3 با هم متفاوت است. اين دو اوليگونوكلئوتيد اختصاصي آلل، در ‘5 خود داراي دیگوکسیژنین (براي سيستم ELISA) و يا تركيبات فلورسنت ميباشند.
- اگر پيغام ضعيفي از آزمايش حاصل شد، تمام مراحل آزمايش بايد مورد بررسي قرار گيرند، به عنوان مثال مرحلۀ اتصال و جوش، با نشانداركردن ‘5 يك اوليگونوكلئوتيد توسط فسفات 32γ و آنزيم پلينوكلئوتيد كيناز بررسي ميشود. محصول اتصال بر روي ژل پلياكريلاميد دناچوران 15% الكتروفورز شده و اتوراديوگرافي ميشود. مرحلۀ اتصال اوليگونوكلئوتيد به سطح جامد، با استفاده از اوليگونوكلئوتيدهاي حاوي دو نوع نشانه[16]، شامل دیگوکسیژنین و بيوتين ارزيابي ميشود. ممكن است اشكال در مرحلۀ پوشش استرپتواويدين موجب ايجاد پيغام ضعيف گردد.
- شدت پيغام حاصل از سيستم ELISA را ميتوان با تغيير نوع سوبستراي آنزيم آلكالين فسفاتاز افزايش داد.
شکل 2: تصویر شماتیک OLA
برای مطالعه یک جایگزینی (جایگزینی G به T در این مثال) دو واکنش OLA بر روی محصول PCR انجام میشود. یک واکنش آلل G (واکنش 1) و واکنش دیگر آلل T (واکنش 2) را شناسایی میکند. هنگامی که کاوشگرها بهطور کامل به DNA هدف متصل شوند، آنزیم لیگاز کاوشگر حاوی بیوتین را به کاوشگر نشاندار شده با دیگوکسیژنین متصل میکند (سمت چپ تصویر)، اما هنگامی که یک نوکلئوتید آزاد وجود داشته باشد واکنش صورت نمیگیرد (سمت راست تصویر). برای مشاهده نتیجه واکنش، کاوشگر حاوی بیوتین به کف پلیت متصل شده و پس از شستشوی پلیت، واکنش ایمونواسی برای دیگوکسیژنین انجام میشود. حضور یا عدم حضور رنگ نمایانگر وضعیت واکنش است. در این مثال، نمونه برای آللG هموزیگوت بوده و رنگ فقط در آن واکنش ظاهر شده است. در نمونههای هموزیگوت، فقط یکی از واکنشها مثبت شده و رنگ ظاهر میشود، اما در نمونههای هتروزیگوت، پیغام مثبت در هر دو واکنش مشاهده میشود.
واکنش زنجیرهای لیگاز
در این سیستم از آنزیم لیگاز بهمنظور اتصال بین اولیگونوکلئوتیدهای مکمل DNA هدف استفاده میشود. نتیجه این روش تکثیر تصاعدی محصول واکنش لیگاز است. همانند تکنیک PCR، در دسترس بودن آنزیم مقاوم به حرارت، موجب سهولت آزمایش شده است. این تکنیک حاوی سه مرحله است: واسرشت شدن DNA الگو، اتصال آغازگر و انجام واکنش اتصال. از آنجا که مطالعه جایگزینی نوکلئوتید، هدف آزمایش است استفاده از تکنیک LCR حساسیت آزمایش را بالا میبرد. اگر از مواد رادیواکتیو بهمنظور نشاندار کردن آغازگر استفاده شود، حساسیت آزمایش افزایش یافته و حدود 200 مولکول DNA هدف را میتوان در نمونه شناسایی کرد، اما اگر این حد از حساسیت نیاز نباشد، میتوان از گزارشگرهای فلورسنته استفاده نموده و با استفاده از دستگاه تعیین توالی DNA که بر اساس فلورسانس عمل میکند، نتیجه واکنش را خواند. همچنین میتوان مشابه تکنیک OLA، از سیستم ELISA برای مشاهده نتیجه واکنش استفاده کرد (شکل 3).
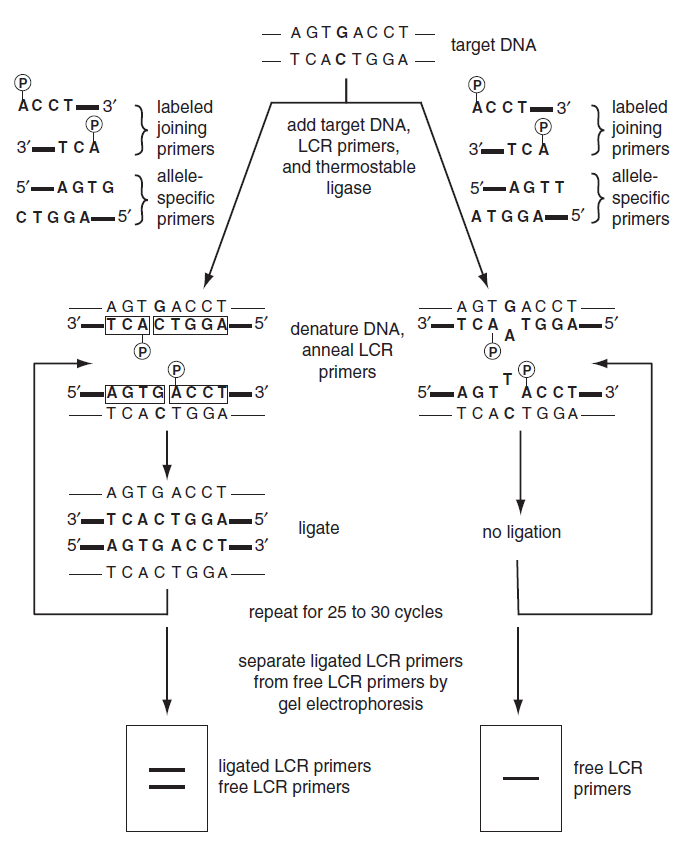
شکل 3: روش اجرای LCR
در این تصویر گروه فسفات با ماده رادیواکتیو نشاندار شده است. چهار آغازگر مورد استفاده در این تکنیک باید طوری طراحی شوند که در انتهای ‘3 خود و در محل اتصال یک نوکلئوتید آزاد باشد (همانطور که در تصویر بهصورت مستطیل آغازگر نشان داده شده است). همچنین بهمنظور جلوگیری از واکنش اتصال مستقل از الگو، میتوان دو نوکلئوتید آزاد به انتهای ‘5 آغازگرهای اختصاصی آلل اضافه نمود.
مواد موردنیاز:
- محلول LCR
20 μl 1M KCl (0.2 M final)
20 μl 10X thermostable ligase buffer (see recipe; 2X final)
20 μl 10 mM NAD+ (Sigma; 2 mM final)
40 μl 0.1% (v/v) Triton X-100 in H2O (0.04% final) Prepare fresh
NAD+ can be stored up to 6 months at −20°C
- آغازگرها: آغازگرهای اختصاصی آلل و آغازگرهای اتصالی نشاندار شده با P32 مکمل هر دو رشته هدف
- 5U آنزیم لیگاز مقاوم به حرارت
- DNA هدف: 10µl از محصول PCR یا 10ng از DNA ژنومی
- روغن معدنی
- فرمامید
- نشانگر اندازه مولکولی DNA
انجام واکنش لیگاز
- محلول واکنش را برای هر یک از آللها تهیه نمایید:
100 μl LCR mix
40 fmol of each of the allele-specific primers for each strand
40 fmol of the 32P-labeled joining primers for each strand
0.5 U thermostable ligase.
هر محلول حاوی دو آغازگر اختصاصی آلل و دو آغازگر اتصالی است.
- DNA مورد آزمایش را به محلول واکنش اضافه نموده و محلول را با 30µl روغن معدنی بپوشانید.
- نمونهها را در دستگاه ترموسایکلر قرار داده و برنامه دمایی زیر را به دستگاه بدهید:
- واسرشت سازی: °94 سلسیوس به مدت 30 ثانیه
- اتصال آغازگر و انجام واکنش اتصال: °65 سلسیوس به مدت 2 دقیقه
- تکرار برنامه فوق به میزان 30-25 چرخه
اگر 30 چرخه واکنش انجام شود، واکنش اتصال زمینهای نیز مشاهده میشود؛ به عبارت دیگر با طولانی شدن مدت واکنش، اتصالات غیراختصاصی آغازگر بهصورت تصاعدی افزایش مییابد.
- تشخیص و شناسایی محصول واکنش با الکتروفورز
- 4µl از محصول واکنش را با 4µl فرمامید مخلوط کرده و به مدت 2 دقیقه در دمای °100 سلسیوس قرار داده تا نمونهها واسرشت شوند. نمونههای واسرشتشده را بر روی ژل 10% پلیاکریل امید ببرید. نشانگر وزن مولکولی نشاندار شده را نیز در کنار نمونه ها قرار داده و با ولتاژ 60W به مدت 2 ساعت الکتروفورز نمایید.
- ژل را خشک نموده و بر روی فیلم رادیولوژی قرار دهید تا محل باندها مشخص شود.
[1] Junction
[2] Oligonucleotide ligation assay
[3] ligase chain reaction
[4] Chlorella
[5] Tag
[6] Microarray
[7] Micro bead
[8] Thermus thermophilus
[9] Common probe
[10] Chelates
[11] Europium
[12] Terbium
[13] Phosphate-buffered saline
[14] Samarium
[15] ELISA plate reader
[16] Label
مقدمهای بر تکنیک واکنش زنجیرهای پلیمراز و انواع آن
مقدمهای بر تکنیک واکنش زنجیرهای پلیمراز و انواع آن
پلیمورفیسم طولی قطعات تکثیرشده (AFLP)
برای دانلود پی دی اف بر روی لینک زیر کلیک کنید
ورود / ثبت نام