مروری برفناوری ریزآرایه (Micro array)
الهام عبدالهی، دانشجوی دکترای تخصصی ایمونولوژی پزشکی دانشکده پزشکی، دانشگاه علوم پزشکی مشهد
امیرعباس ممتازی بروجنی، دانشجوی دکترای تخصصی بیوتکنولوژی پزشکی، دانشکده پزشکی دانشگاه علوم پزشکی مشهد
فتانه توسلیان، دانشجوی دکترای تخصصی ایمونولوژی پزشکی، دانشکده پزشکی، دانشگاه تربیت مدرس
چکیده
واضح است که هزاران ژن و محصولات آنها (مانند RNA و پروتئین) همگی در یک مسیر پیچیده و منظم در بدن یک موجود زنده به فعالیت مشغولند، با اینحال روشهای قدیمی در زیستشناسی مولکولی بر اساس یک ژن در یک آزمایش کار میکنند که در این صورت بدست آوردن تصویر جامع از عملکرد همه ژنها سخت و غیرممکن بنظر میرسد. در چند سال اخیر فنآوری جدیدی، به نام microarray توجه دانشمندان بیولوژی مولکولی و دیگر رشتههای علمی را به خود جلب کرده است. این فنآوری امکان نشان دادن همهی ژنوم یک ارگانیسم را بر روی یک chip فراهم آورده است و لذا به محققین کمک میکند تا بتوانند دید بهتر و گستردهتری از تعامل میان هزاران ژن به طور همزمان داشته باشند. به طور ساده میکرواری عبارت است از فناوری بررسی فعالیت هزاران ژن و یا پروتئین در یک سطح کوچک مانند یک لام میکروسکوپی. هدف از مطالعه حاضر معرفی تکنیک Micro array و زیرمجموعههای متفاوت آن است.
واژههای کلیدی: تکنیک ریزآرایه (Micro array)
مقدمه
تاکنون برای مطالعه خواص، فعالیت و نقش ژنها و پروتئینها و کشف فرایندهای مولکولی درون سلولها و بافتها و همچنین جنبههای مولکولی بیماریها و آسیبهای زیستی اکثراً یک ژن و یا حداکثر چند ژن یا پروتئین بطور خاص و جداگانه مورد بررسی و مطالعه قرار میگرفت. این روشها شامل PCR و انواع روشهای استفاده از نشانگرها، بلاتینگ موسوم به وسترنبلات، نورترنبلات و ساترنبلات و همچنین بررسی القا و فعالیت متفاوت ژنها و پروتئینها و بسیاری از فنون دیگر میشد ولی بررسی همزمان هزاران ژن و پروتئین بسیار دشوار بود. پس از خاتمه طرح ژنوم انسانی و تعیین ردیف کامل ژنوم انسان که شامل ۳ میلیارد نوکلئوتید (واحد تشکیل دهنده ژن) میشد- که در واقع حدود ۳۰ هزار ژن را رمز مینمود- و مطالعه این اطلاعات مشخص شد که مطالعه یک ژن و یا یک پروتئین و کشف یک فرایند بطور جداگانه کمک بسیاری به حل مشکل نمیکند زیرا فرایندهای زیستی بسیار پیچیده است و گاهی دهها و صدها ژن در بروز یک فعالیت نقش دارند، بنابراین با کشف ارتباط بسیار پیچیده بین مولکولهای زیستی و شبکهای از زایندها در ایجاد یک آسیب و یا بروز یک فعالیت مشخص شد که تنها راه امکان بررسی دهها، صدها و بلکه هزاران ژن و پروتئین در سلول سالم و مقایسه آن با سلول آسیبدیده میتواند دانشمندان را در یافتن پاسخ مناسب و درک صحیح از علل بروز عارضه و آسیب و در نهایت تشخیص صحیح و در نتیجه درمان مناسب راهنمایی نماید. در همین مسیر فناوریهای ژنومیک و پروتئومیک کمک بسیاری جهت بررسی فعالیت و تغییرات مجموعه ژنها و پروتئینها نمود که هنوز نیز بعنوان روشهای مفید استفاده میشود، ولی با توسعه این روشها و اطلاع از کاستیهای آنها به علوم و فناوریهای جدیدی نیاز بود که بتواند همزمان کاهش، افزایش و عدم فعالیت دهها، صدها و هزاران ژن و یا پروتئین را بررسی و گزارش نماید. این دانش با ترکیبی از چندین علم و فناوری حاصل شده است.
آرایهی DNA
بكارگيري فناوري ريزآرايه، در سالهاي اخير موجب توليد حجم انبوهي از دادههاي بيان ژني شده است. ريزآرايه ابزاري براي اندازهگيري و كسب اطلاعات از بيان ژنهاست. هر توالي ژني شناخته شده موردنظر به عنوان يك شيشه يا نايلوني (Array) روي يك آرايه (Prob) پروب از بافت يا نمونه خون با رنگهاي mRNA چاپ ميشود. فلورسنت علامتگذاري و پروبها بر روي يك آرايه هيبريد ميشوند. آرایه هیبریداسیون که در بسیاري از موارد، نورترنبلات معکوس نامیده میشود انواع مختلفی دارد. آرایههاي رایجی که استفاده میشود به چهار گروه تقسیمبندي میشوند: درشت آرایه، ریزآرایه، آرایههاي نوکلئوتیدي با تراکم بالا و آرایههاي میکروالکترونیک. نامگذاري روشها در این زمینه بسیار گسترده و متغیر است. در بسیاري از جاها تمامی این روشها تحت عنوان ریزآرایه نامیده میشوند. بهرحال در این تقسیمبندي که توسط فريمن و همکاران در سال 2000 صورت گرفت کلمه آرایه هیبریداسیون بعنوان لفظ کلی در نظر گرفته میشود و لغت ریزآرایه بعنوان زیر مجموعهاي ویژه از آرایه هیبریداسیون میباشد. چهار دسته مذکور در واقع از لحاظ زمینه، تعداد پروب در واحد سطح آرایه (تراکم)، اندازه آرایه و نوع نشانهگذاري متفاوت میباشند. براي ارزیابی قوت و ضعف این روشها باید مفید بودن اینها براي هدفهاي تحقیقات مختلف مشخص شود.
انواع آرایهی DNA
دو نوع آرايهDNA بيشترين كاربرد را دارند:
1- آرایه بر پایه DNA مکمل (DNA complementary spotted)
2- آرايه اليگونوكلئوتيد (DNA complementary spotted) كه به اختصار اليگو گفته ميشود (Oligonucleotide array)
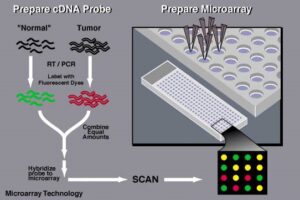
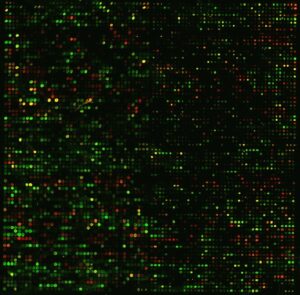
در روش آرايه cDNA هر ژن با یک رشته طولانی (بین 200 تا 500 نوکلئوتید) نشان داده میشود. cDNA از دو نمونه متفاوت بدست ميآيد؛ يكي از نمونهي مورد آزمون و ديگري نمونهي مرجع كه بر روي يك آرايه هيبريد (مخلوط) ميشوند. نمونه آزمون با رنگ فلورسنت قرمز و نمونه مرجع با رنگ سبز علامتگذاري ميشوند، سپس با هدف تحريك رنگهاي فلورسنت در دو طول موج مختلف، آرايه به وسيله اشعه ليزر اسكن ميشود. از هر كدام اين رنگها يك تصوير بوجود ميآيد كه اين تصاوير در كامپيوتر بر روي هم قرار داده ميشود كه حاصل اين كار تراشهاي خواهد بود كه حاوي هزاران لكه رنگي با رنگهاي بسيار متنوع است كه از تركيب دو رنگ قرمز و سبز حاصل شده است. اندازه بيان ژني ميتواند لگاريتم نسبت شدت رنگ قرمز به سبز باشد (شکل 1).
به طور كلى براى تهيه آرايه cDNA باید مطابق مراحل زیر عمل نمود:
- نمونهگيرى
- خالصسازى نمونه و جداسازى mRNA
- متصل كردن cDNA
- انجام رونويسى معكوس و تهيه cDNA
- ريختن محلول رنگهاى فلوئورسنت مانند cy3 روى سطح ريزآرايه كه از قبل توسط توالىهاى ژن موردنظر پوشيده شده است، مدتى صبر ميكنيم تا هيبريداسيون ميان توالىهاى سطح ريزارى انجام گيرد.
- شستشو
- بررسى و پردازش (شکل 2)
شکل 1) آرایه cDNA
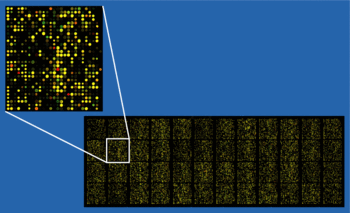
شکل 2) تصاویر حاصله از یک اسلاید ریزآرایه که با مقایسه لکهها و رنگ موجود در آنها میتوان به پروفایل بیان ژنی در دو بافت سالم و بیمار پی برد.
در روش آرايه اليگونوكلئوتيد هر ژن به 16 الي 20 حالت نشان داده ميشود كه هر كدام خود توالي كوتاهي از نوكلئوتيدها از يك PM يا (Perfect Match) هستند و يك جفت كامل قطعه ژن ميباشند، در مقابل اين 20 اليگونوكلئوتيد، 20اليگونوكلئوتيد ديگر وجود دارد كه به جز در باز مركزي، توالي آنها با هم برابر است، كه به اين نوكلئوتيدها غيرجفت MM يا (MisMatch) ميگويند. يك اندازه از بيان ژني به صورت متوسط شدت اختلافات در اين 16 تا 20 حالت ميباشد. ميتوان از نرمافزارهاي STATA ،S-plus و SAS براي بررسي دادههاي ريزآرايه DNA استفاده کرد.
آرایه هیبریداسیون که در بسیاري از موارد نورترنبلاك معکوس نامیده میشود انواع مختلفی دارد. آرایههاي رایجی که استفاده میشود به چهار گروه تقسیمبندي میشوند: درشت آرایه، ریزآرایه، آرایههاي نوکلئوتیدي با تراکم بالا و آرایههاي میکروالکترونیک. نامگذاري روشها در این زمینه بسیار گسترده و متغیر است. در بسیاري از جاها تمامی این روشها تحت عنوان ریزآرایه نامیده میشوند. بهرحال در این تقسیمبندي که توسط فريمن و همکاران در سال 2000 صورت گرفت کلمه آرایه هیبریداسیون بعنوان لفظ کلی در نظر گرفته میشود و لغت ریزآرایه بعنوان زیر مجموعهاي ویژه از آرایه هیبریداسیون میباشد. چهار دسته مذکور در واقع از لحاظ، زمینه، تعداد پروب در واحد سطح آرایه (تراکم)، اندازه آرایه و نوع نشانهگذاري متفاوت میباشند. براي ارزیابی قوت و ضعف این روشها، باید مفید بودن اینها براي هدفهاي تحقیقات مختلف مشخص شود.
ساختن آرایه DNA
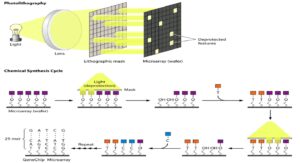
ساختن آرایه DNA به چند روش صورت میگیرد که مهمترین آنها تکنیک ماسک نوری است (photolithography) که به تکنیک Affimetrix هم معروف است. در این روش که به کمک آن پروب (کاوشگر)های کوتاه الیگونوکلئوتیدی سنتز میشود، شامل سنتز در جای پروبهای الیگونوکلئوتیدی بر روی یک ریزآرایه تحت هدایت نور بوده و از طریق آن میتوان بیش از 6 میلیون جمعیت پروب مختلف را بر روی یک سطح به مساحت cm21/7 تولید نمود. هر نوع الیگونوکلئوتید با افزودن پیدرپی مونونوکلئوتیدها به یک مولکول اتصال دهنده ایجاد میشود که این مولکول به یک گروه محافظ و ناپایدار در برابر نور ختم میگردد. از یک ماسک نوری (فوتولیتوگراف) به منظور تشخیص موقعیتهای واقع شونده در معرض منبع نور خارجی بر روی ریزآرایه استفاده می شود. در این موقعیتها، گروههای ناپایدار در برابر نور تخریب شده و سپس یک نوکلئوتید مشخص از طریق پیوند کووالان به جایگاههای اخیراً محافظتزدایی شده اضافه میشود. این روند به صورت متوالی با استفاده از ماسکهای مختلف مربوط به هر یک از سه نوکلئوتید دیگر تکرار میشود تا در نهایت به هر مولکول اتصال دهنده یک نوکلئوتید واحد افزوده گردد.
روند فوق به منظور افزایش طول نوکلئوتیدها به میزان حدود 25 تا 30 نوکلئوتید تکرار میشود. بسته به نوع آرایش منافذ موجود در ماسکهای مورد استفاده در هر مرحله، میتوان توالی الیگونوکلئوتیدی از پیش تعیین شدهای را در موقعیتهای از پیش تعیین شده بر روی ریزآرایه گرد هم آورد (شکل 3).
شکل 3) ساخت آرایه DNA به روش Affimetrix
Miller, etal. Clinical microbiology reviews. 2009
كاربردهاى ريزآرايه DNA
كاربردهاى ريزآرايه DNA عبارتند از:
- بررسى بيان ژن و تغييرات آن در اثر عواملى مانند درمان، عوامل بيماریزا، آسيب سلول
- هيبريدسازى مقايسهاى ژنوم. تعيين محتواى ژنوم موجودات زنده و مقايسه آنها با يكديگر
- شناسايى چندشكلىهاى تكنوكلئوتيدى. با استفاده از اين تكنولوژى مىتوان چندشكلىهاى تكنوكلئوتيدى را در ميان جمعيتهاى مختلف مطالعه كرد. اين مطالعات در زمينههاى مختلفى مىتوانند مؤثر باشند مانند تعيين ژنوتيپ، اندازهگيرى احتمال ابتلا به برخى از بيمارىها، تخمين جهشهاى سلولهاى زايا و جهشهاى سوماتيك در سرطان، تحليل پيوستگى ژنتيكى، تعيين كاهش چندتخمى بودن.
آرايه پروتئين
آرايه پروتئينى يك روش قدرتمند براى شناسايى پروتئينها میباشد. آرايه پروتئينى امكان بررسى هزاران فعل و انفعال را به صورت همزمان فراهم ميكند. پروتئينهاى به كار رفته در اين روش را ميتوان به شكل نوتركيب تهيه كرد. نتايج آرايه پروتئينى و توالى دو روش مرسوم آناليز پروتئومهای الكتروفورز ژل دو بعدى و طيف سنجى جرمى میباشد اما با وجود مؤثر بودن اين روشها، محدوديتهايى نيز وجود دارد. در اين روشها ممكن است پروتئينهاى مورد نظر كه داراى فراوانى كمى باشند، ثبت نگردند، در نتيجه براى كارهاى تشخيصى چندان مناسب نیستند چرا كه اغلب، پروتئينهاى با فراوانى اندك براى تشخيص موردتوجه هستند. در نتيجه به يك روش جامعتر و كاملتر مانند آرايه پروتئينى نياز است. در حال حاضر اين تكنولوژى به عنوان تكنولوژى محورى پروتئوميكس مدنظر ميباشد (7).
از مشخصات خوب يك ريزآرايه پايدارى شيميايى سطح آرايه بعد و قبل از فرآيند اتصال مىباشد. لكهگذارى مناسب، حداقل پيوند غيراختصاصى، داشتن پسزمينه مناسب و سازگار بودن با سيستمهاى متفاوت شناسايى از خصوصيات خوب و مهم يك آرايه پروتئينى است. روش ثابتسازى پروتئين بايد برگشتپذير و براى انواع پروتئينها مناسب باشد. هر دو روش كووالان و غيركووالان ثابتسازى پروتئينها مزايا و معايب خود را دارد. انتشار از سطوح پرمنفذ مانند ژل پلىآكريلآميد روشى موفق است كه سبب ايجاد پيوند غيركووالان بين ساختار هيدروژل و پروتئين مىشود. در اين روش، پسزمينه مناسب بوده و علاوه بر آن عملكرد پروتئين از بين نمىرود. پيوند كووالان پيوندى پايدار بوده كه براى انواعى از پروتئينها قابل استفاده مىباشد و بازگشتپذيرى خوبى دارد اما با اين وجود ممكن است برخى از خواص پروتئينها تغيير يابد. به طور كلى هر روش اتصالى مناسب بايد چنين ويژگىهايى داشته باشد:
- اتصال قوى
- حفظ عملكرد پروتئين
- پسزمينه ناچيز
ايدهآلترين سطوح آرايه عبارتند از اسلايدهاى پوشيده شده با عوامل مختلف مانند پلىآكريلآميد و سيلان نیتروسلولز و PVDF
انواع آرايه پروتئينى
Functional array (1: چيپهايى هستند كه در مقياس وسيع، تحليل پروتئينى را انجام مىدهند. اين چيپها از تعداد زيادى پروتئينهاى خالص شده كه بر روى سطح جامد ثابت شدهاند، ساخته شده و در مورد سنجش طيف وسيعى از واكنشهاى بيوشيميايى كاربرد دارند: فعل و انفعالات پروتئين -DNA پروتئين، پروتئين پروتئين- مولكولهاى كوچك و همچنين بررسى فعاليت آنزيمى، شناسايى آنتىبادىها و تعيين خصوصيات آنها. شکل 4 انواع آرایهی پروتئینی را نشان میدهد.
Capture array (2: اين نوع از چيپها شامل معرفهاى تمايلى آنتىبادىهاى اوليه و يا داربستهاى پروتئينى ميباشند. از اين روش براى شناسايى و بررسى كمى محلولهايى مانند پلاسما، سرم و استخراجات بافتى استفاده مىشود.
Reverse array (3: در اين روش محصولات حاصل از تجزيه سلول و بافت بر روى سطح چيپ ثبت شده و سپس توسط آنتىبادیهايى كه بر روى آنها قرار مىگيرد، شناسايى انجام ميشود.
شکل 4 ) انواع آرایه پروتئینی
Stoll, etal. Drug Discovery Today: TARGETS. 2004
كاربردهاى آرايه پروتئينى
- موارد تشخيصى: شناسايى آنتىبادى و آنتىژن در نمونههای خونى، پيدا كردن ماركرهاى جديد براى انواع بيمارىها، بررسى غذا و محيط، بيمارىهاى خودايمنى، آلرژى و سرطان. به عنوان مثال با كمك اين روش ميتوان در مورد بيمارى سرطان مطالعات مؤثرى از قبيل شناسايى پاسخ آنتىبادىهاى ضد تومور، كشف معرفهاى زيستى براى تومورهاى مخصوص، بررسى پاسخهاى ميزبان و تعيين كميت بيان آنتىژنهاى تومور را انجام داد.
- پروتئوميكس: بررسى بيان پروتئوم
- آناليز عملكردى پروتئينها: انفعالات پروتئين-پروتئين، خصوصيات گيرندههاى متصل شونده به ليگاند، فعاليت آنزيمها
- طبقهبندى آنتىبادىها: بررسى اختصاصى بودن و همپوشانى عملكردى آنتىبادىها و نقشه اپىتوپى آنها
نتیجهگیری
فناورى Micraarrayروشى كمهزينه، مؤثر و پربازده است و مىتواند در يافتن راهحلهاى درمانى بسيارى از بيمارىهاى مهم مانند سرطان پيشتاز باشد. آرايه روشى نوين است كه در حال تحولات و پيشرفتهاى روزانه بوده و مىتواند نقصهاى موجود در روشهاى قديمى را پوشش دهد. از مزاياى بىرقيب اين فناورى، امكان بررسى هزاران فعل و انفعال به صورت همزمان وجود دارد. با توجه به مزاياى بسيار زياد اين روش در حوزههاى پروتئوميكس و ژنوميكس ميتوان چشمانداز قابلتوجهى را براى اين تكنولوژى در حوزه تشخیص قائل شد.
منابع:
Afshari CA, Nuwaysir EF, Barrett JC. Application of complementary DNA microarray technology to carcinogen identification, toxicology, and drug safety evaluation. Cancer Research. 1999;59(19):4759-60.
- Bendixen C, Hedegaard J, Horn P. Functional genomics in farm animals–Microarray analysis. Meat science. 2005;71(1):128-37.
- Bunney WE, Bunney BG, Vawter MP, Tomita H, Li J, Evans SJ, et al. Microarray technology: a review of new strategies to discover candidate vulnerability genes in psychiatric disorders. 2014.
- Dupuy A, Simon RM. Critical review of published microarray studies for cancer outcome and guidelines on statistical analysis and reporting. Journal of the National Cancer Institute. 2007;99(2):147-57.
- Espina V, Mehta AI, Winters ME, Calvert V, Wulfkuhle J, Petricoin EF, et al. Protein microarrays: molecular profiling technologies for clinical specimens. Proteomics. 2003;3(11):2091-100.
- Espina V, Woodhouse EC, Wulfkuhle J, Asmussen HD, Petricoin EF, Liotta LA. Protein microarray detection strategies: focus on direct detection technologies. Journal of immunological methods. 2004;290(1):121-33.
- Hanash S, Robinson W, Steinman L, Utz P. Protein Microarrays: Wiley Online Library; 2003.
- Heller MJ. DNA microarray technology: devices, systems, and applications. Annual review of biomedical engineering. 2002;4(1):129-53.
- Ioannidis JP, Polyzos NP, Trikalinos TA. Selective discussion and transparency in microarray research findings for cancer outcomes. European Journal of Cancer. 2007;43(13):1999-2010.
- Lemieux B, Aharoni A, Schena M. Overview of DNA chip technology. Molecular Breeding. 1998;4(4):277-89.
- Lin Y-H, Friederichs J, Black MA, Mages J, Rosenberg R, Guilford PJ, et al. Multiple gene expression classifiers from different array platforms predict poor prognosis of colorectal cancer. Clinical Cancer Research. 2007;13(2):498-507.
- Liotta LA, Espina V, Mehta AI, Calvert V, Rosenblatt K, Geho D, et al. Protein microarrays: meeting analytical challenges for clinical applications. Cancer cell. 2003;3(4):317-25.
- Lonardi E, Balog CI, Deelder AM, Wuhrer M. Natural glycan microarrays. 2010.
- Miller MB, Tang Y-W. Basic concepts of microarrays and potential applications in clinical microbiology. Clinical microbiology reviews. 2009;22(4):611-33.
- Mueller C, Liotta LA, Espina V. Reverse phase protein microarrays advance to use in clinical trials. Molecular oncology. 2010;4(6):461-81.
- Naghieh E, Peng Y. Microarray Gene Expression Data Mining: Clustering Analysis Review. Department of Computing, University of Bradford, UK. 2004.
- Ntzani EE, Ioannidis JP. Predictive ability of DNA microarrays for cancer outcomes and correlates: an empirical assessment. The Lancet. 2003;362(9394):1439-44.
- Nygaard V, Hovig E. Options available for profiling small samples: a review of sample amplification technology when combined with microarray profiling. Nucleic acids research. 2006;34(3):996-1014.
- Schulze A, Downward J. Navigating gene expression using microarrays—a technology review. Nature cell biology. 2001;3(8):E190-E5.
- Sheehan KM, Calvert VS, Kay EW, Lu Y, Fishman D, Espina V, et al. Use of reverse phase protein microarrays and reference standard development for molecular network analysis of metastatic ovarian carcinoma. Molecular & Cellular Proteomics. 2005;4(4):346-55.
- Stoll D, Bachmann J, Templin MF, Joos TO. Microarray technology: an increasing variety of screening tools for proteomic research. Drug Discovery Today: TARGETS. 2004;3(1):24-31.
- Wulfkuhle JD, Aquino JA, Calvert VS, Fishman DA, Coukos G, Liotta LA, et al. Signal pathway profiling of ovarian cancer from human tissue specimens using reverse‐phase protein microarrays. Proteomics. 2003;3(11):2085-90.
https://medlabnews.ir/%da%98%d9%86%d9%88%d9%85%db%8c%da%a9%d8%b3-%d9%88-%da%a9%d8%a7%d8%b1%d8%a8%d8%b1%d8%af-%d8%a2%d9%86-%d8%af%d8%b1-%d8%aa%d8%b4%d8%ae%db%8c%d8%b5-%d8%a8%db%8c%d9%85%d8%a7%d8%b1%db%8c%e2%80%8c%d9%87/
برای دانلود پی دی اف بر روی لینک زیر کلیک کنید
ورود / ثبت نام