اپیژنتیک و سرطان؛ افقهای جدید در رویکردهای درمانی و تشخیصی در آزمایشگاه
(بخش دوم)
سید آرمان مهدوی1
1 دانشجوی کارشناسی ارشد بیوتکنولوژی پزشکی، مرکز تحقیقات بیولوژی مولکولی، دانشگاه علوم پزشکی بقیهالله، تهران، ایران (نویسنده مسئول) (livesnuff@gmail.com)
مقدمه:
اپیژنتیک به معنای مطالعه تغییراتی در بیان یک ژن است که قابل توارث بوده و برعکس جهشهای ژنتیکی، قابل برگشت هستند. در این زمینه انواع مطالعات انجامگرفته نشانگر نقش اساسی و مهم این موضوع در تنظیم بیان ژن و استفاده کاربردی از آن در درمان بیماریهای مرتبط نظیر سرطان است، از این رو با توجه به پیشرفتهای چشمگیر انجامشده در زمینه مطالعات وراثت و ژنتیک در جهت درمان بیماریهای مرتبط، بر آن شدیم تا با جمعآوری جدیدترین دستاوردهای علمی و مطالعات انجامشده به شکل نظاممند ساختارهای ایجاد و بررسی برهمکنش آنها با محیط و همچنین چشمانداز آینده اپیژنتیک را جهت استفاده محققان و پژوهشگران داخلی ارزیابی کنیم.
در قسمت قبلی نوشتار به بررسی مفاهیم پایه اپیژنتیک و یکی از مکانیسمهای اصلی آن یعنی متیلاسیون DNA و نقش آن در تغییرات بیان ژن و فنوتیپ اثرگذار آن به شکل خاص در سرطان پرداختیم؛ در ادامه به بررسی یکی دیگر از مکانیسمهای اپیژنتیکی مهم یعنی تغییر آرایش هیستونها میپردازیم.
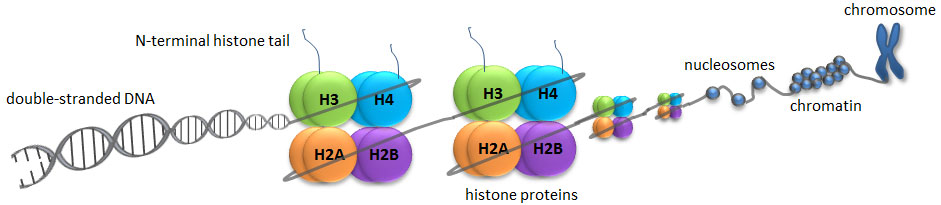
شکل 1: ساختار کروموزوم کروماتین و نوکلئوزوم (فرم فشرده) و نحوه قرارگیری پروتئینهای هیستون در آن
https://www.whatisepigenetics.com/histone-modifications/
تغییر یا اصلاح آرایش هیستونی
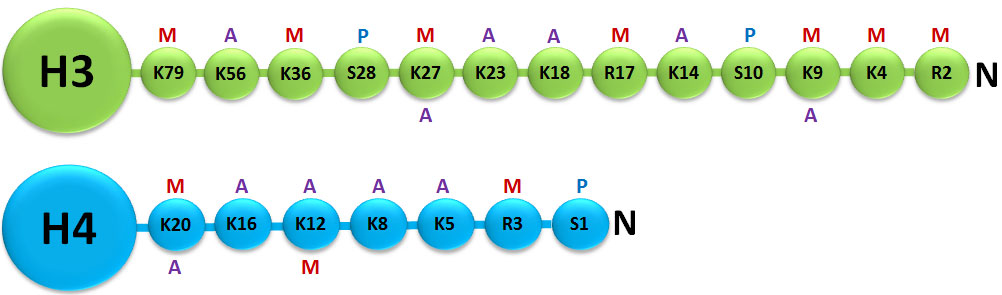
یکی از مکانیسمهای مهم اپیژنتیکی که جزوی از تغییرات کووالانسی پس از ترجمه[1] (PTM) است در پروتئینهای هیستون وجود دارد که شامل متیلاسیون، فسفوریلاسیون، استیله شدن، یوبیکوئتینه شدن[2] و اضافه شدن گروههای SUM[3] است. تغییرات هیستونها میتواند بیان ژن را با تغییر ساختار کروماتین یا تغییر بازآرایی ساختار هیستون و در نتیجه بیان ژن تحت تأثیر بگذارد. پروتئینهای هیستون برای فشردهسازی DNA به فرم فشرده کروموزوم هشت دور به دور آن میپیچند. اصلاحات هیستونها در فرآیندهای بیولوژیکی متنوعی مانند فعالسازی/ غیرفعال کردن رونویسی، بستهبندی کروموزوم و آسیب/ ترمیم DNA نقش ایفا میکنند (شکل 1). در بیشتر گونهها، هیستون H3 در درجه اول در لیزینهای 9، 14، 18، 23 و 56 استیله میشود، در آرژنین 2 و لیزینهای 4، 9، 27، 36 و 79 متیله میشود و در سری 10، ser28،Thr3 و Thr11 فسفوریله میگردد. هیستون H4 در درجه اول در لیزین 5، 8، 12 و 16 استیله میشود، در آرژنین 3 و لیزین 20 متیله و در سرین فسفریله میگردد؛ بنابراین، تشخیص کمی اصلاحات مختلف هیستون علاوه بر اینکه اطلاعات مفیدی را برای درک بهتر تنظیمات اپیژنتیکی سلول فراهم میکند، در شناخت فرآیندهای مختلف و توسعه داروهای هدفمند (اختصاصی بر روی آنزیم) در فرایند اصلاح هیستونها مورد استفاده قرار میگیرد (1).
شکل 2: ساختار توضیحدهنده هیستونهای H3 و H4 و نواحی N ترمینال
متیله شده:M، فسفریله شده:P، استیله شده:A
https://www.whatisepigenetics.com/topic/for-the-scientist/
استیلاسیون هیستون / داستیلاسیون
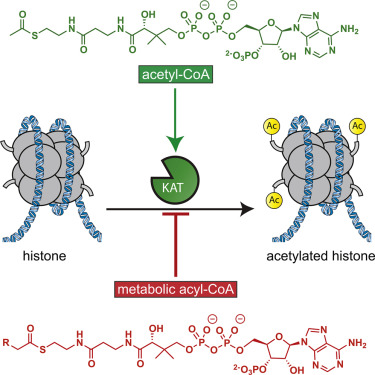
استیله شدن هیستون با افزودن آنزیمی از گروه استیل (COCH3) از استیل کوآنزیم Aرخ میدهد. روند استیلسازی هیستون بهطور جدی در تنظیم بسیاری از فرآیندهای سلولی از جمله دینامیک کروماتین و رونویسی، خاموش شدن ژن، پیشرفت چرخه سلولی، آپوپتوزیس و تمایز دخیل است. تکثیر DNA، ترمیم DNA، نقل و انتقال مواد به داخل هسته و سرکوب انتقال پیامهای عصبی نورون از جمله این نقشها است. آنزیمهای اصلاحشده درگیر در استیلسازی هیستون، استیل ترانسفراز هیستون[4] (HAT) نامیده میشوند و نقش مهمی در کنترل استیلاسیون هیستون H3 و H4 دارند. بیش از 20 نوع آنزیم HAT شناسایی شدهاند (2) که میتوانند به پنج خانواده تقسیم شوند :GNAT1؛MYST، TAFII250،P300 /CBP و مهارکنندههای گیرنده هستهای مانند ACTR.
استیلاسیون هیستون H3 ممکن است بهوسیله مهار هیستون داستیلازها[5] (HDACs) و یا کاهش مهار هیستون استیلازها اتفاق بیافتد (شکل 3).
شکل 3: ساختار کروماتین و تغییرات هیستون در دمهای هیستون N ترمینال
(a) ژنوم یوکاریوتی با بستهبندی DNA در اطراف اکتامرهای هیستون تشکیل میشود تا واحدهای اساسی کروماتین، نوکلئوزومها را تشکیل دهد. (b) اکتامر هیستون از هر نسخه H2A، H2B، H3 و H4 از دو نسخه تشکیل شده است. علاوه بر دامنههای کروی، هر یک از آنها دارای دمهای N-ترمینال هستند که از هسته بیرون میریزند، در حالی که H2A نیز دارای دم C– ترمینال است. این مولکولها تقریباً متناسب با اندازه پروتئین کشیده میشوند، اگرچه تعداد باقیماندههای نشان دادهشده به معنای انعکاس اندازه دقیق دمهای N ترمینال نیست
هیستون داستیلازها (HDAC)
موجب کاتالیز واکنش حذف گروههای استیل از باقیمانده لیزین میشوند. عدم تعادل در استیله شدن هیستون با تومورزایی و پیشرفت سرطان همراه بوده است. تشخیص اینکه آیا هیستون H3 در بقایای لیزین استیله میشود، میتواند اطلاعات مفیدی را برای توصیف بیشتر الگوهای استیلاسیون یا سایتها فراهم کند، در نتیجه منجر به درک بهتر از تنظیم اپیژنتیکی فعالسازی ژن و همچنین توسعه داروهای هدفدار HAT میشود (3).
تاکنون حداقل 4 کلاس HDAC شناسایی شده است. HDAC های کلاس I شامل 1، 2، 3 و 8 هستند. کلاس II HDAC از آنزیمهای 4، 5، 6، 7، 9 و 10 تشکیل شده است. کلاس III که بهعنوان سیتروینها شناخته میشوند، نیاز به +NAD دارند (شکل 4) و SIRT های 1-7 را نیز شامل میشوند. آنزیم Class IV که فقط شامل HDAC11 است، دارای ویژگیهای کلاس I و II است. مهار HDAC اثرات قابلتوجهی در آپوپتوزیس، توقف چرخه سلولی و تمایز در سلولهای سرطانی نشان میدهد (4).
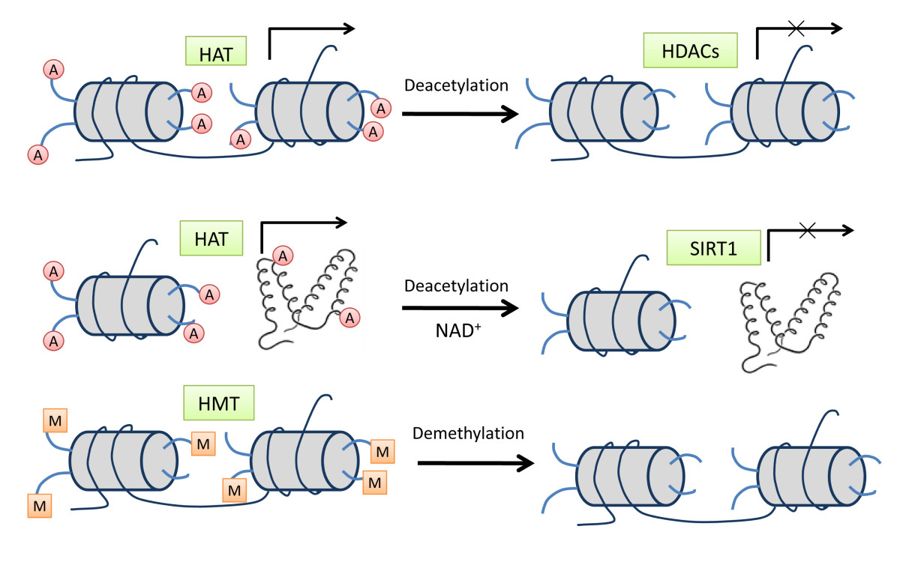
شکل (4): استیلاسیون هیستون توسط HAT واسطه مییابد و داستیلاسیون توسط خانواده HDAC کاتالیز میشود
ردیف بالایی نمایانگر فرآیندهای استیله شدن/ استیلاسیون هیستون با واسطه اعضای کلاسیک خانواده HDAC از جمله کلاسهای I، II و IV است. استیله شدن هیستون باعث ایجاد ساختار کروماتین باز و منجر به رونویسی فعال میشود، در حالی که دیاستیلاسیون هیستون همیشه با سرکوب رونویسی همراه است. ردیف وسط یک عضو خانواده کلاس HDAC کلاس III، SIRT1 را نشان میدهد که هم بسترهای هیستون و هم پروتئین را از بین میبرد و در اکثر موارد باعث خاموش شدن ژن میشود. ردیف پایین متیلاسیون هیستون را بهعنوان یکی دیگر از اصلاحات مهم هیستون نشان میدهد. متیلاسیون هیستون توسط HMT رخ میدهد و فعالسازی ژن یا سرکوب ژن توسط متیلاسیون هیستون به باقیمانده خاص لیزین که اصلاح میشود، بستگی دارد.
متیلاسیون/ دمتیلاسیون هیستونها
متیلاسیون هیستونها به شکل انتقال یک یا دو گروه متیل از S-Adenosyl-L-Methionine به باقیماندههای آرژنین یا لایزین از پروتئینهای هیستون بهوسیله هیستون متیل ترانسفراز[6](HMTs)ها انجام میپذیرد. این آنزیمها(HMTs) متیلاسیون DNA را از طریق سرکوب یا فعالسازی رونویسی وابسته به کروماتین کنترل یا تنظیم میکنند. در هسته سلولی، وقتی متیلاسیون هیستون رخ میدهد، ممکن است ژنهای خاصی در DNA که با هیستون ترکیب شدهاند فعال یا خاموش شوند (5). چندین متیل ترانسفراز هیستون مختلف وجود دارد که مخصوص لیزین یا آرژنین باقیمانده هستند که نقش اصلاحی دارند (شکل 5)،
بهعنوان مثال در هیستون H3 ،در واقع SET1، SET7 /9، Ash1، ALL-1، MLL، ALR،Trx و SMYD3 متیل ترانسفرازهای هیستون هستند که متیلاسیون هیستون H3 را در لیزین 4 (H3-K4) در سلولهای پستانداران کاتالیز میکنند. ESET، G9a، SUV39-h1، SUV39-h2، SETDB1؛ Dim-5 و Eu- HMTase متیل ترانسفرازهای هیستون هستند که متیلاسیون هیستون H3 را در لیزین 9 (H3-K9) در سلولهای پستانداران کاتالیز میکنند. آنزیمهای G9a و گروه چندکاره مانند EZH2 متیل ترانسفرازهای هیستون هستند که متیلاسیون هیستون H3 را در لیزین 27 (H3-K27) در سلولهای پستانداران کاتالیز میکنند. مشخص شده است که افزایش متیلاسیون همهجانبه H3-K27 در برخی فرآیندهای پاتولوژیک مانند پیشرفت سرطان نقش دارد .(6)
شکل 5: استیله شدن هیستون و متیلاسیون DNA
مکانیسمهای اپیژنتیکی که بر بیان ژن تأثیر میگذارند: (A) ساختار کروماتین بسته و خاموش کردن رونویسی. تنظیمشده توسط DNMT. (B) ساختار کروماتین باز و فعالسازی ژن کروماتین باز با افزایش استیله شدن دمهای هیستون توسط HAT و جذب فاکتورهای رونویسی همراه است
از طرف دیگر، متیلاسیون آرژنین هیستونهای H3 و H4 باعث فعالسازی رونویسی میشود و توسط خانوادهای از پروتئین آرژنین متیل ترانسفرازها[7] (PRMTs) کاتالیز میشود. 9 نوع PRMT در انسان وجود دارد، اما فقط 7 عضو بهعنوان متیلهکننده هیستونها گزارش شدهاند (7). آنها میتوانند واسطه تک یا دیمتیلاسیون باقیماندههای آرژنین باشند.
بر اساس موقعیت علاوه بر گروه متیل، PRMTها را میتوان در نوع یک (CARM1، PRMT1، PRMT2، PRMT3، PRMT6) و نوع دو (PRMT8 و PRMT5 و PRMT) طبقهبندی کرد. PRMTs نوع II بهشدت در بیماریهایی مانند سرطان نقش دارد؛ برای مثال PRMT5 در سرکوب برخی از ژنهای سرکوبگر تومور مانند سرکوبگرهای تومور دخیل است، در حالی که بیان بیش از حد PRMT7 در سرطان سینه مشاهده میشود. تشخیص فعالیت و مهار PRMTهای نوع II و سایر HMTها در روشن شدن مکانیسمهای تنظیم اپیژنتیکی فعالسازی و خاموش کردن ژن و همچنین بهرهمندی از تشخیص و درمان سرطان مهم است (4).
دمتیلاسیون هیستون
حذف گروههای متیل در پروتئینهای اصلاحشده هیستون از طریق دمتیلازهای هیستون است. این دمتیلازها دارای عملکردهای انکوژنیک بالقوه و درگیر شدن در سایر فرآیندهای پاتولوژیک هستند.
کشف دمتیلازهای هیستون نشان میدهد که متیلاسیون هیستون یک اصلاح دائمی نیست بلکه فرایندی پویاتر است. دو خانواده بزرگ از دمتیلازها کشف شده است: Lysine demethylase خاص 1 (LSD1) و دامنه Jumonji حاوی (دامنهjmc) هیستون دمتیلازها (JMJD2، JMJD3 /UTX و JARIDs).
بهعنوان مثال در هیستون H3 فرم مونو و دیمتیلهشده لیزین 4 توسط LSD1 (BHC110, KDM1) دمتیله میشوند. متیلاسیون مجدد در باقیماندههای خاص برای پویایی کروماتین و بیان ژن مهم است. علاوه بر این، تشخیص فعالیت و مهار این آنزیمها در روشن شدن مکانیسمهای تنظیم اپیژنتیکی فعالسازی و خاموش کردن ژن مهم بوده و ممکن است در تشخیص و درمان سرطان مورد استفاده قرار بگیرد (8).
منابع:
- Zhao QY, Lei PJ, Zhang X, Zheng JY, Wang HY, Zhao J, et al. Global histone modification profiling reveals the epigenomic dynamics during malignant transformation in a four-stage breast cancer model. Clin Epigenetics. 2016;8:34.
- Yadav CB, Pandey G, Muthamilarasan M, Prasad M. Epigenetics and Epigenomics of Plants. Adv Biochem Eng Biotechnol. 2018;164:237-61.
- Tauheed J, Sanchez-Guerra M, Lee JJ, Paul L, Ibne Hasan MOS, Quamruzzaman Q, et al. Associations between post translational histone modifications, myelomeningocele risk, environmental arsenic exposure, and folate deficiency among participants in a case control study in Bangladesh. Epigenetics. 2017;12(6):484-91.
- Stoll S, Wang C, Qiu H. DNA Methylation and Histone Modification in Hypertension. Int J Mol Sci. 2018;19(4).
- Kabekkodu SP, Chakrabarty S, Ghosh S, Brand A, Satyamoorthy K. Epigenomics, Pharmacoepigenomics, and Personalized Medicine in Cervical Cancer. Public Health Genomics. 2017;20(2):100-15.
- Ali I, Conrad RJ, Verdin E, Ott M. Lysine Acetylation Goes Global: From Epigenetics to Metabolism and Therapeutics. Chem Rev. 2018;118(3):1216-52.
- Shindo N. [Histone modification: a new era of targeting epigenetics]. Rinsho Ketsueki. 2009;50(4):282-8.
- Bennett SA, Tanaz R, Cobos SN, Torrente MP. Epigenetics in amyotrophic lateral sclerosis: a role for histone post-translational modifications in neurodegenerative disease. Transl Res. 2019;204:19-30.
[1]:Post Translational Modification
[2]: Ubiquitin Addition
[3]: Sumoylation
[4]: Histone acetyltransferase
[5]: Histone deactyltransferase
[6]: Histone methyltransferase
[7]: protein arginine methyltransferase
پانل لوکمی لنفوبلاستیک حاد کودکان (ALL) بوسیله تکنیک FISH
برای دانلود فایل pdf بر روی لینک زیر کلیک کنید
ورود / ثبت نام