MicroRNA و سرطان
(بخش اول)
زهرا اصغری لالمی (دانشجوی دکتری ژنتیک مولکولی دانشگاه آزاد پرند)
در سالهای اخیر انواع متفاوتی از RNAها شناسایی شدهاند که وظایفی بیش از نقش معمول RNA در نظریهی اساسی زیستشناسی مولکولی واتسون دارند. در نظریه واتسون، RNA واسطهی انتقال اطلاعات میان DNA و پروتئین است. این نقش به عهدهی mRNA است، اما گروه بزرگی از RNAها، مانند tRNA، rRNA، snoRNA،snRNA ، miRNA وsiRNA ، نیز از جمله مولکولهای کارکردی هستند که در گروه RNAهای غیرکدکننده (ncRNAs) قرار میگیرند و 10 درصد ژنهای کدکننده هستند (1). طبق بررسیهای اخیر، مسیرهای جالبی برای تنظیم بیان ژن شناسایی شدهاند که باواسطهی ncRNA کوچک صورت میگیرند. از جمله این مسیرها میتوان به خاموشی ژن، متیلاسیون DNA، رونویسی ژن و مسیر تداخلی RNA (RNAi) اشاره کرد. شناخت بیشتر این مسیرها و مولکولهای تنظیمکنندهی آنها موجب درک بهتری از روند زیست سلول میشود. علاوه بر این، احتمالاً ncRNAها، بهخصوص با بکارگیری مسیر RNAi، توان زیادی برای استفادهی درمانی در پزشکی و بهطور عمومیتر، تنظیم دلخواه ژنها دارند (1،2). امروزه مشخص شده که روشهای درمانی رایج در سرطان (جراحی، شیمیدرمانی و رادیوتراپی) بازده و اثربخشی کمی داشته و در دهههای اخیر مطالعات زیادی در استفاده از داروهای گیاهی و ژندرمانی که اثرات جانبی کمتری داشته باشند، انجام شده که نتایج امیدبخشی را نشان دادهاند (3).microRNA ها در کنار متیلاسیونDNA و استیلاسیون هیلتونها بهعنوان مکانیسمهای اپیژنتیکی معرفی شدهاند و در تنظیم بیان ژنها نقش مهمی دارند. توجه بیش از پیش به ساختار و عملکرد microRNAها به علت تأثیر آنها در فرآیندهای متنوع تکوینی و فیزیولوژیکی مانند آپوپتوزیس، ترشح انسولین، خونسازی، ریختزایی مغز یا تمایز بافتی و درگیری آنها در دفاع ایمنی و بیماریهای ویروسی است (4،5). microRNAها میتوانند بهعنوان انکوژن و یا مهارکنندهی تومور از طریق مهار بیان ژنهای هدف وابسته به سرطان عمل کنند (6). مکانیسم عمده تغییرات [1]miRNome در سلولهای سرطانی، بیان نابجای ژن میباشد که توسط سطوح غیرطبیعی microRNAهای بالغ تشخیص داده میشود (7). دیگر مکانیسمهای مؤثر در این امر، SNP، جهشهای رخ داده در توالی Pri-miRNA، تغییر تعداد نسخه توالیهای کدکنندهی microRNAها و رونویسی غیرطبیعی میباشد (8). ازاینرو میتوان از microRNAهای سرطانی بهعنوان نشانگرهای زیستی برای تشخیص، پیشبینی و حتی درمان استفاده کرد. در ادامه به بررسی microRNAها، تاریخچهی آنها، ساختار ژنی و بیوژنز آنها و کاربردی که در درمان سرطان در انسان دارند خواهیم پرداخت.
microRNAها
microRNAها (miRNAها)، گروهی از RNAهای تنظیمی درون سلولی غیرکدکنندهی حفاظتشدهای هستند که حدود 25-18 نوکلئوتید طول دارند و محصول shRNA و Pre-miR هستند و پس از رونویسی بیان ژن را از طریق مسیر تداخل RNAi (RNA interference) تعدیل میکنند. RNAi نوعی مکانیسم خاموشکننده پس از رونویسی در یوکاریوتها است که با ایجاد RNA دو رشتهای، تجزیهی mRNAهای مشابه را القا میکند. microRNAها بیان ژن را پس از رونویسی از طریق مهار ترجمه mRNA یا القا تجزیهی آن کنترل میکنند و آنها این عمل را از طریق اتصال بهتوالی مکمل که اغلب در ناحیهی ′UTR 3 مربوط به mRNA هدف واقع شده، انجام میدهند. البته مطالعاتی هم بیانگر وجود این توالیهای مکمل در نواحی کدکنندهی UTR′5 و حتی پروموتر میباشد. رونوشتهای اولیه miRNA طی دو مرحله پردازش تبدیل به مولکول بالغ کوتاهتری میشوند (9-7)؛ بهعنوان مثال محصول ژن کنترلکنندهی Lin-4 در Caenorhabditis elegans یک RNA، 22 نوکلئوتیدی است که از یک سنجاقسر پیشساز 60 نوکلئوتیدی ایجاد میشود و از طریق برهمکنش با یک توالی تکراری در UTR3′ Lin-14، ترجمهی
Lin-14 را مهار میکند. پراکندگی ژنهای microRNA در ژنوم بهصورت منفرد یا خوشهای است و برخی در نواحی بین ژنی و حداقل نیمی از آنها در واحدهای رونویسی معینی مثل اینترون و افزونهای کدکننده پروتئین و رونوشتهایی که پروتئین کد نمیکنند، یافت شدهاند (7،14-9). صدها ژن در ژنوم انسان این RNAها را کد میکنند. تخمین زده میشود که miRNAها کنترل حدود 30 درصد از ژنوم کدکنندهی پروتئین را بر عهده دارند (15). مطالعات اخیر نقشهای بسیار مهمی در بسیاری از عملکردهای بیولوژیکی برای این دسته از RNAها نشان دادهاند که از آن جمله میتوان به نقش آنها در تمایز، تکامل، متابولیسم و فرآیندهای مربوط به سرطان و حتی بیماریهایی مثل دیابت، آلزایمر و همچنین بیماریهای قلبی، اوتیسم و سندروم X شکننده اشاره نمود (12،16). یک نکتهی قابل توجه اینکه در نامگذاری microRNAها از پسوند حروفی و عددی استفاده میکنند (بهعنوان مثال miR-34a، miR34b، miR-9-1 و miR-9-3). پسوندهای حرفی نشاندهندهی تفاوت آنها در 2 یا 3 نوکلئوتید و اعداد، نشاندهندهی این است که کروموزومهای مختلف، این microRNAها را کد میکنند (17).
تاریخچهی microRNAها
این مولکولهای تنظیمی کوچک اولین بار در سال 1993 توسط تلاشهای آزمایشگاهی امبروس[2] و راوکان[3] بهعنوان یک گروه از ncRNA شناخته شدهاند. Lin-4 اولین microRNA است که در C.elegans کشف شد. Lin-4 برای انتقال از L1 به L2 در مرحلهی تکامل لاروی C.elegans موردنیاز است. فرگوسن[4] و همکارانش در سال 1987 دریافتند که یک جهش سرکوبگر در ژن Lin-14 میتـواند فنوتیپ جهش null مربوط به Lin-4 را برگرداند. امبروس و راوکان روی کلون کردن این دو ژن کار کردند. آنها قطعهای 700 جفت بازی را یافتند که پروتئینی کد نمیکرد و شامل Lin-4 بود، از طرفی دریافتند که Lin-14 توسط ′UTR3 در سطح پس از رونویسی تنظیم منفی شده بود. پس گزارش دادند که رونوشت کوچک و غیرکدکنندهی مربوط به Lin-4، Lin-14 را از طریق ناحیهی مکمل واقع در UTR′3 آن تنظیم میکند (13،15،18). بنابراین نشان داده شد که Lin-14 یکی از اهداف این microRNA است که بیان این microRNA در انتقال از مرحلهی اول به دوم لاروی لازم است، اما اهمیت این مولکول بهعنوان یک تنظیمکنندهی زیستی تا سال 2001 که نمونهی دیگری به نام Let-7 شناسایی شد، هنوز مشخص نشده بود. در سال 2001 دومین microRNA یعنی Let-7a هم در C.elegans شناخته شد. در سال 2005،microRNA های زیادی در ژنوم انسان کشف شدند که نشان داده شد تقریباً تمام کروموزومهای انسانی حاوی ژن برایmicroRNA هستند (19). بیشتر از نصف microRNA ها شناخته شده، در نواحی شکنندهی کروموزومها قرار دارند که در بیماریهای مختلف از جمله سرطان، مستعد حذف، اضافه و انتقال کروموزومی هستند (20).
ساختار ژنی و بیوژنز microRNAها و نحوهی مهار ترجمه
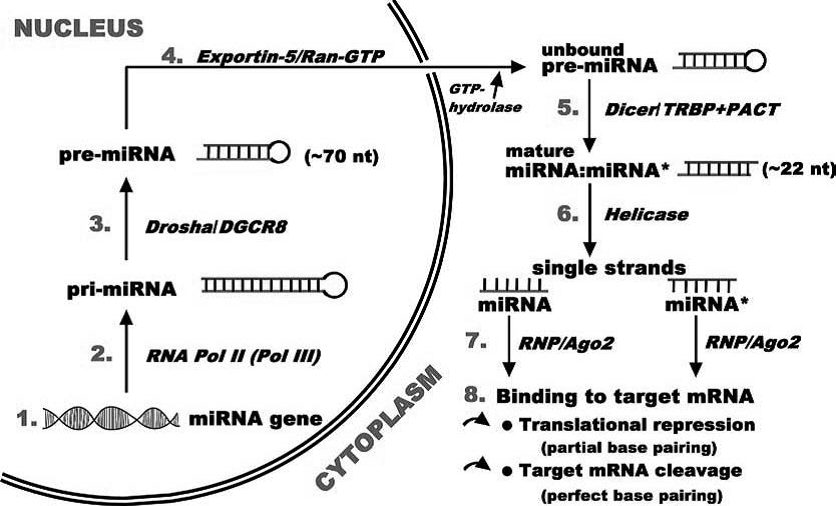
ژنهای microRNAها تقریباً یک درصد ژنوم گونههای مختلف را شامل میشوند و هر کدام از آنها صدها ژن هدف دارند. تخمین زده شده است که آنها، تنظیم کنندهی 30 درصد ژنهای کدکننده هستند (24-21). اغلب ژنهای کدکنندهی microRNAها در نواحی بین ژنی واقع شدهاند، در حالیکه برخی از آنها در نواحی درون ژنی قرار گرفتهاند (11،25). بیوژنز microRNAها در هسته و سیتوپلاسم صورت میگیرد (شکل 1). رونوشت آغازین[5] microRNAها چندین کیلو جفت باز طول دارد و توسط RNA.Pol II رونویسی شده و پلیآدنیله میشود. (گروه کوچـکی که توسـط توالــیهای تکراری مانند Alu احاطه شدهاند توسط RNA.Pol III رونویسی میشوند، اما محصول رونویسی در هر دو حالت یکی میباشد
(Pri-miRNA) که شامل کلاهک در ′5 و دم پلیآدنیله در ′3 میباشد) (11،25). ساختار ساقه- حلقه این رونوشتها توسط یک کمپلکس آنزیمی مستقر در هسته که 650 کیلو دالتون وزن دارد تشخیص داده شده و پردازش میشود. کمپلکس مذکور حاوی یک RNAse III مخصوص برش RNA دو رشتهای به نام Drosha و پروتئین متصل شونده به RNA دو رشتهای DGCR8/ Pasha است (26). پردازش اولیه منجر به ایجاد یک پیشساز سنجاقسری 110-60 (معمولاً 70) نوکلئوتیدی میشود که این microRNA پیشساز توسط فاکتور صادرکنندهی هستهای Exportin-5 و فاکتور کمکی Ran-GTP به سیتوپلاسم منتقل میشود. در سیتوپلاسم، RNAse III دیگری به نام Dicer، منجر به پردازش نهایی microRNA میشود. Dicerبه همراه عامل کمکی متصلشونده به RNA دو رشتهای (ds RNA binding partner) که در انسان HIV1-TRBP[6] و در مگس Loqs[7] نامیده میشود، لوپ انتهایی Pri-miRNA را برش میدهد و microRNA (دو رشتهای) 25-18 نوکلئوتیدی را ایجاد میکند. (در بعضی منابع 22-19 نوکلئوتید قید شده و در بعضی دیگر 24-21 نوکلئوتید). TRBP دیگری، پروتئین آرگونات[8] انسانی hAg2(EIF2C2) را به کمپلکس Dicer متصل نموده و یک کمپلکس خاموشکنندهی القاشده توسط [9]RNA را ایجاد میکند. معمولاً فقط یک رشته از microRNA بالغ دو رشتهای، به نام رشتهی راهنما[10]، به کمپلکس میکروریبونوکلئوپروتئینی[11] وارد شده و یک miRISC را ایجاد میکند که توالی این رشته جایگاه اتصال به mRNA هدف را مشخص میکند. پروتئینهایی از خانوادهی آرگونات بخش ضروری RISC هستند که حاوی دو منطقهی حفاظتشده[12] با قابلیت اتصال به RNA میباشند: منطقهی PAZ که به انتهای ′3 microRNA تکرشتهای متصل میشود و منطقهی PIWI که از نظر ساختمانی مشابه ریبونوکلئاز H بوده و با رشتهی راهنمای microRNA در انتهای ′5 برهمکنش میکند (24-21). پروتئینهای آرگونات اعضای یک خانوادهی بهشدت حفاظتشده هستند که در مسیرRNAi و microRNA درگیرند. یک پروتئین آرگونات به همراه یک RNA کوچک تکرشتهای، هستهی کمپلکس RISC را تشکیل میدهد. از 4 عضو زیرخانوادهی Ago پستانداران که همه جا یافت میشوند (شامل Ago1 تا Ago4) فقط Ago2 (برشدهنده[13]) در RNAi از طریق برش درون نوکلئوتیدی[14] mRNA هدف عمل میکند (24-21). از آنجایی که فقط یکی از رشتههای دوتایی microRNA میتواند نقش رشتهی راهنما و هدایتکنندهی RISC به ′3 UTR mRNA های هدف را بر اساس جفت شدن با mRNA ایفا کند، رشتهی دوم (پسنجر[15]) حذف میشود. رشتهی حاوی جفت باز ضعیف در پایانهی ′5 بهعنوان رشتهی راهنما انتخاب میگردد. microRNAهای متصلشونده به RISC به ′3 UTR mRNA همجنس[16] جفت میشوند و پس از رونویسی ژن، بیان آن را از طریق برش یا مهار ترجمهی mRNA هدف کنترل میکنند. غالبترین مکانیسم مهار ترجمهی mRNA، مکانیسم “خاموشی ژنی” بوده و از روش بکارگیری فاکتور برهمزنندهی تجمع ریبوزوم، eIF6، یا اتصال Ago2 به کلاهک[17] mRNA اعمال میشود، ازاینرو مانع از بکارگیری eIF4E شده و ترجمه مهار میشود. mRNAهایی که ترجمهی آنها مهار میشود، در جایگاههای سیتوپلاسمی مشخصی که اجسام پردازشی (P-bodies[18]) نامیده میشوند، مستقر شده و در آنجا ذخیره یا تجزیه میشوند (27،24-21). به منظور تجزیه، اجسام پردازشی حاوی آنزیمهای کلاهکبرداری[19] Dcp1/Dcp2، اگزونوکلئاز ′3-′5 Xrnl و فعالیت دآدنیلاسیونی[20] هستند. علاوه بر این پروتئینهای آرگونات، microRNAها و mRNAهای مهارشده نیز در اجسام پردازشی تجمع مییابند. در غیاب RNAهای کوچک یا زمانی که آرگوناتهای موتاسیون یافته قادر به اتصال بهmicroRNA نباشند، پروتئینهای آرگونات در سیتوپلاسم بهصورت پراکنده باقی میمانند. تمامیت اجسام پردازشی از طریق برهمکنش پروتئین 182 GW با پروتئینهای آرگونات حفظ میشود. مکانیسم دیگر مهار ترجمه، محصور شدن mRNA در اجسام پردازشی است، ازاینرو mRNA به پروتئین ترجمه نمیشود. این گونه mRNAها در پاسخ به تحریکات طبیعی از اجسام پردازشی رهاشده و مکانیسم ترجمه دوباره راهاندازی میشود (24-21،6)، لذا فرآیند مهار ترجمه توسط microRNA میتواند برگشتپذیر باشد. پیچیدگی فرآیندهای ذکرشده دلیلی بر اهمیت تنظیمی گستردهی microRNAهاست. یک microRNA میتواند چندین mRNA متفاوت را هدف قرار دهد و یا یک mRNA ممکن است توسط چندین microRNA کنترل شود، لذا برای تعیین عملکرد microRNA، شناسایی مولکولهای هدف microRNA اهمیت زیادی دارد (11،12،25-21،28،27).
شکل 1- مدل بیوژنز microRNA و نحوهی عملکرد آن (16)
شناسایی مولکولهای هدف microRNA
یکی از مهمترین مباحث مربوط به microRNA، شناسایی مولکولهای هدف میباشد. ایجاد تعداد کمی جفت باز مکمل برای برهمکنش عملکردی بین microRNA و توالی مولکول هدف ضروری است. در اغلب موارد، ایجاد جفت باز مکمل در 7-6 نوکلئوتید صورت میگیرد که معمولاً شامل نوکلئوتیدهای 2 تا 9 از انتهای ′5 microRNA هستند و به این ناحیه “seed” میگویند. بقیه بازهای microRNA ظرفیت جفت شدن محدودی با توالیهای ′3 UTR مجاور جایگاه seed نشان میدهند و همین اتصالات گذرا به microRNA اجازهی اتصال به چندین جایگاه درونی در یک ′3 UTR میدهد. از روشهای محاسبهای متفاوتی برای پیشبینی جایگاههای هدف microRNA استفاده میشود که شامل الگوریتمهای کامپیوتری است، اما چون جفتشدگی با توالی هدف بهصورت ناقص و محدود است، پیشبینی دقیق جایگاه هدف microRNA هنوز دشوار است. یکی از الگوریتمهای پیشبینیکننده مولکول هدف، بر مبنای جفتشدگی و حفاظتشده بودن توالی microRNA-seed در ′3 UTRهای گونههای متفاوت طراحی میشود. mRNAهایی که بهطور ترجیحی با 8-7 نوکلئوتید از توالی seed جفت میشوند، بر اساس معیارهایی مثل حفاظتشده بودن تکاملی توالی هدف و پایداری ترمودینامیکی برهمکنشهای صورت گرفته بین مابقی بازهای microRNA و توالیهای دو طرف آن در′3 UTR طبقهبندی میشوند. در نوع دیگری از جفت شدن mRNA:microRNA هدف، جفت شدن ناقص در ناحیهی seed-5′ صورت میگیرد، اما از طریق جفت شدن یک باز اضافی در انتهای ′3 microRNA، جبران میشود. مطالعات بیوانفورماتیکی و نرمافزارهای متعددی، به منظور تشخیص هدفهای microRNA از روی توالی seed بکار رفتهاند؛ برای مثال 1000 ژن microRNA برای 1 درصد ژنوم انسان تخمین زده شده است و این احتمال وجود دارد که بیش از یکسوم ژنوم انسان توسط microRNA تنظیم شود (4،5،26،29).
:منابع
1.Strachan T, Read AP. Human Molecular Genetics. GS Garland Science:Taylor & Francis Group; 2004.
2.Josien C, René FK. The role of small noncoding RNAs in genome stability and chromatineorganization. J Cell Sci 2010; 123:1825-39.
3.Dastpeyman M, Motamed N, Azadmanesh K. Inhibition of silibinin on migration and adhesion capacity of human highly metastatic breast cancer cell line, MDAMB-231, by evaluation of β1-integrin and downstream molecules, Cdc42, Raf-1 and D4GDI. Med Oncolog 2012; 29:2512-8.
- Cho WC. MicroRNAs: Potential biomarkers for cancer diagnosis, prognosis and targets for therapy. Int J Biochem Cell Biol. 2010 Aug;42(8):1273-81.
- Giovannetti E, Erozenci A, Smit J, Danesi R, Peters GJ. Molecular mechanisms underlying the role of microRNAs (miRNAs) in anticancer drug resistance and implications for clinical practice. Crit Rev Oncol Hematol. 2011 May 4. [Epub ahead of print]
- Wouters MD, van Gent DC, Hoeijmakers JH, Pothof J. MicroRNAs, the DNA damage response and cancer. Mutat Res. 2011 Apr 6. [Epub ahead of print].
- Negrini M, Nicoloso MS, Calin G. MicroRNAs and cancer–new paradigms in molecular oncology. Curr Opin Cell Biol. 2009 Jun;21(3):470-9.
- Lim, Li J, Ding X, He M, Chang SY. microRNA and Cancer. AAPS J. 2010 Sep;12(3):309-17.
- Montano M. MicroRNAs: miRRORS of health and disease. Transl Res. 2011 Apr;157(4):157-62.
- Rukov JL, Shomron N. MicroRNA pharmacogenomics: Post-transcriptional regulation of drug response. Trends Mol Med. 2011 Jun 6. [Epub ahead of print].
- Wahid F, Shehzad S, Khan T, et al. MicroRNAs: Synthesis, mechanism, function, and recent clinical trials. Biochimica et Biophysica Acta. 2010; 1803: 1231-1243.
- Quesne JL, Caldas C. MicroRNAs and breast cancer. Molecular Oncology. 2010;4:230-241.
- Imeida MI, Reis RM, Calin GA. MicroRNA history: Discovery, recent applications, and next frontiers. Mutation Research. 2011;717:1-8.
- Kim M, Kasinski AL, Slack FJ. MicroRNA therapeutics in preclinical cancer models. Lancet Oncol. 2011 Apr;12(4):319-21.
15.Shenouda Sk, Alahari SK. MicroRNA function in cancer: oncogene or a tumor suppressor. Cancer Metastasis Rev. 2009;28:369-378.
- Huang Y, Shen XJ, Zou Q, et al. Biological functions of microRNAs: a review. J Physiol Biochem. 2011;67:129-139.
- Ambros V, Bartel B, Bartel DP. A uniform system for microRNA annotation. RNA 2003; 9:277-9.
- Kasahara Y, Nakamura RM, Kim PS. The role of microRNAs in cancer: In: Grody WW, Nakamura RM, Kiechle FL, Storm Ch, editors. Molecular Diagnostics: Techniques and Applications for the Clinical Laboratory. 1 ed. California:academic press; 2010;205-14.
- Almeida MI, Reis RM, Calin GA. MicroRNA history: Discovery, recent applications and next frontiers. Mutat Res 2011 ;717:1-8.
20.Calin GA, Sevignani C, Dumitru CD. Human microRNA genes are frequently located at fragile sites and genomic regions involved in cancers. Proc Natl Acad Sci USA 2004; 101:2999-3004.
- 21. Ji W, Sun B, Su C. Targeting MicroRNAs in Cancer Gene Therapy. Barry M, ed. Genes. 2017;8(1):21.
- Peng Y, Croce CM. The role of MicroRNAs in human cancer. Rev. Signal Transduction and Targeted Therapy.2016 Jan 28;1:15004.
- Bimonte S, Leongito M, Barbieri A, et al. The Therapeutic Targets of miRNA in Hepatic Cancer Stem Cells. Stem Cells International. 2016 Mar 28;2016.
- Zhang T, Liu C, Huang S, Ma Y, Fang J, Chen J. A Downmodulated MicroRNA Profiling in Patients with Gastric Cancer. Gastroenterology Research and Practice. 2017 May 4;2017.
- Fabian MR, Sonenberg N, Filipowicz W. Regulationof mRNA Translation and Stability by microRNAs. Annu. Rev. Biochem.2010;79:351-379.
- Babashah S, Soleimani M. The oncogenic and tumour suppressive roles of microRNAs in cancer and apoptosis. Eur J Cancer. 2011 May;47(8):1127-37.
- Garza EV. microRNA: biogenesis, function and objectives identification. Rev Hematol Mex. 2011;12(1):39-46.
28.Gruppen FA. microRNAs in breast cancer [Master Thesis]. [Netherlands]: University of Utrecht;2010.
- Hanahan D, Weinberg RA. Hallmarks of cancer: the next generation. Cell. 2011 Mar 4;144(5):646-74.
[1] تمام میکرو ریبونوکلئوئیک اسیدهای موجود در ژنوم
[2] Ambros
[3] Ruvkun
[4] Ferguson
[5] Pri-miRNA
[6] HIV1-Trans activating response RNA binding protein
[7] Loquacious
[8] Argonaute
[9] RISC: RNA-induced silencing complex
[10] Guide
[11] miRNP
[12] Domain
[13] Slicer
[14] Endonucleolytically
[15] Passenger
[16] Cognate
[17] M(7) GCaP
[18] Processing
[19] Decapping
[20] De-adenylation
نقش mi-RNAها در پاتوژنز و درمان بیماری لوپوس اریتماتوس سیستمیک
Micro M.RNA، مارکری برای تشخیص سرطان
برای دانلود فایل pdf بر روی لینک زیر کلیک کنید
ورود / ثبت نام