نورترن بلاتینگ
حمیدرضا جهانتیغ، دانشجوی کارشناسی ارشد ایمنولوژی
چکیده:
از آنجا که RNA به سختی به کاغذهای نیتروسلولزی متصل میشود، از روش ساترن نمیتوان برای مولکولهای RNA استفاده کرد (21). برای انتقال مولکولهای RNA از روشی بنام نورترن بلاتینگ استفاده میشود. اساس این روش مشابه روش ساترن بلاتینگ است (19-21)، با این تفاوت که در آن از کاغذهای فعال شده یا کاغذهای DBM، استفاده میشود (15). این کاغذها را میتوان به راحتی با تیمارهای شیمیایی خاصی از کاغذ صافی تهیه کرد (12). در این روش، به علت تک رشتهای بودن RNA و تفکیک آن بر روی ژل واسرشت کننده نیازی به مرحله مجاورت با هیدرکسید سدیم نمیباشد. در ضمن به علت حساس بودن مولکولهای RNA دقت بیشتری در انجام مراحل لازم است (16).
کلید واژه: نورترن بلاتینگ، بلاتینگ، ژل پلیاکریلامید
بلاتینگ چیست؟
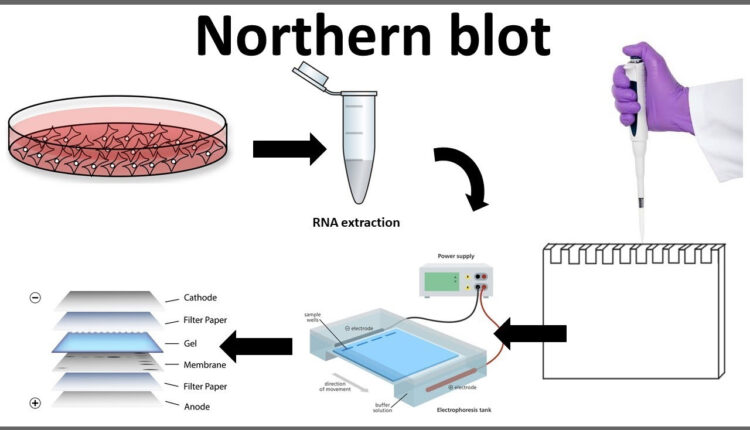
بلاتینگ روشی است برای جدا کردن DNAیا RNAیا پروتئین بر روی یک مادهی ناقل که عموماً از ژل الکتروفورز استفاده میشود (1). تکنیک وسترن بلاتینگ برای آنالیز پروتئین، ساترن بلاتینگ برای آنالیز DNA و نورترن بلاتینگ برای آنالیز RNA استفاده میشود (2).
نورترن بلاتینگ
نورترن بلاتینگ تکنیکی است که در ردیابی توالی خاص RNAکاربرد دارد. این تکنیک توسط جیمز آلوین و جوج استارک در دانشگاه استندفورد در سال 1979 ابداع شد. نام این روش به صورت متضاد از نام ساترن بلاتینگ گرفته شده است (3).
مراحل پیش از انجام آزمایش:
آماده کردن ژل آگاروز
ما در این روش نیازمند ژل آگاروز 1/2% به میزان 350 میلیلیتر میباشیم که برای این کار مقدار 4/2 گرم ژل اگاروز را در مقدار 304/5 میلیلیتر آب حل میکنیم، سپس تا دمای° 60 سانتیگراد گرم کرده و در نهایت به میزان 35 میلیلیتر از 10x MOPS و 10/5 میلیلیتر بافر فرمالدهید 37% به آن اضافه میکنیم (3).
آماده کردن پرمیکس:
- 5 ml of 10x MOPS running buffer
- 75 ml of 37% formaldehyde
- 25 ml of formamide.
آماده کردن نمونه RNA:
- 75 ml of premix
- RNA (0.5 to 10 mg)*
- water to 50 ml
*اگر نمونه ما از نظر درصد MRNA بالاتر از 0/05> باشد از MRNA توتال سرمی استفاده میشود ولی اگر میزان MRNA کم باشد میتوان از POLY(A) استفاده کرد، سپس در دمای 55 درجه به مدت 15 دقیقه انکوبه میکنیم. سپس 10 میکرولیتر از فرمالدهید به هر نمونه اضافه میکنیم و در ولتاژ100 تا 120 ولت به مدت 3 ساعت قرار میدهیم. پس از آن ژل را از تانک درمیآوریم و در SSC به مدت 45 دقیقه قرار میدهیم (5).
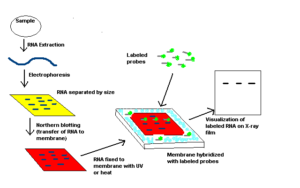
مراحل نورترن بلاتینگ:
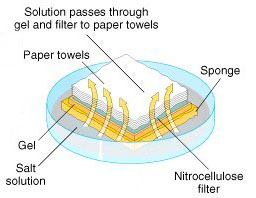
نمونه RNA از خیلی از بخشهای زیستی مانند بافتهای مختلف گرفته میشود. نمونه RNA بر روی ژل گذاشته میشود و نمونهها بر اساس سایزشان بعد از انجام الکترفورز مشخص میشوند (6). ژل بر روی یک ورقه از جنس نایلون یا نیتروسلولز که به روش ساندویچ قرار داده شده است بلاتینگ میشود، سپس پروب را که با موادی که اشعه رادیوگرافی ساطع میکنند یا با مواد کمیلومینسانس نشانهگذاری شدهاند و از خود نور ساطع میکنند را در اتاقک تاریک قرار میدهیم و جواب را بر روی فیلم X-ray مشاهده میکنیم (7).
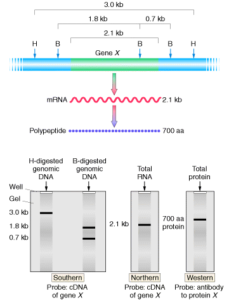
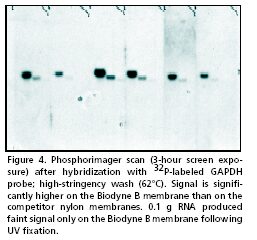
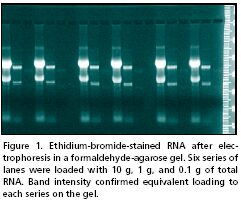
نمونههایی از انواع نتایج نورترن بلاتینگ
شناسایی microRNAs توسط نورترن بلاتینگ
microRNAs که از سلولهای مختلف میزبان و ویروسها منشأ میگیرند نقش مهمی در سیکل عفونت ایفا میکنند. شناسایی microRNAs و اینکه چه اثری بر میزبان خود دارند اساساً با روش نورترن بلاتینگ انجام میشود (8). همانطور که گفته شد روش نورترن بلاتینگ سبب جداسازی microRNAs از نظر سایز میشود و میتواند اطلاعات جامعی از سایز و ساختمان اصلی آن بدهد، بنابراین میتواند یک وسیله باارزش برای کشف و شناسایی انواع جدید microRNAs باشد (10 )
روش حساس نورترن بلاتینگ با استفاده از مواد غیر رادیواکتیو برای شناسایی microRNAs
ادامه داشتن شناسایی RNAهای کوچک (small RNAs) فعال نیازمند آن است که میزان دقیق درصد و اندازهی آنها با یک روش دقیق شناسایی شود (12). امروزه نورترن بلاتینگ به صورت گسترده به عنوان یک روش مرجع برای شناسایی small RNAs مورد استفاده قرار میگیرد. در تکنیک جدیدی که ارائه شده است نوعی از بلاتینگ که بر اساس پروتکل نورترن بلاتینگ برای small RNAs میباشد جهت small RNAs با طول 40-15 باز استفاده میشود (13). در این تکنیک از دایگوگسین که با الیگونوکلوئیک اسید و 1-ethyl-3-(3-dimethylaminopropyl) برای اتصال با RNA استفاده شده است.
این روش به طور مشخص برای شناسایی RNAها با طول کمتر از 0.05FMOL مورد استفاده قرار میگیرد که چند ثانیه بعد از مواجه شدن غشا با ماده نشاندار شده که در بالا ذکر شد میتواند از خودش نور در محفظه تاریک ساطع کند و این روش 1000 بار تولید نور را بهبود بخشیده است. در مقایسه با روش اصلی که خیلی خطرناک است و نیازمند نمونه تازه میباشد این روش خیلی ارزانتر است و نسبت به محیط زیست کمتر آسیبزا بوده و بسیار دقیقتر میباشد (14).
مزیتهای نورترن بلاتینگ
- روشی استاندارد در مورد مطالعه میزان بیان ژن MRNA
- شناسایی میزان سایز MRNA
- مطالعه بر روی اسپلایسینگ RNA
- مطالعه بر روی نیمه عمر RNA
- اغلب برای تأیید و چک کردن موشهای ترانشژنیک و ناکاوت شده استفاده میشود.
- میتواند برای پروبهای ناقص استفاده شود.
- غشا میتواند ذخیره شود و سالها بعد دوباره بازبینی و همچنین دوباره پروبگذاری شود.
معایب نورترن بلاتینگ:
1- شناسایی چند پروبی مشکل است.
2- اگر نمونه ما حتی به میزان اندک با آنزیم RNAase آسیب ببیند کیفیت جوابها و میزان بیانها دچار مشکل منفی کاذب میشوند.
3- روش استاندارد نورترن بلاتینگ حساسیت کمتری نسبت به روشهای دیگر مثل RT-PCR دارد.
:References
- Ghildiyal,M. and Zamore,P.D. (2009) Small silencing RNAs: an expanding universe. Nat. Rev. Genet., 10, 94–108.
- Li,Z., Kim,S.W., Lin,Y., Moore,P.S., Chang,Y. and John,B. (2009) Characterization of viral and human RNAs smaller than canonical MicroRNAs. J. Virol., 83, 12751–12758.
- Varallyay,E., Burgyan,J. and Havelda,Z. (2008) MicroRNA detection by northern blotting using locked nucleic acid probes. Nat. Protoc., 3, 190–196.
- Holtke,H.J. and Kessler,C. (1990) Non-radioactive labeling of RNA transcripts in vitro with the hapten digoxigenin (DIG); hybridization and ELISA-based detection. Nucleic Acids Res., 18, 5843–5851.
- Ramkissoon,S.H., Mainwaring,L.A., Sloand,E.M., Young,N.S. and Kajigaya,S. (2006) Nonisotopic detection of microRNA using digoxigenin labeled RNA probes. Mol. Cell Probes, 20, 1–4.
6-Kornberg, A., Baker, T. A., DNA Replication. (2nd ed.) pp 15, W. H. Freeman & Co. (1992)
7-Sobel, R., Dubitsky, A., Brickman, Y. (2002), Genetic Engineering News, 23, 2.
8-Southern, E. M., (1975) J. Mol. Biol. 98:503-51
9-Voet, D., Voet, J. Biochemistry. (vol. 1, 3rd ed.), pp 110-112, John Wiley & Sons Inc. (2004).
10-Jump up to: a b c Alberts, B., Johnson, A., Lewis, J. Raff, M., Roberts, K., Walter, P. 2008. Molecular Biology of the Cell, 5th ed. Garland Science, Taylor & Francis Group, NY, pp 538–539.
12- Kevil, C. G., Walsh, L., Laroux, F. S., Kalogeris, T., Grisham, M. B., Alexander, J. S. (1997) An Improved, Rapid Northern Protocol. Biochem. and Biophys. Research Comm. 238:277–279.
13- Jump up to: a b c d e Schlamp, K.; Weinmann, A.; Krupp, M.; Maass, T.; Galle, P. R.; Teufel, A. (2008). “BlotBase: A northern blot database”. Gene 427 (1–2): 47–50. doi:10.1016/j.gene.2008.08.026. PMID 18838116.
14- b c d e Trayhurn, P. (1996) Northern Blotting. Pro. Nutrition Soc. 55:583–589.
15- Alwine JC, Kemp DJ, Stark GR (1977). “Method for detection of specific RNAs in agarose gels by transfer to diazobenzyloxymethyl-paper and hybridization with DNA probes”. Proc. Natl. Acad. Sci. U.S.A. 74 (12): 5350–4. doi:10.1073/pnas.74.12.5350. PMC 431715. PMID 414220.
16-15- Bor, Y.C.; Swartz, J.; Li, Y.; Coyle, J.; Rekosh, D.; Hammarskjold, Marie-Louise (2006). “Northern Blot analysis of mRNA from mammalian polyribosomes”. Nature Protocols. doi:10.1038/nprot.2006.216
16-^ Valoczi, A., Hornyik, C., Varga, N., Burgyan, J., Kauppinen, S., Havelda, Z. (2004) Sensitive and specific detection of microRNAs by northern blot analysis using LNA-modified oligonucleotide probes. Nuc. Acids Research. 32: e175.
17- Gortner, G.; Pfenninger, M.; Kahl, G.; Weising, K. (1996). “Northern blot analysis of simple repetitive sequence transcription in plants”. Electrophoresis 17 (7): 1183–1189. doi:10.1002/elps.1150170702. PMID 8855401.
18-Liang, P. Pardee, A. B. (1995) Recent advances in differential display. Current Opinion Immunol. 7: 274–280.
19-Engler-Blum, G.; Meier, M.; Frank, J.; Muller, G. A. (1993). “Reduction of Background Problems in Nonradioactive Northern and Southern Blot Analysis Enables Higher Sensitivity Than 32P-Based Hybridizations”. Anal. Biochem 210 (2): 235–244. doi:10.1006/abio.1993.1189. PMID 7685563.
20- Baldwin, D., Crane, V., Rice, D. (1999) A comparison of gel-based, nylon filter and microarray techniques to detect differential RNA expression in plants. Current Opinion in Plant Biol. 2: 96–103.
21-Utans, U.; Liang, P.; Wyner, L. R.; Karnovsky, M. J.; Russel, M. E. (1994). “Chronic cardiac rejection: Identification of five upregulated genes in transplanted hearts by differential mRNA display”. Proc. Natl. Acad. Sci. USA 91 (14): 6463–6467. doi:10.1073/pnas.91.14.6463. PMC 44222. PMID 8022806.
https://medlabnews.ir/%d9%88%d8%b3%d8%aa%d8%b1%d9%86-%d8%a8%d9%84%d8%a7%d8%aa/
برای دانلود پی دی اف بر روی لینک زیر کلیک کنید
ورود / ثبت نام