تكنيك Invader
دکتر مهدی فصیحی رامندی (عضو هیئت علمی دانشگاه علوم پزشکی بقیه ا… (عج))
زهرا کریمی (مرکز تحقیقاتی زیست سلول پژوهان تدبیر)
دکتر رضا میرنژاد (دانشیار دانشگاه)
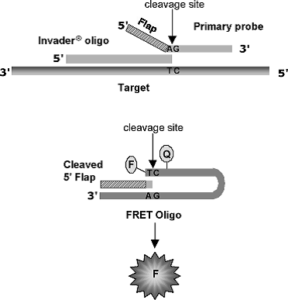
تکنیک Invader assay يك روش مناسب تعيين ژنوتيپ براي بررسي DNA ژنوميك ميباشد. علیرغم اینکه در اين تكنيك الزامي به تكثير قطعه موردنظر نيست (هرچند كه ميتوان اين قطعه را تكثير نمود)، اما میتوان از آن براي شناسايي تغييرات تكنوكلئوتيدي، حذف، اضافه، تعداد كپيهاي يك ژن، تشخيص عوامل عفوني و مطالعه بيان ژن از صحت بالايي استفاده کرد. اين تكنيك بر روي DNA ژنوميك، RNA، محصول PCR و RT-PCR قابل انجام ميباشد. در واكنش Invader، بهجای تكثير هدف، پيغام اختصاصي هدف تكثير ميشود. اين تكنيك بر پايه توانايي آنزيمهاي برشدهنده[1] در شناسايي و برش ساختارهاي خاصي از اسيد نوكلئيك مهاجم راهاندازي شده است. اين ساختار خاص هنگامي بهوجود ميآيد كه دو اوليگونوكلئوتيد[2] (اوليگوي مهاجم[3] و كاوشگر اوليه[4]) كه به هدف چسبيدهاند، در قسمتي از ساختار خود بر روي هم قرار گيرند.
آنزيم Cleavage
اين آنزيمها يك مجموعه آنزيمي، شامل ‘5 اندونوكلئازهاي اختصاصي ساختار ترموفيل طبيعي و مهندسیشده ميباشند. در Invader DNA assay از آنزيمهاي Cleavage بهدستآمده از باكتريهاي ترموفيل مثل آركیباكتر استفاده ميشود. اين آنزيمها جزء خانواده اندونوكلئازي flap (FEN1) ميباشند. قسمت اصلي اين آنزيمها مربوط به ناحيه ‘5 اگزونوكلئازي آنزيم DNA پليمراز I يوباكترهاي ترموفيل ميباشد.
ساختار اوليگونوكلئوتيدها
كاوشگر اوليه داراي دو بخش است؛ انتهاي ‘3 كه اختصاصي هدف ميباشد[5] (TSR) و بالة[6] ‘5. توالي بالة ‘5 مستقل از توالي هدف است. اين باله داراي يك توالي عمومي براي شناسايي میباشد. در حضور هدف اختصاصي، انتهاي ‘3 اوليگومهاجم بر روي بخش متصل به هدف كاوشگر اوليه قرار ميگيرد. اين روي هم قرار گرفتن در موقعيت باز شماره 1 رخ ميدهد. اگر باز شماره 1 كاوشگر اوليه مكمل هدف باشد، كاوشگر و اوليگومهاجم ساختار روي هم[7] را تشكيل ميدهند. در اين حالت آنزيم Cleavage انتهاي ‘3 كاوشگر اوليه را در موقعيت باز 1 برش ميدهد. در اینجا باله ‘5 و انتهاي ‘3 TSR آزاد ميشود. اگر باز موجود در ‘3 مكمل هدف نباشد، بهجاي تشكيل ساختار Invader، يك ساختار شكافدار تشكيل ميشود. در نتيجه باز شماره 1 كاوشگر اوليه، جزئي از ساختار بالۀ آزاد ‘5 ميشود. در اين حالت آنزيم نميتواند ساختار شكافدار[8] حاصل را شناسايي و برش دهد.
چرخه كاوشگر و تقويت پيغام
انتهاي TSR كاوشگر طوري طراحي شده است كه دماي ذوب آن نزديك دماي انجام واكنش باشد. اين امر موجب ميشود كه كاوشگر بریدهشده با كاوشگر سالم بهطور دائمي تعويض شوند. پس از برش آنزيمي قسمت باله و TSR از هدف جدا ميشوند و يك كاوشگر سالم جديد جايگزين آن میگردد. ازآنجاکه كاوشگر اوليه به مقدار زيادی در دسترس است، تعداد زيادي از آنها تحت واكنش برش آنزيمي قرار ميگيرند، در نتيجه مقدار زيادي از بالۀ بریدهشده در محلول واكنش تجمع پيدا ميكند. بالۀ آزاد بهعنوان اوليگوي انتقالدهنده انرژي فلورسانس[9] (FRET) عمل ميكند. اين باله در يك انتها فلوروفور و در انتهاي ديگر محل برش آنزيم داراي رنگ خاموشکننده[10] ميباشد. آنزيم برشدهنده، فلوروفور را از خاموشکننده جدا ميسازد و موجب ايجاد پيغام فلورسنت ميشود. اگر از دو باله داراي توالي و رنگ فلوروفور متفاوت استفاده شود، دو توالي مختلف در يك چاهك قابل شناسايي خواهد بود. محلول واكنش را به مدت 4 ساعت در دماي 63 درجه سانتیگراد انكوبه كرده و با استفاده از دستگاه Fluorescence plate reader، فلورسانس ساطعشده، اندازهگيري ميشود. از روي ميزان فلورسانس ساطعشده از نمونه طبيعي[11] و نمونه جهشیافته نسبتي به دست ميآيد كه با استفاده از اين نسبت، هموزيگوت و هتروزيگوت بودن نمونهها تعيين ميشود. تعداد كپيهاي يك ژن از مقايسه آن ژن با يك ژن بيانشونده دائمي (خدمتكار) مثل βتوبولين به دست ميآيد (شکل 1).
شکل 1: تصویر شماتيك از تكنيك Invader
مدل DNA
در مدل DNA[12]، باله ‘5، يك ساختار دورشتهاي مهاجم موقتي را با اوليگو FRET به وجود ميآورد. آنزيم Cleavage ساختار حاصل را شناخته و فلوروفور را از ‘5 اوليگو FRET جدا ميسازد، سپس باله از اين ساختار جدا شده و ميتواند مجدداً با اوليگو FRET سالم ديگري اتصال يافته و دوباره ساختار تهاجمي تشكيل دهد. برش خودبهخودي كاوشگر و اوليگو FRET در يك دماي ثابت كه نزديك دماي ذوب كاوشگر است، روي ميدهد.
مدل RNA
آنزيمهاي Cleavase نوع FEN كه در مدل DNA به كار ميروند، اهدافي كه از نوع RNA باشد را شناسايي نميكنند، بنابراين در مدل RNA از آنزيمهاي مشتق از DNA پليمراز استفاده ميشود (نوع pol). آنزيمهاي نوع pol هم DNA و هم RNA را مورد شناسايي قرار ميدهند. در اين مدل دو قدم متوالي تحت عنوان واكنش اول و دوم وجود دارد؛ در واكنش اول كاوشگر و اوليگوي مهاجم، يك ساختار تهاجمي را روي RNA الگو تشكيل ميدهند. يك اوليگوي اضافه ديگر تحت عنوان Stacker به انتهاي ‘3 كاوشگر چسبيده و باعث میشود که كاوشگر در دماي بالاتر نيز به هدف بچسبد. اين امر حساسيت روش را افزايش ميدهد. در واكنش دوم، محصول برش واكنش اول (باله ‘5 و TSR) با الگوي واكنش دوم[13] (SRT) و اوليگو FRET يك ساختار روي هم را تشكيل ميدهد. برش آنزيمي اوليگو FRET باعث ميشود كه فلوروفور از مولكول خاموشکننده جدا شده و پيغام فلورسانس آن ساطع شود. يك اوليگوي متوقفكننده[14] به واكنش دوم اضافه شده و به TSR و قسمتي از باله ‘5 متعلق به كاوشگر سالم متصل ميشود. ازآنجاکه اين متوقفكننده به كاوشگر سالم متصل شده، اما به بالۀ ‘5 آزاد نميچسبد، باعث ايجاد پيغام قويتري ميشود.
روشهاي فرعي آشكارسازي
تركيب و توالي باله ‘5، مستقل از هدف بوده و به روشهاي مختلفي (اندازۀ باله، توالي، شارژ و فلورسانس) قابل شناسايي ميباشد. اين خصوصيت به تكنيك Invader اين توانايي را ميدهد كه از روشهاي فرعي متعددي براي تشخيص و آشكارسازي استفاده نمايد (مثل Mass spectrometry، الكتروفورز لوله موئينه، Microfluidics، استفاده از آرايه تراشهها[15]، تسخير[16]، حسگر و فلورسانس)
شركت ACLARA Biosciences مولكولهاي فلورسنت كوچك و قابل جداسازي با الكتروفورزي را توليد كرده است كه به نام مولكولهاي گزارشگر eTag معروف شدهاند. اين مولكولها بهعنوان باله ‘5 به كاوشگر اوليه متصل ميشوند. eTag آزاد با استفاده از تجهيزات تعيين توالي DNA به روش استاندارد لوله موئينه، بهصورت كمّي اندازهگيري ميشود. با توسعه صدها نوع eTag، امكان انجام اين تكنيك بهصورت چندگانه[17] به وجود آمده است. شرکت ACLARA همچنین نشان داد كه امكان شناسايي 26 آزمايش Invader با مدل RNA در يك لوله آزمايش وجود دارد.
اختصاصيت تکنیک Invader
اين تكنيك تركيبي از شناسايي توالي با تشخيص آنزيمي ساختار است، بنابراين اختصاصيت آن از ديگر روشها بالاتر ميباشد. اين اختصاصيت از نوع ساختار تهاجمي آن حاصل ميشود. اگر توالي محل برش کاملاً مكمل هدف نباشد، ساختار تهاجمي تشكيل نميشود، در نتيجه بالۀ ‘5، جهت شناسايي در واكنش دوم آزاد نميشود. اختصاصيت بالا، قدرت تمايز و صحت روش را در تشخيص جهشها، افزايش ميدهد. با توجه به موارد فوق اين تكنيك ميتواند هر سه ژنوتيپ را بهخوبی شناسايي و دستهبندي نمايد. ازآنجاکه اين تكنيك در آزمايشات كمّي نيز قابل استفاده است، از آن در شناسايي مستقيم كروموزوم و تعداد كپيهاي ژن از روي DNA ژنومي، ميتوان استفاده كرد. بدين منظور ميتوان از يك ژن مرجع مثل βتوبولين (بهشرط اينكه اين ژن فاقد چندشكلي و حذف و اضافه باشد) استفاده كرده و پيغام فلورسانس حاصل از ژن مورد بررسي را با اين ژن مرجع مقايسه نمود. در اين حالت نتيجه بهتر و قابلقبولتری بهدست ميآيد. طراحي اوليگو مهاجم بايد بهدقت صورت گيرد تا كمترين تشابه ممكن را با ديگر نقاط ژنوم داشته باشد. اين طراحي دقيق موجب افزايش صحت اندازهگيري كمّي توالي هدف ميشود.
اين تكنيك با بسياري از روشهاي آمادهسازي نمونه و همچنين نمونههاي مختلف از منابع متفاوت سازگار است. با استفاده از اين تكنيك ميتوان نمونههاي جانوري و گياهي را با دقت بالايي تعيين ژنوتيپ نمود. همچنين روشهاي آمادهسازي نمونه بهصورت تجاري براي اين تكنيك وجود دارد.
سهولت انجام كار، انعطافپذيري و قابليت كمّي شدن روش، از مزاياي اين تكنيك ميباشد. اين تكنيك در پليتهاي ميكروتيتر قابل انجام است. همچنين اين تكنيك ميتواند بهصورت دستي، نيمه اتوماتيك و در شرايطي كه حجم كار بالاست، بهصورت اتوماتيك انجام شود.
كاربردهای تکنیک Invader
كاربرد اين روش عبارت است از:
- شناسايي جهش
- تعيين ژنوتيپ در سطح وسيع
- تشخيص و طبقهبندي بيماريهاي عفوني
- شناسايي تراريخته و اندازهگيري كمّي آن
- تعيين تعداد كپيهاي يك قطعه
- آزمايش بيماريهاي ژنتيكي
- مطالعه بيان ژن
[1] Cleavage
[2] Oligonucleotide
[3] Invader oligonucleotide
[4] Primary probe
[5] 3′ target-specific region
[6] Flap
[7] Overlapping structure
[8] Nicked structure
[9] Fluorescence resonance energy transfer
[10] quencher
[11] Wild type
[12] DNA format
[13] Secondary reaction template
[14] Arrestor oligonucleotide
[15] Array chips
[16] Capture
[17] Multiplex
برای دانلود فایل pdf بر روی لینک زیر کلیک کنید
ورود / ثبت نام