تشخیص مولکولی بیماریهای ارثی و استفاده از آن در تشخیص قبل از تولد و تشخیص پیشکاشتی
استفاده از روشهای نوین تعیین توالی
نویسندگان:
مریمالسادات دانشپور، دکتری ژنتیک ملکولی *
محمدصادق فلاح، پزشک، دکتری ژنتیک ملکولی *
ساجده مسجودی، کارشناس ارشد ژنتیک *
* شرکت خدمات تخصصی ژنتیک، ژن کاوش آزما
خلاصه
به دنبال پیشرفتهای حاصل در روشهای تشخيصی بیماریهای ارثی و مادرزادی، اکنون امکان تشخیص مولکولی در بسیاری از بیماریهای ارثی که ژن مسئول آن شناخته شده است، فراهم میباشد. با استفاده از روشهای سنتی و نیز نوین تشخیص ژنتیک و حصول به تشخیص مولکولی، میتوان از نتايج حاصله در تشخيص قبل از تولد[1] و تشخیص پیشکاشتی[2] بهره برد تا از تولد نوزاد معلول بعدی پيشگيری شود.
اولــين توالیهای DNA در اوايــل دهــه 1970 توســط پژوهشگران با بکارگیری روشهای بسيار مشكل و پرزحمـت بر اساس كروماتوگرافي دوبعدي به دست آمـد و بهتدریج بـه دنبال پيشرفتِ روشهای آناليز خودكار مبتني بـر رنـگ، انجـام تعيين تواليِ DNA آسانتر و سریعتر شد [1]. استفاده از تعیین توالی مستقیم ژنوم در بسیاری از بیماریهای ژنتیک بهعنوان استاندارد طلایی[3] مطرح بوده و هنوز نیز مورد استفاده میباشد.
به دنبال انجام پروژه ژنوم و معرفی نـسل جدید روشهای تعيـين تـوالي ژنوم[4] بـا تـوان عمليـاتي بـالا[5] امروزه امکان استفاده از روشهای نوين ژنوميک در تشخیص بیماریهای ارثی فراهم گردیده است. با استفاده از این روشهای نوین تشخیصی ژنتیک میتوان در کودکان مبتلا به اختلالات ژنتیک پیچیده با ارائه نتایج بهموقع و دقیق، به پزشک و خانواده بیمار جهت تشخیص و پیشگیری کمک نمود.
انجام آزمايشات تشخيص ژنتيک با استفاده از نسل جديد روشهای تعيين توالي برای بيماران مبتلا به بیماریهای ارثی مانند عقبماندگی ذهنی، ناشنوايی، بیماریهای عصبی عضلانی، بیماریهای متابوليک و … در موارد زیر کمککننده است [2]:
- مواردی که فنوتیپ بیماری قویاً مؤید یک بیماری ارثی بوده ولی آن را نمیتوان به یک بیماری مشخص با ژن مشخص نسبت داد.
- در مواردی که با یک بیماری مشخص با ژنهای متعدد روبرو هستیم[6] و یا ژن مسئول بیماری به حدی بزرگ است که استفاده از روشهای نوین تشخیصی، هزینه بررسی و زمان موردنیاز برای حصول جواب نسبت به روشهای سنتی بهطور چشمگیری کاهش مییابد.
- در مواردی که تستهای اختصاصی یک بیماری ارثی به نتیجهای نرسیده ولی همچنان مشکوک به یک بیماری ارثی با ظن قوی میباشیم.
مقدمه:
اولــين توالیهای DNA در اوايــل دهــه 1970 توســط پژوهشگران با بکارگیری روشهای بسيار مشكل و پرزحمـت بر اساس كروماتوگرافي دوبعدي به دست آمـد و بهتدریج بـه دنبال پيشرفتِ روشهای آناليز خودكار مبتني بـر رنـگ، انجـام تعيين تواليِ DNA آسانتر و سریعتر شد [1]. يكي از نخستين نمونههای تعيينِ توالي كامل، تعيين توالي RNA مربوط به ژنوم باكتريوفاژ MS2 بود كـه توسـط والتـر فيـرز[7] و همكـارانش در دانـشگاه Ghent بـين سالهای 1972 و 1976 انجام شد [5-3]. قبل از پيشرفت سريع روشهای تعيـين تـوالي DNA، در اوايل دهه 1970، روشهای بسيار پرزحمتـي توسـط فردريك سانگر[8] در دانشگاه Cambridge انگلستان و همچنين والتر گيلبـرت[9] و آلـن ماكسام [10]در Harvard به كار گرفتـه شد [6]، بهعنوان مثـال در سـال 1973 گيلبـرت و ماكـسام توسـط بکارگیری روش شناختهشده آنـاليز نقطهای[11] تعيينِ توالي 24 جفت باز را گزارش دادند [7]. ايـن روش بـه روش بـرش شـيمیايي[12] معروف شد که يك روش پرزحمت و مبتني بر استفاده از مواد راديواكتيو و برش شيميايي بود. در سال 1977 سانگر و همكارانش روش تعيين تـوالي بـر مبناي ختم زنجيره[13] را پيشنهاد نمودنـد كه بعدها به دليل آسان بـودن نـسبي و قابـل اطمينـان بـودنش تبديل به روش انتخابي تعيين توالي شد [9،8،6]. اسـاس ايـن روش بر مبناي تعيين توالي بازهاي DNA بر اساس ختم سـنتز DNA در حال تكثيـر در نقـاط مختلـفDNA و جداسـازي قطعات كوچك DNA است. در مقاله حاضـر روشهای تعيـين تـوالي ماكـسام- گيلبرت، ختم زنجيره و ختم زنجيره با استفاده از رنگ معرفي و چالشهای موجود در اين زمينه بررسي میشود، سـپس نـسل جدید روشهای تعيـين تـوالي بـا تـوان عمليـاتي بـالا[14] و مواردي كه امـروزه بهصورت تجـاري در دنياي ژنتيك كاربرد بيشتري دارد بهاختصار معرفي میشود [10].
روشهای تعيين توالي
- ماكسام- گيلبرت
- ختم زنجيرسازي (روش سانگر)
- ختم زنجيرسازي با استفاده از رنگ[15]
- تعيين توالي انبوه
الف- تعيين توالي به روش ايلومينا
ب- تعيين توالي به روش SOLiD
ج- روش تعيين توالي حرارتي[16]
د- روش SMRT
5- روشهای کمکاربرد ولي مطرح
الف- نانو منفذ[17]
ب- Helioscope
1: ماكسام- گيلبرت:
يك روش تعيين توالي DNA بر اساس تغيير شيميايي DNA و متعاقب آن ايجاد شكاف در بازهاي خاص میباشد [6]. این روش نيازمند نشاندار كردن انتهاي’5 DNA بهوسیله راديواكتيو- بهطور معمول بهواسطه يـك واكـنش كينـاز و بـا استفاده از Gamma-32P ATP- اسـت. ابتدا قطعـه DNA براي تعيين توالي استخراج و تخلیص میشود و تحت تیمار شیمیایی قرار میگیرد. این تيمار شيميايي باعث ایجاد شكست در نـسبت كـوچكي از 1 يـا 2 نوكلئوتيـد از مجمـوع 4 نوكلئوتيد میشود. قطعات حاصل از این تیمار عبارتند از: گوانین، سیتوزین، پورینها (آدنین+گوانین) و پیریمیدین (سیتوزین+گوانین). هماکنون قطعات حاصله میتوانند تحت اثر مواد شیمیایی تغییر شکل دهند تا جهت ردیابی آماده شوند. بهعنوان مثال:
1) پورينها (A+G) با استفاده از فرميك اسيد، پورينزدايـي[18] میشوند.
2) گوانينها (و تا حدي آدنينهـا) بـا ديمتيـل سـولفات، متيله میشوند.
3) پيريميدينها (C+T) با هيدرازين متيله میشوند.
4) اضافه نمودن نمك (سديمكلرايد) به واكنش هيدروژني كه متيله شدن تيمين را در واکنشهای فقـط حـاوي سـيتوزين مهار میکند.
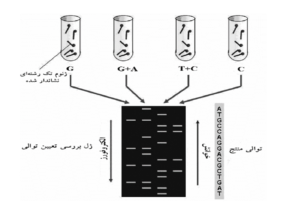
ســپس DNA تغييــرشــكليافتــه بهواسطه پيپريــدينِ[19] داغ در مكان باز تغییریافته برش داده میشود. غلظت مواد شيميايي كه باعث تغييـر میشود، طـوري كنتـرل میشود كه بهطور متوسط يـك تغييـر در هـر مولكـول DNA ايجاد شود. درنتیجه يـك سـري از قطعـات نشاندار شـده بـا فاصله متفاوت بين انتهاي نشاندار شده و اولين محـلِ بـرش در هر مولكول توليد میشود. قطعات حاصل از 4 واكنش در كنـار يكديگر براي جداسازي بر اساس اندازه روي ژل آكريـلآميـد تغييرماهيتدادهشده[20] الكترفـورز میشود و جهت مشاهده قطعات، ژل و فيلم راديولوژي كنـار هـم قرار میگیرند. فيلم در نقاطي تيـره و روشـن خواهـد بـود كـه باندهاي تيره نشانگر يك قطعه نشاندار DNA است كه توسـط آن توالي DNA استنباط میشود (شكل 1). اين روش كه تعيين توالي شيميايي ناميده میشود، بعدها منجـر بـه ابـداع سـنجش متيلاسـيون تـداخلي[21] شد كه در پيدا كردن مکانهای اتـصال پروتئینها بـه DNA استفاده میشود.
شكل 1 – روش تعيين توالي ماكسام- گيلبرت
2: ختم زنجيرسازي (روش سانگر):
این روش دارای كارآمدي بيشتر، استفاده كمتر از مواد شيميايي سـمي و ميزان راديواكتيو كمتر در مقايسه با روش ماكسام و گيلبرت میباشد. مبناي روش سانگر كـاربرد ديدئوكسي نوكلئوزيد تريفسفات[22] بهعنوان ختمكنندة زنجيره DNA است. روش كلاسيك خـتم زنجيـرسـازي نياز به DNA تکرشتهای بهعنوان الگـو، آغـازگر، DNA پليمـراز، ديدئوكـسي نوكلئوتيـد تـريفـسفات و نوكلئوتيدهاي تغییریافته[23] براي خـتم طويـل شـدن رشـته DNA دارد. در ايـن روش ديدئوكـسي نوكلئوتيـد تـريفـسفات بهوسیله راديواكتيو نشاندار شده) معمولاً با (S35و روي ژل اكريل آميـد الكتروفورز و در خاتمه ژل از لايه شیشهای جدا میشود و يك فيلم راديولـوژي روي ژل قـرار میگیرد و بعـد از اثـرگـذاري راديواكتيو روي فيلم تفسير نتايج صورت میگیرد (شكل 2 الـف و ب). بعـدها بـا اسـتفاده از روش ديدئوكـسي نوكلئوتيـد تـريفـسفات نشاندار شـده بـا فلورسنس براي تعيين توالي و الكتروفورز روي ژل اكريلآميـد روي دستگاه، تعيين تـوالي نیمهخودکار شـد. در ايـن روش نمونه DNA براي واكنش تعيين توالي بـه چهـار گـروه مجـزا تقسيم میشود كه شامل تمام چهار نوع دئوكـسي نوكلئوتيـدهـاي پليمـراز استاندارد (dTTP و dCTP و dGTP و dATP) و DNA پلیمراز است. سپس در هر لوله واكنش فقـط يكـي از چهار ديدئوكـسي نوكلئوتيــد (ddTTP و ddCTP و ddGTP و ddATP) که نوكلئوتيدهاي ختمكننده زنجيره هستند اضافه میشود كـه ايـن نوكلئوتيدها فاقد گروه ‘OH3 لازم براي تـشكيل پيونـد فـسفودياستر بين دو نوكلئوتيد است. نكته مهم در اين روش استفاده بسيار كم از هر يك از ديدئوكـسي نوكلئوتيـد تـريفسفاتها در هر لوله بود تا زنجيره در حال ساخت بهطور اتفاقي ديدئوكـسي نوكلئوتيـد تـريفـسفات را بهجای دئوكـسي نوكلئوتيـد تـريفـسفات استفاده كرده و درنتیجه تكثير متوقف شود، بنابراين تكثير در محلهای مختلفي كه مثلاً باز) dATP در لوله حاوي (ddATP قرار دارد متوقف میشد. بدين ترتيب طويل شـدن رشـته DNA متوقـف شده و منجر به تشكيل قطعات DNA با طول متفاوت میشود. قطعات DNA نشاندار و تازه ساختهشده بهواسطه گرمـا واسرشت میشود و براساس انـدازه (بـا تفکیکپذیری فقـط1 نوكلئوتيـد) روي ژل آكريـلآميـد- اوره تغييـرشـكليافتـه، الكتروفورز میشود، بدين ترتيب كـه هرکدام از 4 واكـنش در يكي از 4 چاهكA ،C ، G و T بهطور جداگانـه الكتروفـورز میشود و سـپس بانـدهاي DNA بهوسیله تـصويربـرداري خودكار راديوايزوتوپي قابلمشاهده شـده و تـوالي DNA بهطور مستقيم از روي تصوير ژل يا فيلم راديولوژي قابلخواندن میشود. وجود هر خط يا قطعه يا باند تيره در يك خط عمودي تعیینکننده يك قطعه از تـوالي DNA اسـت كـه نتيجـه خـتم زنجيره بعد از تلفيـق يـك ديدئوكـسي نوكلئوتيـد (ddTTP و ddCTP و ddGTP و ddATP) است. در اين روش موقعيـتِ نسبي باندهاي متفاوت در بـين 4 مـسير عمـودي (از انتهـا بـه سمت بالا) براي خواندن توالي DNA استفاده میشود؛ يعنـي پایینترین باند، مربوط به همان بازي است كه در مسير آن ديـده شده است، بهطور مثال اگر پایینترین باند مربوط به مسير رشته A باشد نشان میدهد كه شروع با اين باز اسـت و اگـر دومـين باز نيز بالاتر از اين باز باشد نشان میدهد كه دومين باز نيـز A است. اگر باز بعدي در خانه مربوط بـه T باشـد بـاز بعـدي T خواهد بود، بنابراين براي سه باز خـواهيم داشـت AAT و بـه همين ترتيب خواندن به سمت بالا بين چهار خانـه ادامـه پيـدا میکند تا توالي تا حد امكـان خوانـده شـود و معلـوم شـود. از سوي ديگر، براي تعيين توالي ختم زنجيره میتوان از برچـسب نوكلئوتيــدهاي حــاوي فــسفر راديواكتيــو يــا از يــك آغــازگر[24] نشاندار در انتهــاي ‘5 بـا رنــگ فلورســنس بــراي نشانداركردن نيز استفاده كرد. تعيـين تـوالي بـا آغـازگر داراي رنگِ فلورسنس، خواندن در يك سيستم نوري را سریعتر كرده و امكان انجام خودكار را فراهم میآورد كه سـبب معرفـي ايـن روش بهعنوان روشي مقرونبهصرفه میشود. پیشرفتهای بعدي توسط لروي هوود[25] و همكارانش بـا بکارگیری ديدئوكـسي نوكلئوتيـد تـريفسفاتهای نشاندار و آغازگرها، زمينـه را بـراي انجـامِ خودكار اين روش توسط دستگاهها و تعيـين تـوالي DNA در حجم انبوه فراهم كرد (شكل 2-ج). در ايـن روش در ابتـدا از مواد رادیواکتیو استفاده میشد و بعدهـا شـركت Pharmacia (سوئد) در دستگاه تعيين توالي نيمهاتوماتيك ALF از تکرنگ فلوئورســين[26] و در دســتگاه ALF Express از رنگ CY5 استفاده كرد. در هر دو دستگاه لازم بود نمونهها در چهار خانه جداگانه ريخته شـود و جداسـازي بـا اسـتفاده از ژل اكريلآميد انجام میشد. در سالهای ابتدايي 90 ميلادي شـركت Applied Biosystems (آمريكا) دستگاهي را وارد بازار كرد كه با استفاده از چهار رنگ فلورسنتي و دستگاه ليزر متفاوت با طولموج مختلف تعيين توالي میکرد. در ايـن روش بهجای چهـار خانه براي يك نمونه از يك خانه استفاده میشد؛ بنـابراين چهـار برابر بيشتر از روش ALF میتوانستند نمونه را تعيين توالي كنند. اين دستگاه نيز به ژل اكريلآميد وابسته بود. اين روشها تا حد زيادي تعيين تـوالي DNA را سادهتر كرد، بهعنوان مثال در حالحاضر کیتهایی بـر اسـاس خـتم زنجيره بهصورت تجاري در دسترس است كه بهصورت آماده داراي معرفهای موردنیاز براي تعيينِ توالي اسـت؛ اگرچـه در اين روش محدودیتهایی مثـل غیراختصاصی بـودن محـل اتصال آغازگر به DNA يا ساختار دوم DNA میتواند ميـزان صحيح بودن نتايج بهدستآمده از توالي DNA را تحتتـأثير قرار دهد [10].
3: ختم زنجيرسازي با استفاده از رنگ:
در اين روش از نشانداركردن ديدزوكسي نوكلئوتيد ختمكننده زنجيره كه امكان تعيين تـوالي در يـك واكـنش واحـد را میدهد- بهجای چهـار واكـنش در روش آغـازگر نشاندار- استفاده میشود. در اين روش هر يـك از چهـار ديدزوكـسي نوكلئوتيد ختمكننده با يك رنگ فلورسنت نشاندار میشود كـه در طولموجهای مختلف هر يك از آنها نور منتشر میکند. با توجه بهسرعت بيشتر روش ختم زنجيرسـازي رنگـي، ايـن روش در حالحاضر روش اصلي تعيين توالي خودكـار اسـت. درعینحال ازجمله محدودیتهای اين روش میتوان به اثـر رنگ اشاره كرد كه باعث تفاوت در اتصال ختمكننده نشاندارشده به قطعات مختلف DNA میشود كـه درنهایت سـبب ارتفاع نابرابر و اشكال نابرابر در رنگنگار[27] حاصل از الكتروفورز مويين میشود (شكل 2- د)؛
شكل 2 – (الف) قسمتي از رادیوگرافی ژل قطعات تعيين توالي شده نشاندار (ب) آغازگرها با فلورسنت يا رادیواکتیو نشانهگذاری میشوند و درنهایت قطعه DNAبا ddNTP یا dNTPنشاندار میشود ج) نقاط اوج (Peaks) فلورسنس در مقايسه با باندهای رادیواکتیو د) الكتروفورز مویینهای
هرچند ايـن مشكل با تغيير در غلظت آنزيم DNA پليمراز و رنگ برطـرف شده است. اين كار موجب به حداقل رساندن تنوع در اتصال به زنجيره DNA و نيز روشي براي از بـين بـردن «حبـاب رنـگ» است. روش تعيين توالي ختم زنجيرسازي، همراه با آنـاليز تـوالي DNA بهصورت خودكار و توان عملياتي بالا در حال حاضر در اكثريت قريب بهاتفاق پروژههای تعيين توالي استفاده میشود.
4: تعيين توالي انبوه (نسل جدید تعیین توالی):
تعیین توالی ژنوم مرجع انسان در پروژه ژنوم (رفرنس) نتیجه سالها تلاش گروه کثیری دانشمند در سراسر دنیا و هزینههایی میلیون دلاری بود. تجربیات حاصل از کار در پروژه ژنوم باعث ایجاد تغییرات زیادی در روش کار گردید و تعیین توالی از روشهای مبتنی بر استفاده از کلونهای کروموزومی مصنوعی باکتریایی[28] به سمت استفاده از تعیین توالی کل ژنوم[29] رفت. تا مدتها پس از پروژه ژنوم، روشهای نوین نیز مبتنی بر تعیین توالی بهوسیله الکتروفورز کاپیلری بود، اما امروزه روشهای موسوم به روشهای نوین تعیین توالی بهسرعت در حال تجاری شدن هستند و در موارد تعیین توالی کل ژنوم و یا قسمت بزرگی از آن استفاده میگردند. این روشها رویکرد جدیدی همراه با هزینه و زمان کمتر در رسیدن به نتیجه نهایی را ممکن ساختهاند. سه روش اصلی مورد استفاده در تعیین توالی انبوه شامل روش 454[30]، ایلومینا[31] و بیوسیستم سولید[32] میباشند. دو روش هلیکوس[33] و ریلتایم[34] نیز در حال مطرح شدن و رواج میباشد.
الف- تعيين توالي به روش ايلومينا:
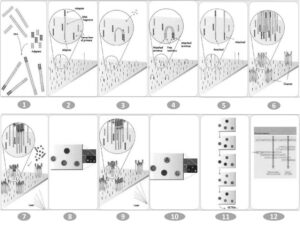
این روش شامل چهار مرحله اصلی آمادهسازی کتابخانه ژنی، ایجاد کلاستر ژنی، تعیین توالی و آنالیز دادهها میباشد. (شکل 3) [11]. اين روش تعيين توالي با استفاده از خاصيت رنگهای پاياندهنده ولي برگشتپذیر[35] طراحي شده است. ابتدا قطعات شكسته شده مولكول DNA به آغازگرهاي متصل به اسلايد وصل و سپس تكثير میشود. درنهایت کلونیهای متمركـز ايجـاد شـده و پلهایی[36] تشكيل میدهد، سپس
شکل 3- مراحل طی شده در روش تعیین توالی ایلومینا
1: فراهم کردن کتابخانه 2: اتصال نمونه ژنی به سطح فلوسل 3: تکثیر به روش پل 4: قطعات دورشتهای شده 5: واسرشته شدن مولکولهای دورشتهای 6: اتمام تولید خوشهها 7: اولین مرحله تعیین توالی 8: اولین مرحله عکسبرداری 9: دومین مرحله تعیین توالی 10: مرحله دوم عکسبرداری 11: چندین چرخه عکسبرداری 12:اطلاعات حاصل از عکسبرداری
چهار نوع بـاز متـصل به رنگهای پاياندهنده ولـي برگشتپذیر[37] بـه واكنش افزوده میشود و هر باز درصورتیکه محـل مناسـبي براي اتصال وجود داشته باشد، به آن ناحيه متـصل میشود و سپس نوكلئوتيدهاي آزاد شسته شده و از محيط واكنش حـذف میشود و تنها يك نوكلئوتيد در هر مرحلـه بـه DNA اضـافه میشود. در اين لحظه دوربيني از نوكلئوتيدهـاي نشاندار شـده با فلورسنت تصويربرداري مینماید. در پايـان متوقـفكننـده رشد DNA از سر ‘3 توسط واکنشهای شيميايي جدا میشود و چرخه بعدي آغاز میشود [12] (شكل 3). اين روش توسـط شركت Solexa (آمريكا) ابداع شد و با توجه به اینکه ايـن شركت هماکنون بخشي از شركت Illumina (آمريكا) است، بـه روش تعيين تـــــــوالي ايلومينا[38] معروف شد.
ب- تعيين توالي به روش SOLiD:
در اين روش پيش از تعيين توالي، DNA توسط دانههای پوشيده از آغازگر در حبابهای روغني[39] تكثير میشود. ايـن روش امولـسيون[40]PCR ناميده میشود. هرکدام از ايـن حبابها حاوي يك نسخه از مولکولهای DNA مـشابه اسـت كه بر سطح اسلايد شیشهای قرار دارد. اليگونوكلئوتيدهاي دورشتهای باز میشود و درصورتیکه جایگاههای مشابه براي اتصال پيدا كند به آن متـصل میشود. لازم به ذكر است كه نتيجه تعيين توالي، كيفيت و طول قطعـات در اين روش، قابلمقایسه با روش ايلومينا است (شکل 4).
شکل 4 – روش تعیین توالی SOLID
(الف) قطعات پرایمر برای همانندسازی بهمنظور تعیین توالی به قطعات نمونه ژنی مجهول متصل به قطعات آداپتور وصل میشوند. لازم به ذکر است که آداپتورها از قبل به دانهها وصل شدهاند تا باعث تثبیت تمامی قطعات بروی دانهها گردند. 4 نوع پروب بهکاررفته بهمنظور توالییابی که هرکدام به یک رنگ فلوئورسنس خاص متصل میباشند که پس از اتصال هرکدام عامل برشدهنده انتهای پروبها را برش داده و ادامه چرخه بهمنظور تعیین توالی را میسر میکند. (ب) هر باز در سیستم 2 بار توسط 2 پرایمر مختلف خوانده میشود تا توسط دستگاه بهعنوان خوانشی از آن جایگاه در نظر گرفته شود.
ج- روش تعيين توالي حرارتي (454):
يكي از روشهایی اسـت كـه بهموازات روش تعيـين تـوالي حرارتی [41]توسط شركت 454 Life Sciences (آمريكا) پيشنهاد شد و اين روش بعدها توسط شركت Roche (سوئيس) خريداري شد. در اين روش قطعات DNA در داخل قطرات آب در يك محيط روغنـي تكثيـر میشود )امولـسيون (PCR در هرکدام از اين قطرات آب يك الگوي DNA قـرار دارد كه به يك دانه حاوي آغازگر متصل میشود و درنهایت بر اثر تكثير آن يـك كلـوني فـشرده ايجـاد میشود. دسـتگاه توالیخوان حاوي تعداد زيادي چاهك با حجم در حد پيكوليتر است كه هرکدام از اين چاهکها حـاوي يـك دانـه و آنـزيم مخصوص تعيين توالي است. ايـن سيـستم از آنـزيم لوسـيفراز[42] كه توليد نور میکند بـراي تـشخيص هر نوكلئوتيد كه بـه رشـته DNA تـازه تـشكيلشـده افـزوده میشود، استفاده میکند [13]. اين روش نسبت بـه روش تعيـين توالي سانگر، دقت خوانش بيشتر و هزينه كمتري دارد؛ هرچند كه روشهای Solexa و SOLiD از ايـن نظـر داراي برتـري هستند [14] (شکل 5).
شکل 5- روش تعيين توالي حرارتي (454)Pyrosequensing
پس از تشکیل کتابخانه از قطعات، تکثیر قطعات ژنی در داخل قطرات آب در یک محیط روغنی صورت میگیرد. داخل هر قطره آب دانهای پوشیده از پرایمر به همراه یک نمونه ژنی قرار میگیرد و سپس نمونه ژنی متصل به پرایمرهای روی دانه با روش PCR ایجاد کلونی از نمونه ژنی روی دانه میکند. دستگاه توالیخوان دارای چاهکهایی است که حاوی 1 دانه و تعداد زیادی آنزیم لوسیفراز به همراه تعداد زیادی آنزیم پلیمراز میباشد. هر بار با اضافه شدن نوکلئوتیدهای مشخص در صورت همخوانی با توالی و اتصال، تولید pp آزاد که به کمک سولفوراز باعث تولید نور توسط آنزیم لوسیفراز شده و دستگاه توالی را مشخص مینماید.
د- روش (Single Molecular Real Time) SMRT:
شركت PacBio (كانادا) ابداعکننده روش تعيـينِ تـوالي[43]SMRT است. اين روش براساسِ فرآيند طبيعي همانندسازي DNA طراحي شده كـه از كارآيي و درستي بالايي برخـوردار اسـت. فنآوری SMRT مشاهده سنتز (ساخت) DNA را در همان زمان وقوع امکانپذیر میسازد. در اين روش آنـزيم DNA پليمـراز در تـه چاهــك كــه[44] ZMWناميــده میشود، ثابت میگردد، سـپس بـر اسـاس رشـته الگـو، از ميـان انبوهي از نوكلئوتيدهاي نشاندار شده بـا فلورسـنت، نوكلئوتيـد مناسـب انتخـاب شـده و سـنتز انجـام میشود و بـر اسـاس فلورسنتِ آزادشده توالي خوانده میشود [15]. تعيين توالي بر اساس اين روش داراي مزاياي زير است:
1) امكان تجزيه و تحليل منحصربهفرد تك مولكول DNA و تشخيص تنوع توالي خاص از يك سلول به سلول ديگر
2) امكان خوانش توالیهای طولاني بيشتر از 1000 جفـت باز كه امكانِ تعيين نقش ژني و جمع كردن كليه اطلاعات توالي موردنظر را تسهيل میکند.
3) نتيجه تعيين توالي میتواند در يك روز آماده شود.
5: روشهای کمکاربرد ولي مطرح:
الف-نانومنفذ (Nanopore):
اين روش بر اساس بازخواني پیامهای الكتريكـي ناشـي از عبور نوكلئوتيدها از منافذ آلفاهمـولزين[45] پوشيده از سـيكلودكـسترين[46] طراحـي شـده است. هنگامي كه DNA از حفرهها عبور میکند جريـان يـوني آن تغيير میکند. اين تغييرات بر اسـاس شـكل، انـدازه و طـول توالیهای DNA متفاوت است. هر نـوع از ايـن نوكلئوتيـدهـا هنگام عبور از حفره، با دورههای زماني متفاوت جريان یونها را قطع میکند (شـكل 6). ايـن
شكل 6 – روش تعيين توالي DNA نانومنفذ
بازخواني پیامهای الكتريكي ناشي از عبور نوكلئوتيدها از منافذ، نمونه ژنی با عبور از بین پروتئینهای آلفا همولزين پوشيده از سيكلودكسترين هلیکسش باز و از بین سوراخی میکروسکوپی از خلال غشا عبور میکند، همزمان با عبور نمونه ژنی از خلال پروتئین، یونها نیز از آن میان جریان هر باز در نمونه با سد کردن جریان عبور یونها میزان متفاوتی تغییر جریان ایجاد میکنند که نوع باز را مشخص مینماید.
روش از آنجـا كـه نيـاز بـه نوكلئوتيد تغييرشكليافته ندارد، قابليـت توسـعه را دارد ولـي هنوز قدرت تشخيص در حد يك نوكلئوتيد را ندارد.
ب- Heliscope:
روش تعيين توالي Heliscope بر اسـاس فنآوری “تـوالي درست مولكـولي[47]” از قطعات DNA متصل به آداپتورهايي با دنباله پلي A براي اتـصال به سطح فلوسل[48] اسـتفاده مینماید. مرحلـه بعـد شامل تعيين توالي مبتني بر طويـلسـازي، بـا شستـشوي دورهای سلولهای در جريان، با افـزودن نوكلئوتيـدهـاي نشاندار اسـت (مشابه روش سانگر- يك نوع نوكلئوتيد در هـر مقطـع زمـاني) (شکل 7). دستگاه Helioscope میتواند در هر خوانش 55 باز را بخوانـد، امــا بــا پیشرفتهای جديــد ايــن فنآوری درســتي خــوانش هوموپليمرها[49] و تعيين تـوالي RNA افـزايش يافته است.
شکل 7- روش تعيين توالي Heliscope
1) همانندسازی رشتهها با تکنوکلئوتیدهای اضافه شده حاوی رنگ فلئورسنس و خاتمهدهنده واکنش 2) شستشوی نوکلئوتیدهای متصل نشده و اضافی 3) عکسبرداری از خوشههای قطعات تکثیرشده که به علت اتصال رنگ فلئورسنس خاص قابلشناسایی هستند 4) جداسازی مواد خاتمهدهنده واکنش از انتهای نوکلئوتیدها و ادامه همانندسازی و تعیین توالی
کاربردهای نسل جدید تعیین توالی در پزشکی امروز:
از مزایای روشهای نوين تشخیص بیماریهای ژنتیک نسبت به روشهای سنتی ژنتيک تشخيصی قابلیت این روشها در تشخيص مواردی از بيماری است که در آن تشخيص دقيق بيماری مشخص نیست و تنها برخی علائم کلی آن مانند عقبماندگی و معلوليت مشخص است. همچنین در بیماریهایی که ژنهای متعددی در بروز بیماری دخیل هستند، با استفاده از روشهای نوین تشخیصی، هزینه بررسی و زمان موردنیاز برای حصول جواب نسبت به روشهای سنتی بهطور چشمگیری کاهش مییابد.
روشهای مورد استفاده جهت بررسی بیماریهای ارثی:
- Targeted exome sequencing: بررسی مجموعهای از ژنهای شناخته شده
- Whole exome sequencing: بررسی کل نواحی کدکننده ژنوم با استفاده از روشهای نسل جديد تعيين توالی. در این روش ژنهایی که تاکنون برای بیماری موردنظر معرفی نگردیدهاند نیز موردبررسی قرار میگیرد.
- Whole genome sequencing: بررسی کل ژنوم با استفاده از روشهای نسل جديد تعيين توالی. در این روش علاوه بر نواحی کدکننده پروتئین، نواحی تنظیمی نیز ازنظر وجود واریانتهای عامل بیماری موردنظر بررسی میگردد.
- بررسی کامل ژن با استفاده از روش تعیین توالی مستقیم
در حال حاضر پانلهای تشخيصی برای بررسی اختلالات ژنتیکی هتروژن مندلی[50] توسط پانل ژنی[51] با استفاده از روشهای نوين تعيين توالی[52] قابلارائه میباشد. در صورت عدم امکان استفاده از پانل اختصاصی برای یک بیماری، امکان بررسی همه ژنها و نواحی کدکننده با تعیین توالی همه اگزونها (Whole Exome Sequencing) همراه با بررسی بیوانفورماتیک تغییرات ژنومی حاصله، تفسیر بالینی و تأیید با روش تعیین توالی مستقیم[53] فراهم میباشد.
منابع:
- Olsvik, O., et al., Use of automated sequencing of PCR-generated amplicons to identify three types of cholera toxin subunit B in Vibrio cholerae O1 strains. Journal of Clinical Microbiology, 1993. 31: p. 22-25.
- Points to consider in the clinical application of genomic sequencing. Genet Med, 2012. 14(8): p. 759-61.
- Fiers, W., et al., Complete nucleotide sequence of bacteriophage MS2 RNA: primary and secondary structure of the replicase gene. Nature, 1976. 260(5551): p. 500-507.
- Jou, W.M., et al., Nucleotide sequence of the gene coding for the bacteriophage MS2 coat protein. Nature, 1972. 237: p. 82-88.
- Pettersson, E., J. Lundeberg, and A. Ahmadian, Generations of sequencing technologies. Genomics, 2009. 93(2): p. 105-111.
- Maxam, A.M. and W. Gilbert, A new method for sequencing DNA. Proceedings of the National Academy of Sciences, 1977. 74(2): p. 560-564.
- Gilbert, W. and A. Maxam, The nucleotide sequence of the lac operator. Proceedings of the National Academy of Sciences, 1973. 70(12): p. 3581-3584.
- Sanger, F. and A.R. Coulson, A rapid method for determining sequences in DNA by primed synthesis with DNA polymerase. Journal of molecular biology, 1975. 94(3): p. 441-448.
- Sanger, F., S. Nicklen, and A.R. Coulson, DNA sequencing with chain-terminating inhibitors. Proceedings of the National Academy of Sciences, 1977. 74(12): p. 5463-5467.
- Daneshpour, M.S., M.S. Fallah, and P. Eshraghi, Revolution of DNA Sequencing Method from the Past until Today. Modares Journal of Medical Sciences: Pathobiology, 2014. 16(4): p. 1-13.
- Mardis, E.R., Next-generation DNA sequencing methods. Annu. Rev. Genomics Hum. Genet., 2008. 9: p. 387-402.
- Church, G.M., Genomes for all. Scientific American, 2006. 294(1): p. 46-54.
- Margulies, M., et al., Genome sequencing in microfabricated high-density picolitre reactors. Nature, 2005. 437(7057): p. 376-380.
- Hall, N., Advanced sequencing technologies and their wider impact in microbiology. Journal of Experimental Biology, 2007. 210(9): p. 1518-1525.
- Blow, N., Metagenomics: exploring unseen communities. Nature, 2008. 453(7195): p. 687-690.
[1] PND
[2] PGD
[3] Gold standard
[4] NGS:Next Generation Sequencing
[5] High Throughput
[6] genetic heterogeneity
[7] Walter Fiers
[8] Frederick Sanger
[9] Walter Gilbert
[10] Allan Maxam
[11] Wandering Spot Analysis
[12] Chemical cleavage
[13] Chain-termination
[14] High Throughput
[15] Dye-termination
[16] Pyrosequensing 454
[17] Nanopore
[18] Depurinated
[19] Piperidine
[20] Denaturing Acrylamide
[21] Methylation Interference Assay
[22] ddNTP
[23] dideoxyNTPs
[24] Primer
[25] Leroy Hood
[26] Fluorescein
[27] Chromatogram
[28] BAC clone
[29] Whole genome sequencing
[30] Roche/454
[31] Illumina/Solexa Genome Analyzer
[32] Applied Biosystem SOLiD TM System
[33] Helicos Heliscope
[34] Single molecule real time sequencing
[35] Reversible Dye-Terminators
[36] Bridge Amplification
[37] RT-bases
[38] Illumina (Solexa) Sequencing
[39] Aqueous droplets within an oil phase
[40] Emulsion PCR
[41] Pyrosequencing
[42] Luciferase Enzyme
[43] Single Molecular Real Time
[44] Zero-Mode Waveguide
[45] Alpha Hemolysin
[46] Cyclodextrin
[47] True Single Molecule Sequencing
[48] Flow Cell
[49] Homopolymer
[50] locus heterogeneity)
[51] Targeted Exome Sequensing
[52] NGS
[53] Sanger Sequencing
ژنومیکس و کاربرد آن در تشخیص بیماریها (2)
ژنومیکس و کاربرد آن در تشخیص بیماریها
برای دانلود پی دی اف بر روی لینک زیر کلیک کنید
ورود / ثبت نام