پيروسكوئنسينگ
دکتر مهدی فصیحی رامندی (عضو هیئت علمی دانشگاه علوم پزشکی بقیه ا…(عج))
زهرا کریمی (مرکز تحقیقاتی زیست سلول پژوهان تدبیر)
دکتر رضا میرنژاد (دانشیار دانشگاه)
پيروسكوئنسينگ[1] يك تكنيك جديد تعيين توالي DNA بر پايۀ ساخت زنجيره و بر اساس بيولومينومتري غیرمستقیم[2] ميباشد. در اين تكنيك توالي قطعه موردنظر با استفاده از سيستم آبشار آنزيمي در زمان واقعي[3] تعيين ميشود. در اين سيستم چهار آنزيم (قطعه كلنو فاقد فعاليت اگزونوكلئازي، آپيراز، ATP سولفوريلاز و لوسيفراز) با سوبستراي خاص خود استفاده میگردد.
مکانیسم توالیخوانی:
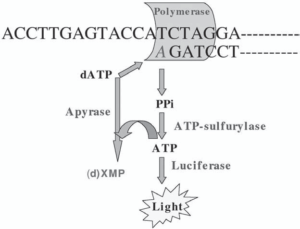
طبق پروتكل كاري، چهار نوكلئوتيد بهطور متوالي به محلول واكنش اضافه ميشوند. اگر نوكلئوتيدي مكمل الگو بود، به زنجيرۀ در حال ساخت اضافه شده و يك مولكول پيروفسفات آزاد ميشود. PPi و آدنوزين ‘5 فسفوسولفات (APS) بهعنوان سوبستراي آنزيم ATP سولفوريلاز ميباشند كه نتيجۀ اين واكنش توليد ATP ميباشد. آنزيم ديگر اين مجموعه يعني لوسيفراز از ATP و لوسيفرين استفاده كرده، اكسيلوسيفرين، AMP، Ppi و نور توليد ميكند. اين نور توسط دستگاه لومينومتر[4] يا وسايل مشابه اندازهگيري ميشود. ميزان نور حاصل بستگي به تعداد نوكلئوتيد اضافه شده به پرايمر دارد. همين موضوع موجب شده كه تكنولوژي تعيين توالي Pyro به روش تعيين توالي كمّي منحصر به فردي تبديل شود. نوكلئوتيدهاي اضافه، قبل از اضافه شدن نوكلئوتيد بعدي، توسط آنزيم آپيراز[5] موجود در واكنش از بين ميروند. استفاده از يك پيكومول DNA موجب توليد مولكول ATP، از اتصال يك نوكلئوتيد به رشتۀ الگو ميشود. در نتيجه در حالت فوق در اثر اتصال يك نوكلئوتيد، فوتون با طولموج 560 نانومتر توليد میگردد. اين ميزان نور توليدي، بهراحتی با ديودهاي نوري، فتومولتيپلاير و يا دوربين CCD قابل اندازهگيري است (شکل 1).
پيروسكوئنسينگ داراي مزايايي از جمله صحت بالا، انعطافپذيري، آزمایش همزمان چندین نمونه، و اتوماسيون آسان ميباشد. در این روش به لکهگذاری، الکتروفورز و یا هر تکنیک دیگری برای جداسازی قطعات نیاز نیست (برخلاف روش ختم زنجیره[6] (روش سانگر) که نیاز به الکترفورز پلیآکریل آمید دارد) و بنابراین سریعتر از روش ختم زنجیره است. همچنين در اين تكنيك از پرايمرها و نوكلئوتيدهاي نشاندار استفاده نميشود. اين تكنيك براي تعيين توالي DNA، تعيين توالي cDNA، تعيين جهشهای ژنی دخيل در بيماري، تعيين تيپ ميكروبها و تعيين ژنوتيپ SNP كاربرد دارد. اين تكنيك اخیراً با بکارگیری پروتئين متصلشونده به DNA تکرشتهای[7] (SSB) بهتر شده است، زيرا اين پروتئين مانع از تشكيل ساختار دوم در DNA الگو ميشود.
در هر دور آزمایش این روش، تنها تا 150 جفت باز را میتوان تعیین توالی کرد که شاید گرچه در نگاه اول کماهمیتتر از روش ختم زنجیره با 750 توالییابی در هر دور آزمایش به نظر برسد، اما این مزیت در این روش وجود دارد که قابلیت ماشینی شدن را دارد؛ بنابراین با این روش توالیها بسیار سریعتر از روش ختم زنجیره ایجاد میشوند که این موضوع نشان میدهد چرا این روش، بهتدریج، روش ترجیحی برای پروژههای ژنومی میشود.
شکل 1: تصوير شماتيك از فعاليت آنزيمي در تكنيك پيروسكوئنسينگ
DNA الگو و پرايمر و چهار آنزيم موردنیاز اين تكنيك، به چاهك پليت ميكروتيتر اضافه ميشود. چهار نوكلئوتيد مختلف طي مراحل مجزا، به واكنش اضافه ميشود. نوكلئوتيد به الگو متصل شده و واكنش با كمك آنزيمهاي ATP سولفوريلاز و ليگاز ادامه پيدا ميكند. نوكلئوتيدها بهطور مدام توسط آنزيم آپيراز تجزيه ميشوند تا اجازۀ افزودن نوكلئوتيد بعدي داده شود. (d)XMP شامل AMP و dNMP است
روش پربازده سازی پیروسکوئنسینگ:
معمولاً برای استفاده از این روش در سطح وسیع و توالییابی DNA ژنومی، ابتدا ژنوم را به قطعات بین 300 تا 500 جفت بازی میشکنند. سپس به دو انتهای هر قطعه، یک آداپتور وصل میکنند. نقش اول این آداپتورها در وهله اول این است که DNA را به ذرات فلزی وصل کنند؛ بدین طریق که سر یکی از این آداپتورها، در سمت ‘5 رشته، بیوتینیله شده است که میتواند به ذرات پوشیده شده با استرپتوآویدین متصل شود (بیوتین تمایل بالایی برای اتصال به استرپتوآویدین دارد). نسبت قطعات DNA به این ذرات به نحوی تنظیم میشود که بهطور متوسط یک قطعه به یک ذره وصل میگردد. در مرحله بعد این قطعات توسط PCR تکثیر میشوند تا تعداد نسخه کافی برای تعیین توالی، تولید شود. آداپتور در این مرحله نقش دوم خود بهعنوان جایگاه اتصال پرایمر را ایفا میکند و چون آداپتورهای همه قطعات یکسانند از یک جفت پرایمر یکسان میتوان برای همه قطعات استفاده کرد.
مسلماً اگر PCR بلافاصله انجام بگیرد تنها چیزی که بدست میآید مخلوطی از همه محصولات خواهد بود که برای بدست آوردن توالی هر قطعه مناسب نیست. بهمنظور حل این مشکل، PCR در یک امولسیون روغنی انجام میگیرد که در آن، هر ذره به همراه قطره آبکی مربوط به خودش درون امولسیون قرار میگیرد. هر قطره آبکی تشکیل شده است از کل مواد موردنیاز برای PCR و بهصورت فیزیکی توسط سدی که امولسیون فراهم میکند، از سایر قطرهها جدا شده است.
بعد از انجام PCR، این قطعات آبکی به چاهکهای یک نوار پلاستیکی منتقل میشوند، بهنحویکه تنها یک قطره (یعنی محصول یک PCR) در هر چاهک قرار میگیرد، سپس واکنش پیروسکوئنسینگ، در هر چاهک انجام میشود.
تعيين ژنوتيپ SNP با استفاده از تكنولوژي پيروسكوئنسينگ:
اين تكنولوژي توسط يك شركت سوئدي به نام Biotage AB بهصورت تجاري درآمد. اين شركت مجموعهاي از مواد، نرمافزار، دستگاه و سيستمها را براي آزمايش بر روي 96 نمونه بهطور موازي و بر روي فاز جامد توليد و عرضه نموده است. اين روشها، براي آمادهسازي و انجام آزمايش بهصورت سريع[8] طراحي شدهاند. همچنين اين روش طوري است كه قابليت اتوماتيك شدن را داراست. نرمافزار آن نيز طوري طراحي شده است كه بتواند انواع SNP و جهشها را در نقاط مختلف و همچنين در نمونههاي مختلف، بهطور همزمان شناسايي كند[9]. اين نرمافزار همچنين قابليت تشخيص چندين SNP در يك نمونه، بررسي حذف و اضافه، مطالعه كمّي فراواني آلل و تعيين ژنوتيپ قطعات كوچك در حد 40-20 بازي را دارد. در اين تكنيك احتياجي نيست كه انتهاي ‘3 پرايمر در نزديكي SNP قرار گیرد. اين انعطافپذيري پرايمر، يك مزيت براي اين روش محسوب ميشود.
در اين بخش مراحل آمادهسازي الگو، پيروسكوئنسينگ و نحوۀ استفاده از اين تكنيك در آناليز SNP بيان ميشود.
اندازهگيري فراواني آلل
سه موضوع مهم مطرح در نقشهبرداري ژنتيكي بر پايه SNP عبارت است از: هزينه تعيين ژنوتيپ در سطح گسترده، صحت اندازهگيري و امكان بكارگيري تعيين هاپلوتيپ مستقيم (مولكولي) بهمنظور افزايش قدرت پيشگويي.
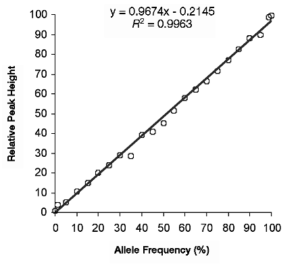
مطالعات عدم پيوستگي بر روي نمونه افراد، نياز به تعيين ژنوتيپ وسيعي دارد كه اين امر نيز بهشدت هزينهبر است، اما يك روش جايگزين و فرعي، اندازهگيري آللهاي SNP در يك حوضچۀ DNA[10] ميباشد. اين روش تعيين ژنوتيپ، كمّي است؛ زيرا ارتفاع نمودار[11] به تعداد بازهاي متصلشده بستگي دارد. با استفاده از اين امر ميتوان هتروزيگوتها را بهدقت تعيين كرد. همچنين با استفاده از اين نمودار، فراواني آللهاي موجود در استخر حاوي انواع DNAها را نيز ميتوان محاسبه نمود. نمونۀ اين نمودار را در شكل زير مشاهده ميكنيد:
در اين نمودار، ارتباط بين فراواني آلل يك SNP و ارتفاع قله پيروگرام[12]، در نمونۀ حاوي DNA دو فرد هموزيگوت به تصوير كشيده شده است.
شکل 2: نمودار خط رگرسیون بین فراوانی آلل و ارتفاع قله پیروگرام حاصل از مطالعه بر روی دو فرد هموزیگوت برای یک SNP مشخص. هر نقطه میانگین دو آزمایش میباشد
تعيين هاپلوتيپ
قدرت و توانايي تعيين ژنوتيپ SNP در نقشهبرداري ژنتيكي ميتواند گستردهتر شده و به تعيين و ساخت هاپلوتيپ ارتقاء يابد. تكنيكهاي مولكولي از قبيل تعيين توالي Pyro براي ساخت هاپلوتيپ مناسب هستند، زيرا با استفاده از اين تكنيكها، ميتوان چندين SNP نزديك به هم را بهآسانی شناسايي كرد. يك روش تعيين هاپلوتيپ، استفاده از PCR اختصاصي آلل[13] به همراه تكنيك تعيين توالي Pyro ميباشد. در اين موارد استخر DNA، تكثير اختصاصي آلل هاپلوتيپ و تكنولوژي تعيين توالي pyro همه در كنار هم، براي تعيين دقيق فراواني هاپلوتيپ بكار ميروند. بهطور مشابه از روش تعيين توالي pyro براي بررسي چندين SNP موجود بر روي قطعات تکثیرشده استفاده ميشود. قرار دادن يك باز غير مكمل[14] در دومين باز انتهاي ‘3 موجب افزايش اختصاصيت PCR ميشود. تعيين ژنوتيپ نمونههاي هتروزيگوت، بعد از انجام AS-PCR، يك الگوي تك آللي از هر SNP ارائه ميدهد. از آنجا كه تعيين توالي pyro يك روش كمّي است، بنابراين هرگونه تكثير غیراختصاصی آللها مشخص ميشود. يكي از محدوديتهاي اين تكنيك عدم شناسايي مستقيم هاپلوتيپ است. براي شناسايي هاپلوتيپ، ابتدا بايد AS-PCR انجام شود. انتظار ميرود كه اين مشكل نيز در آينده مرتفع گردد.
مواد لازم جهت اجرای پيروسكوئنسينگ:
آمادهسازي نمونه:
- نمونۀ لازم براي واكنش پيروسكوئنسينگ بهوسیلۀ PCR توليد ميشود. يكي از پرايمرهاي مورد استفاده در اين PCR بيوتينيله ميباشد.
- ذرات مغناطيسي پوشيده شده با استرپتاويدين
- بافر اتصال/شستشو:
10 mM Tris-HCl, pH 8.0
2 M NaCl
0.1% Tween 20
اين بافر براي اتصال و تثبيت محصول بيوتينيلۀ PCR به سطح ذرات مغناطيسي و شستشوي آن كاربرد دارد.
- پرايمر تعيين توالي (يكي از پرايمرهاي PCR را ميتوان بهعنوان پرايمر تعيين توالي بكار برد، البته اين امر به شرطي امكانپذير است كه SNP در نزديكي پرايمر قرار داشته باشد).
واكنش پيروسكوئنسينگ:
- محلول آنزيمي:
5 U exonuclease-deficient Klenow DNA polymerase
40 mU apyrase
500 ng purified luciferase
15 mU purified recombinant ATP sulfurylase
اين مخلوط را ميتوان بهصورت ليوفيليزه به مدت طولاني ذخيره كرد و قبل از استفاده بهصورت محلول درآورده و رقيق كرد.
- مخلوط سوبسترا:
5 mM magnesium acetate
0.1% bovine serum albumin (BSA)
1 mM dithiothreitol (DTT)
5µM adenosine 5′-phosphosulfate (APS)
0.4 mg/mL polyvinylpyrrolidone (360,000)
100 µg/mL D-luciferin
اين مخلوط حساس به نور بوده و ميتوان آن را بهصورت ليوفيليزه و يا در دماي 20- درجه سلسیوس به مدت حداقل يك سال نگهداري نمود.
- µL20 از نوكلئوتيدهاي dCTP, dGTP, dATP-αS و dTTP با غلظت M0/1. هر كدام از اين نوكلئوتيدها با Lµ1 از پيروفسفات mM100 تيمار ميشوند تا پيغامهاي غیراختصاصی حاصل از آلودگي با PPi حذف شوند. اين نوكلئوتيدها رقيق شده و به Lµ1000 ميرسند و ميتوان آنها را در دماي 4 و يا 20- درجه سلسیوس به مدت حداقل يك سال نگهداري نمود.
- پليتهاي ميكروتيتر: اگر از سيستم PSQ96 براي پيروسكوئنسينگ استفاده ميشود، بايد از پليتهاي مخصوص استفاده كرد.
- Inkjet cassette: از اين كاست براي انتقال آنزيمها و سوبسترا استفاده ميشود. همۀ مواد ذکرشدۀ فوق بهصورت تجاري وجود دارند.
دستگاه موردنیاز:
- دستگاه پيروسكوئنسينگ: دو نوع دستگاه PSQ96 و PTP384 امروزه بهصورت تجاري براي اين كار وجود دارد.
- رك مغناطيسي براي رسوب ذرات مغناطيسي
روش كار:
آمادهسازي نمونه:
- تثبيت Lµ40-50 از محصول بيوتينيلۀ PCR بر روي gµ200 از ذرات مغناطيسي پوشيده از استرپتواويدين. قبل از تثبيت، اين ذرات در Lµ40-50 از بافر اتصال/شستشو به مدت 15 دقيقه در دمای 43 درجه سلسیوس قرار گرفته و بهآرامی هم زده ميشوند.
- ذرات را سانتريفيوژ كرده و مايع رويي را خارج نماييد. ذرات حاوي نمونه را در Lµ20 از NaOH با غلظت M0/1 به مدت 5 دقيقه در دماي اتاق و يا در دماي 43 درجه سلسیوس انكوبه كنيد تا DNA کاملاً تکرشتهای شود.
- بعد از شستشو، رشتۀ تثبیتشده را در بافر اتصال پرايمر به حالت سوسپانسيون درآورید.
بافر اتصال پرايمر:
10 mM Tris-HCl
2 mM MgAC2, pH 7.5
اين بافر همچنين حاوي pmol10 پرايمر تعيين توالي است. حجم نهايي در اين مرحله Lµ10 ميباشد.
- بايد پرايمر تعيين توالي به DNA الگو متصل شود. بدين منظور محلول فــوق را در دماي ̊ 94 سلسیوس به مدت 20 ثانيه و ̊ 65 سلسیوس به مدت 2 دقيقه انكوبه كنيد. پس از آن محلول را در دماي اتاق سرد نماييد.
واكنش پيروسكوئنسينگ:
- DNA الگو حاوي پرايمر تعيين توالي را به يك پليت ميكروتيتر منتقل كرده و با بافر TEA حجم آن را به Lµ50 برسانيد.
بافر TEA:
100 mM Tris-Acetate, pH 7.75
0.5 mM EDTA
- مخلوط آنزيمي، مخلوط سوبسترا و نوكلئوتيدها را به درون كارتريج inkjet ريخته و درون دستگاه پيروسكوئنسينگ قرار دهيد.
- دستگاه پيروسكوئنسينگ بهطور اتوماتيك واكنش را آغاز كرده و مخلوط آنزيمي، سوبسترا و نوكلئوتيدها را طبق برنامه اختصاصي SNP به واكنش اضافه ميكند. SNP بهطور اتوماتيك مشخص خواهد شد.
دستگاه پيروسكوئنسينگ:
دستگاه پيروسكوئنسينگ اتوماتيك از يك كارتريج inkjet براي انتقال دقيق حجمهاي در حد كمتر از ميكروليتر استفاده ميكند. اين كارتريج توانايي انتقال شش ماده به درون پليت ميكروتيتر را دارد. اين پليت بهطور مدام هم زده ميشود تا سرعت واكنش افزايش يابد. يك سيستم لنزي نيز در دوربين CCD وجود دارد كه بهطور مؤثری لومينسانس توليدشده در هر چاهك را پيگيري ميكند. نوكلئوتيدها با تأخیر به درون چاهك بعدي ريخته ميشود تا نور حاصل از چاهك قبلي اشكالي در آزمايش ايجاد نكند. دوربين CCD بهطور منظم هر ثانيه يك عكس از پليت ميگيرد تا فرايند دقيق واكنش پيروسكوئنسينگ پيگيري شود. دادههاي حاصل توسط يك نرمافزار به كامپيوتر منتقل ميشوند. توسط اين نرمافزار ميزان نوكلئوتيدهايي كه به هر چاهك وارد ميشود را ميتوان كنترل كرد.
آناليز دادهها:
دادههاي خام توسط دستگاه پيروسكوئنسينگ گردآوري ميشود. اين دادهها بهصورت كمّي است، زيرا ميزان نور حاصل از واكنش، نسبت مستقيمي با تعداد مولكول PPi دارد. تعداد مولكول PPi نيز نشاندهندۀ تعداد نوكلئوتيد متصلشده به رشتۀ در حال ساخت ميباشد، بنابراين پيغام نوري حاصل، نشاندهندۀ تعداد نوكلئوتيد متصلشده به رشتۀ در حال ساخت ميباشد. هماکنون نرمافزارهاي متفاوتي براي آناليز انواع پليمورفيسمها وجود دارد كه بسته به نوع مطالعه ميتوان از آنها استفاده كرد.
نكات قابلتوجه در اين تكنيك:
- ميزان DNA الگو براي انجام يك واكنش پيروسكوئنسينگ موفق، حياتي است. براي انجام پيروسكوئنسينگ با سيستم PSQ96، حداقل يك پيكومول از DNA الگو موردنیاز ميباشد.
- خلوص نوكلئوتيدها از اهمیت خاصي برخوردار است. اگر نوكلئوتيدها خالص باشند، تعيين توالي بهدرستی و با صحت بالا انجام خواهد شد. قبل از استفادۀ نوكلئوتيدها در واكنش پيروسكوئنسينگ، بايد آلودگيهاي آن از قبيل PPi، حذف شوند. بايد دقت شود كه در اين تكنيك بهجای ATP از dATP-αS استفاده ميشود، زيرا dATP-αs توسط لوسيفراز مصرف نميشود؛ ولي به كمك آنزيم پليمراز ميتواند به رشتۀ الگو متصل شود.
- از آنجا كه آنزيمهاي مورد استفاده در اين تكنيك بهخصوص لوسيفراز حساس به گرما ميباشند، دماي طويلسازي رشتۀ در حال ساخت بايد روي ̊ 28 سلسیوس تنظيم شود. در اين شرايط دماي اتصال پرايمر به الگو و همچنين اتصال پرايمرها به يكديگر افزايش مييابد، به همين دليل در طراحي اين پرايمرها بايد نهايت دقت را بكار گرفت تا به نقاط غیراختصاصی متصل نشود. در غير اين صورت، اين نقاط غیراختصاصی است كه تعيين توالي ميشوند. البته با افزودن پروتئين متصل به DNA تکرشتهای، قسمت عمدۀ پيغامهاي غیراختصاصی و ناخواسته حذف ميشود.
- مشكل عمدۀ اين تكنيك عدم توانايي تعيين توالي دقيق نقاط هموپليمر ميباشد. اگر بيش از 6-5 نوكلئوتيد يكسان پشت سر هم قرار بگيرند، پاسخ نوري غیرخطی شده و تعيين توالي دچار اشكال خواهد شد.
- در هنگام تعيين توالي مناطق هتروزيگوت، ممكن است پيغامهاي خارج از فاز ايجاد شود. با تغيير الگوهاي توزيع نوكلئوتيد، ممكن است اين مشكل مرتفع شده و توالي اين نقاط راحتتر تعيين شود.
- در هر آزمايش بهطور معمول توالي 50-40 نوكلئوتيد تعيين خواهد شد. اين امر يكي از محدوديتهاي اين تكنيك ميباشد.
[1] Pyrosequencing
[2] Indirect bioluminometric assay
[3] Real time
[4] Luminometer
[5] Apyrase
[7] Single stranded DNA binding protein
[8] Rapid
[9] Multiplexing of SNP or mutation detection
[10] DNA pool
[11] Peak
[12]Pyrogram
[13] Allele-specific polymerase chain reaction (AS-PCR)
[14] mismatch
نشانگرهای مولکولی و تكنيكهاي نقشهبرداري ژنومي و انگشتنگاري DNA
پلیمورفیسمهای تکنوکلئوتیدی در درمان و تحقیقات سرطان
برای دانلود پی دی اف بر روی لینک زیر کلیک کنید
ورود / ثبت نام