مروری تازه بر مایکوباکتریوم ها
دكتر رضا ميرنژاد (استاد دانشگاه)
مایکوباکتریوم ها باکتریهای هوادوست و باسیلی شکل هستند که اسپور تولید نمیکنند. با وجود اینکه بهراحتی رنگ نمیگیرند، ولی وقتی رنگ گرفتند در برابر از دست دادن رنگ خود بهوسیله اسید یا الکل مقاومت میکنند، بنابراین باسیلهای مقاوم به اسید (اسیدفست) نامیده میشوند.
کمپلکس مایکوباکتریوم توبرکلوزیس (MTBC) شامل مایکوباکتریوم بوویس سویه باسیل کالمت گرین (BCG)، مایکوباکتریوم کاپرائی، مایکوباکتریوم پینیپدی، مایکوباکتریوم آفریکانوم، مایکوباکتریوم میکروتی و مایکوباکتریوم کانتتی است. مایکوباکتریوم توبرکلوزیس (عامل کخ) عامل یکی از مهمترین عوامل بیماریزای انسانی بنام سل است. مایکوباکتریوم لپره عامل جذام و مایکوباکتریوم آویوم– اینتراسلولار و سایر مایکوباکتریوم های آتیپیک اغلب بیماران مبتلا به ایدز را آلوده میکنند و در سایر بیماران دچار ضعف ایمنی بهعنوان یک بیماریزای فرصتطلب عمل کرده، گاهی نیز در اشخاص دارای سیستم ایمنی طبیعی ایجاد بیماری میکنند.
مایکوباکتریوم بوویس سویه باسیل کالمت گرین (BCG) در بیشتر کشورها غیر از ایالات متحده بهمنظور واکسن بهکار میرود، همچنین بهصورت داخل مثانهای برای تحریک سیستم ایمنی در برابر سرطان مثانه تجویز میشود و در چنین شرایطی بهندرت ممکن است سبب بیماری منتشره شود. مایکوباکتریوم کاپرائی بیشتر بزها را آلوده میکند، اما گاوها و بهندرت انسان را نیز آلوده میکند. مایکوباکتریوم کانتتی نخستین بار در سال 1969 جدا شد و پس از سالها مشخص شد که توانایی ایجاد بیماری در انسان را دارد. میزبان طبیعی این باکتری شیرهای دریایی هستند، اما میتواند سایر حیوانات را آلوده کرده و گاه به انسانها نیز منتقل شود.
مايكوباكتريوم توبركلوزيس (Mycobacterium tuberculosis)
مايكوباكتريوم توبركلوزيس باكتري دورن سلولي اختياري است كه به دليل كمپلكس ديواره سلولي چندلایه و آبگریز به اسيد و قليا مقاوم است. باكتري بهصورت ضعيف گرم مثبت بوده، به شكل باسيلهاي راست يا كمي خميده با اندازه 1 تا 10 میکرومتر طول و 0/2 تا 0/6 میکرومتر عرض ديده میشود. باسیل کخ، هوازي اجباري (در غياب O2 رشد نمیکند)، كاتالاز و پراكسيداز مثبت بوده و رشد بطئي دارد و در pH=7 رشد میکند. احتياجات غذايي مايكوباكتريوم توبركلوزيس ساده بوده، ولي بهعلت تولید اسيدهاي چرب، نيازمند تركيباتي جهت خنثي نمودن آن است. مايكوباكتريوم توبركلوزيس که تنها در انسان بيماريزا است، هيچ اگزوتوكسيني ترشح نکرده و فاقد اندوتوكسين در ديواره سلولي خود است.
شاخص بيماريزايي آن آدنيلات سيكلاز، آرابينوگالاكتان، ليپوآرابينومانان، مایکولیک اسيد، مايكوزيدها (مثل فاكتور طنابي)، سولفاتيدها، توبركلوپروتئين، واكس D، Phop و فیتوسرول دیمایکوسروزات است. سیستم ترشحی ESX نیز که به نام سیستم ترشحی ویژه در بیشتر باکتریهای گرم مثبت حضور دارند، در بیماریزایی اهمیت فراوانی داشته و موتانتهای بدون این سیستم یا زیرواحدهای آن در ماکروفاژهای کشت دادهشده و عفونت در مدلهای حیوانی تضعیف شدهاند. موتانتهائی با حدف دربرگیرنده لوکوس ESX-1 دارای نقص در انتشار سلول به سلول و پروفایل سایتوکینی تغییریافته بوده و نارسائی در مهار ادغام فاگولیزوزوم در کشتهای سلولی را نشان میدهد. این باکتری عامل بيماري سل است که یک بيماري عفوني نكروزدهنده حاد يا مزمن است كه باعث گرفتاري ارگانهاي مختلف بدن خصوصاٌ ريهها ميشود.
ضايعات ایجادشده در این بیماری بهصورت اگزوداتيو و پروداكتيو (گرانولوماتوز)، به علت وجود ارگانيسم و پاسخ ميزبان است. تخمين زده ميشود كه ساليانه حدود 5-4 ميليون مورد سل شديد با خلط مثبت و 5-4 ميليون مورد سل با شدت كمتر و يا كشت منفي در جهان رخ ميدهد كه هرساله حدود 3 ميليون نفر در اثر ابتلا به اين بيماري جان خود را از دست ميدهند. طبق گزارشهای سازمان جهاني بهداشت، هر 4 ثانيه 1 نفر در سطح جهان مبتلا به سل ميشود و هر 10 ثانيه يك نفر جان خود را در اثر ابتلا به اين بيماري از دست ميدهد.
شايان ذكر است كه ميزان بروز اين بيماري در نقاط مختلف كشور ما از توزيع يكساني برخوردار نیست و با توجه به اینکه كشورهاي همسايه شرقی کشورمان جزء آلودهترين كشورهاي جهان بهحساب میآیند، طبيعي است كه بيشترين ميزان بروز بيماري در شهرهای مجاور این کشورها (سيستان و بلوچستان، خراسان جنوبی و گلستان) باشد. در دنیا، کشورهای هند، چین، افریقای جنوبی، اندونزی و پاکستان پنج کشور دارای بالاترین موارد بیماری بودند. امروزه سل مقاوم به چند دارو (MDR-TB) و سل مقاوم به دامنه گستردهای از داروها (XDR-TB)، مشکل اصلی کشورهای آلوده به سل هستند. میزان بروز بیماری سل در افراد سالم از نظر سیستم ایمنی 5 تا 10 درصد است، در مقابل در افرادی که همزمان با مايكوباكتريوم توبركلوزيس و HIV آلوده میشوند ریسک ایجاد بیماری فعال سل حدود 7 تا 10 درصد به ازای هر سال است.
افرادی که ریسک عفونت مايكوباكتريوم توبركلوزيس در آنها بالاست عبارتند از:
افرادی که با مبتلایان به بیماری فعال شناختهشده یا مشکوک به آن در تماس نزدیک هستند،
افرادی که به یک کشور با میزان شیوع بالای سل فعال سفر میکنند،
ساکنان و کارکنان اماکن پرازدحام که مراجعهکنندگان آنها ریسک بالایی از عفونت فعال را دارند (مانند زندانها، پناهگاههای افراد بیخانمان)،
کارکنان مراقبتهای سلامتی که از بیماران با ریسک بالای سل فعال مراقبت میکنند،
افراد با سوءمصرف مواد مخدر و الکل
فاکتورهایی که ریسک پیشرفت بیماری سل فعال را افزایش میدهند شامل موارد ذیل است:
عفونت با HIV
کودکان زیر 5 سال
درمان با مهارکنندههای سیستم ایمنی
عفونت با مايكوباكتريوم توبركلوزيس در دو سال گذشته
سابقه درمان نامناسب سل فعال
سلیکوزیس
دیابت شیرین
نارسائی مزمن کلیه
سرطانهای گوناگون
گاستروکتومی یا جراحی و برداشتن ایلیوم-ژژنوم
کشیدن سیگار و سوءمصرف مواد مخدر و الکل مهمترین منبع عفونت افراد مبتلا به بیماری سل حفرهای (و خلط با اسمیر مثبت) که تشخیص داده نشدهاند
تشخيص آزمايشگاهي:
در بيماري سل بعد از تست پوستي و اشعه x، تنها جدا كردن و تشخيص باسيل كخ تعيينكننده قطعي در تشخيص بيماري است.
نمونهبرداري:
نمونهبرداري صحيح، فاکتور اصلي در كمك به تشخيص سريع و قطعي بيماري است، ازاینرو كاركنان (پزشك، پرستار و …) بايد توجه كافي به اين مسئله داشته باشند. نمونهها شامل خلط، چرك غدد، مايع جنب، مايع صفاق، مايع مفصلي، مايع نخاع، خون، ادرار، شيره معده، ضايعات جلدي، بيوپسي از بافت پریکارد و غشای خارجی قلب، اندومتر، لوله فالوپ و مدفوع، براي بررسي باسيل سل هستند.
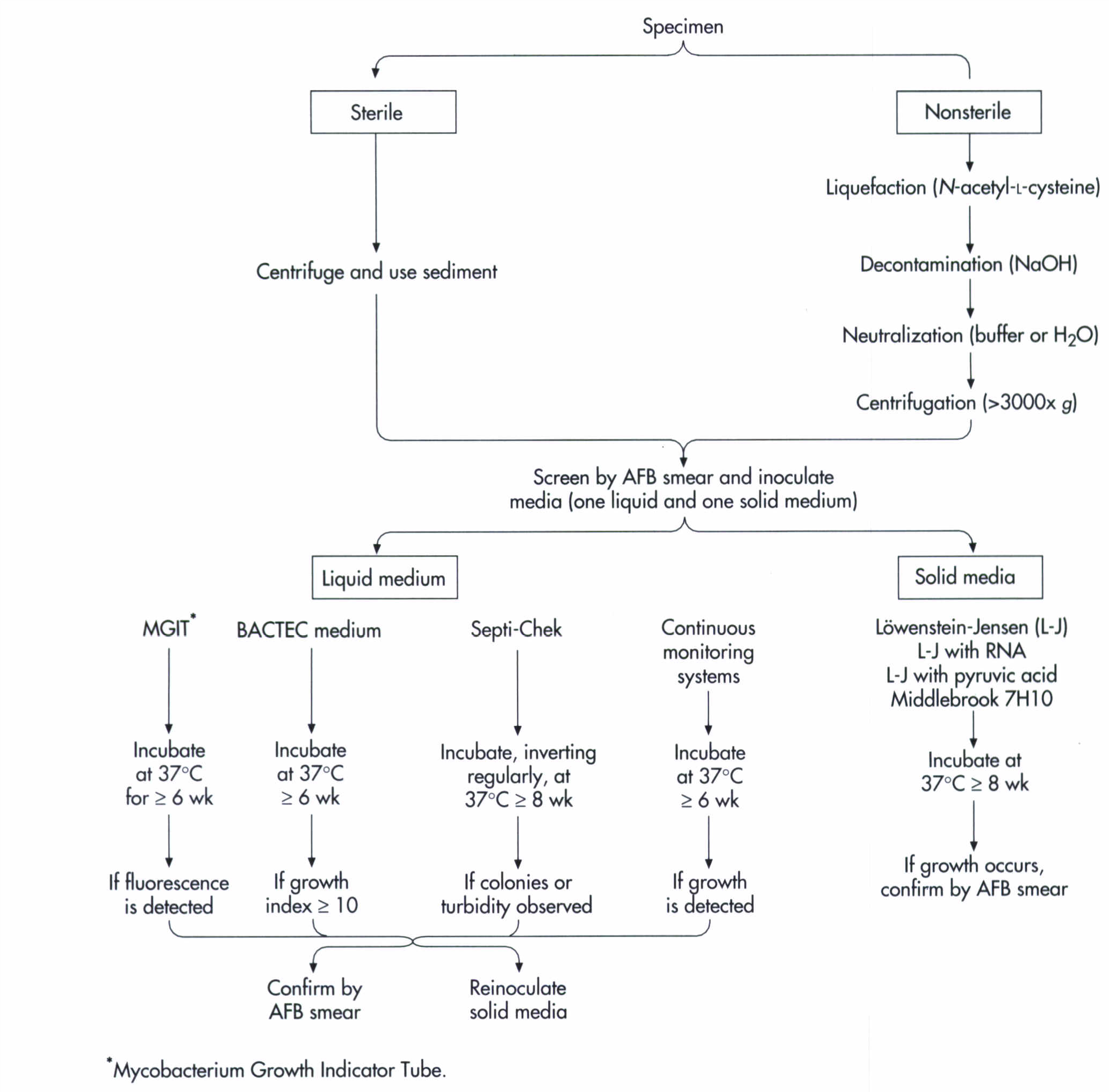
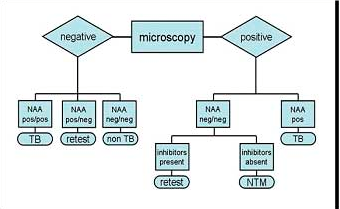
در شکل زیر فلوچارت پروسسینگ نمونهها و تشخیص مایکوباکتریومها نشان داده شده است.
شکل (1): فلوچارت پروسسینگ نمونهها و تشخیص مایکوباکتریوم ها
تهیه نمونه خلط:
نمونه خلط در هنگام صبح و پس از برخاستن از خواب پس از 3-2 بار شستشوی دهان جمعآوري ميشود. بیمار باید در هوای آزاد درحالیکه روبروی او کسی قرار ندارد با سرفههای عمیق درصدد تهیه خلط برآید. در گذشته که خلط 24 ساعته جمعآوري ميگرديد، از آن جهت به تشخيص كمك ميكرد كه تعداد بيشتري باسيل دفعشده مورد آزمايش قرار ميگرفت، ولي در عوض احتمال آلودگي به انواع باكتريها و قارچها افزايش يافته و سبب كند شدن تشخيص و ايجاد خطا ميگردید. ازاینرو بهتر است خلط صبحگاهی را به مدت چند روز متوالي از بيمار جمعآوري و مورد آزمايش قرار دهیم.
نمونه را در ظرف دهانگشاد از جنس پروپیلن یا پلیاتیلن، تميز و استريل جمعآوري نموده و درب آن را بسته به آزمايشگاه ارسال نمایید. باید توجه داشت که حداقل میزان نمونه 5 میلیلیتر باشد. لازم به ذکر است که از افرادی که خلط کمی دارند باید نمونه با شستشوی برونش و لاواژ برونکوآلوئولار (BAL) تهیه گردد. بهتر است نمونهها پس از انتقال به آزمایشگاه، سریع کشت داده شوند یا از آنها اسمیر تهیه شود، ولی وقتی امکان آن وجود ندارد، میتوان نمونه را برای کشت حداقل 3 روز و برای تهیه اسمیر 10 روز نگهداری نمود.
پس از انتقال نمونه خلط به آزمایشگاه بایستی باکتریهای مزاحم را در نمونه با روش هموژنیزاسیون از بین برد و برای افزایش تعداد باسیل سل آن را تغلیظ نمود. در ادامه روشهایی برای نمونههای مختلف توضیح داده میشود.
روش تغلیظ نمونه خلط با ان- استیل- ال سستئین (NALC)- هیدروکسید سدیم (سود):
در مواردی که تعداد باسیلها در خلط کم است و بهوسیله آزمایش مستقیم مشاهده نمیشوند، با استفاده از ان- استیل- ال سستئین (NALC)- هیدروکسید سدیم (سود) 2% نمونه را تغلیظ نمایید. برای این کار:
- حجم مساوی از ان- استیل- ال سستئین (NALC)- هیدروکسید سدیم (سود) 2% و خلط (حدود 10 میلیلیتر از هرکدام) را در شیشه درپیچدار بریزید و درب شیشه را محکم ببندید. توجه داشته باشید محلول ان- استیل- ال سستئین (NALC)- هیدروکسید سدیم (سود) موقع استفاده در همان روز تهیه گردد.
- روی دستگاه همزن به مدت 30-15 ثانیه مخلوط گردد.
- به مدت 15 دقیقه نمونه را در لولههای حاوی گلولههای شیشهای قرار داده و بهشدت تکان دهید و آن را مخلوط نمایید.
- افزودن 20 میلیلیتر از بافر فسفات (pH=8/6) به لولهها و بستن درب لولهها و سروته کردن برای مخلوط شدن نمونهها
- نمونه را به مدت 15 دقیقه با دور 3000 تا 3600 سانتریفوژ کنید. میتوان در دور 2500 به مدت 20 دقیقه سانتریفوژ کرد.
- از رسوب برای تهیه اسمیر جهت رنگآمیزی و تلقیح به محیط کشت استفاده شود.
روش تغلیظ نمونه خلط با سود:
در مواردی که تعداد باسیلها در خلط کم است و بهوسیله آزمایش مستقیم مشاهده نمیشوند، با استفاده از سود 3% نمونه را تغلیظ نمایید. برای این کار:
- حجم مساوی از سود 3% و خلط (حدود 10 میلیلیتر از هر کدام) را در شیشه درپیچدار بریزید و درب شیشه را محکم ببندید.
- به مدت 15 دقیقه نمونه را در لولههای حاوی گلولههای شیشهای قرار داده و بهشدت تکان و آن را مخلوط نمایید.
- نمونه را به مدت 15 دقیقه با دور 3000 سانتریفوژ کنید.
- مایع رویی را دور ریخته و به رسوب باقیمانده آنقدر اسید معرفدار اضافه نمایید تا رنگ آن از قرمز به زرد برگردد، سپس نمونه را کشت دهید.
طرز تهیه اسید معرفدار
1- محلول الف: 0/4گرم فنل را در 100 سیسی سود 3% حل کنید.
2- محلول ب: 85 سیسی اسید کلرئیدریک غلیظ
محلول اسید معرفدار: به 500 سیسی آب مقطر، 20 سیسی از محلول الف و 85 سیسی اسید کلریدریک غلیظ اضافه نمایید سپس حجم را به یک لیتر برسانید.
نکته مهم 1: توصیه میگردد تمام مراحل تغلیظ و هموژنیزه کردن، رنگآمیزی و کشت و سایر کارها روی نمونههای مشکوک به مایکوباکتریوم توبرکلوزیس در زیر هود کلاس II انجام پذیرد.
نکته 2: قبل از اضافه کردن سود، بایستی به نمونه خلط، N-استیل-L- سیستئین (NALC) جهت هضم و مایع کردن نمونه اضافه گردد. لازم به ذکر است که سود سبب از بین رفتن آلودگی باکتریایی و قارچی از نمونه میشود، همچنین میتوان از موادی مانند هیدروکسید سدیم 4-2%، اسيد سولفوريك 4%، اسيد اگزاليك 5%، تريسديم فسفات (TSP=Trisodium phosphate) و زفیران (بنزالکونیوم کلراید) جهت هموژنیزه کردن خلط استفاده کرد.
نکته 3: نمونههایی که از محلهای استریل بدن (مانند CSF) بدست میآیند احتیاج به رفع آلودگی ندارند و میتوانند مستقیماً سانتریفوژ و بررسی شده و کشت داده شوند.
آزمایش شیره معده:
عموماً نمونه شستشوي معده از کسانی که خلط خود را ميبلعند و نميتوانند خلط را دفع كنند (مخصوصاً در كودكان) تهیه میشود.
توجه: بدلیل اینکه باسیل سل سریعاً توسط اسید معده از بین میرود، لذا چنانچه شیره معده بلافاصله مورد آزمایش قرار نمیگیرد، باید نمونه به طریق زیر خنثی شود:
1- افزودن بیکربنات سدیم 10% در حضور فنل رد (0/004) تا معرف شروع به ظهور رنگ قرمز نماید.
2- روش دیگر افزودن 1/5 میلیلیتر فسفات سدیم 40% به حدود 30-20 میلیلیتر شیره معده است. شیره معده را در لوله سانتریفوژ ریخته و با دور 3000 به مدت 30 دقیقه سانتریفوژ نمایید. سپس محلول رویی را دور ریخته و رسوب حاصل را در 5-2 میلیلیتر آب مقطر استریل حل نموده و همحجم آن سود 3% افزوده و بقیه مراحل را مانند تغلیظ خلط انجام دهید.
نکته: حتماً بایستی از آب مقطر استریل استفاده شود و از آب لولهکشی که سبب اختلال در رنگآمیزی و آلودگی محیط کشت میشود، استفاده نگردد.
کشت ادرار از نظر باسیل سل:
ادرار مانند خلط یا شیره معده نیاز به تریتمنت کردن قبلی ندارد و بعد از سانتریفوژ میتوان آن را رنگآمیزی و کشت داد.
برای انجام سانتریفوژ:
- 50 میلیلیتر ادرار را در چند لوله سانتریفوژ به مدت 30 دقیقه با دور 3000 سانتریفوژ نمایید.
- مایع فوقانی را دور ریخته و رسوب را در 5-2 میلیلیتر آب مقطر حل کنید.
- مایع حاصله را با حجم مساوی از سود 3% مخلوط نمایید.
- بقیه مراحل مانند کشت خلط انجام گردد (مخلوط کردن– سانتریفوژ و کشت)
نکته: لازم به ذکر است که برای آزمایش ادرار باید از رسوب ادرار در سه روز متوالی و یا برای جلوگیری از اتلاف وقت از رسوب تهنشینشده ادرار 24 ساعته گسترش تهیه نمود. ممکن است گاهی اوقات باسیل اسمگما (مایکو باکتریوم اسمگماتیس (smegmatis.M) در رسوب ادرار وجود داشته باشد و باعث اشتباه گردد.
روش کشت مدفوع از نظر باسیل سل:
روش اول:
- 5 گرم مدفوع را در 30-20 میلیلیتر آب نمک استریل اشباع یا آب مقطر حل نمایید.
- نیم ساعت در دمای اتاق باقی گذاشته، سپس بهوسیله قاشقک استریل باکتریهای شناور در سطح مایع را جمعآوری و به لوله سانتریفوژ منتقل کنید.
- همحجم آن سود 3% اضافه نمایید و مدت 3 ساعت در 37 درجه سلسیوس قرار دهید.
- سپس آن را با محلول معرفدار خنثی کنید و به مدت 20 دقیقه با دور 3000 سانتریفوژ نمایید.
- از محلول رویی در 5 لوله و از رسوب در 5 لوله بهطور جداگانه کشت دهید.
روش دوم:
- مدفوع را قبلاً در آبگوشت استریل حل کرده و سه برابر حجمش به آن اتر بیافزائید.
- به مدت 20 دقیقه با سرعت 3000 دور سانتریفوژ نمایید.
- با پیپت، اتر رویی را خارج ساخته و از رسوبی که در زیر آن قرار دارد و حاوی باکتری است، برداشت نموده و با آن مانند خلط رفتار نمایید.
کشت نمونههای بیوپسی و خون:
بيوپسي از محلهاي مختلف و بافت استخوان تهیه میشود. در سل منتشره از نمونه خون بيمار ميتوان باكتري را بدست آورد. همچنین در افراد مبتلا به ايدز ممکن است از نمونه خون، مايكوباكتريوم آويوم- انتراسلولار را جدا کرد. چنانچه بافت به طریق استریل تهیه شده باشد، نیازی به رفع آلودگی با سود نیست. ابتدا نمونه را با قیچی استریل به قطعات کوچک تقسیم نموده و در هاون استریل سائیده و بهتدریج به آن سرم فیزیولوژی استریل اضافه کنید. سپس آن را در لوله آزمایش بریزید و بهطور عمودی قرار دهید تا رسوب ایجاد شود و از رسوب و محلول رویی در لولههای مختلف کشت دهید.
کشت مایع نخاعی و سایر مایعات بیولوژیک استریل:
مایع نخاعی را به مدت یک ساعت یا بیشتر در لوله سربسته بهصورت عمودی در یک محل نگه دارید. معمولاً در این حالت یک لخته شبیه تار عنکبوت در مایع ایجاد میشود. لخته را با دقت روی لام گذاشته و لام خشکشده را بهوسیله حرارت فیکس نمایید و پس از رنگآمیزی زیر میکروسکوپ مشاهده کنید. درصورتیکه لخته تشکیل نشده بود، مایع نخاعی را سانتریفوژ کرده (30 دقیقه با دور 3000) و رسوب آن را بهوسیله یک پیپت برداشته مستقیماً روی لام گذاشته و بدون تهیه گسترش، لام را خشک و رنگآمیزی نمایید. از رسوب برای کشت هم میتوانید استفاده کنید. همچنین ميتوان مايع نخاع و ادرار را توسط صافيهاي باكتريولوژيك (μm0/22) تغليظ نمود. پژوهشها نشان داده كه 96% بيماران مسلول را توسط اين روش میتوان تشخيص داد.
تشخیص آزمایشگاهی
رنگآمیزی ذیل- نلسون:
رنگ:
- فوشین بازی 3 گرم
- اتانول 95-90% ml 10
- فنل 5%(ذوب شده) ml 90
فوشین را در الکل حل کنید. فنل را بهآرامی در آب حل نمایید و دو محلول را با هم اضافه نمایید. بهتر است محلول یک شب در 37 درجه سیلسیوس باقیمانده و سپس از کاغذ صافی عبور داده شود.
محلول رنگبر:
- اسید کلرئیدریک ml 3
- اتانول 95-90% ml 97
رنگ زمینه:
- متیلن بلو 3/0 گرم
- آب مقطر ml 100
روش رنگآمیزی ذیل- نلسون
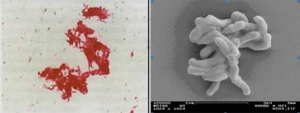
شکل (2): نمای مايكوباكتريوم توبركلوزيس در رنگآمیزی اسیدفست (چپ) و زیر میکروسکوپ الکترونی (سمت راست)
در جدول زیر روش گزارشدهی لام رنگآمیزیشده با دو روش کربول فوشین و فلوروکروم ارائه شده است:
جدول 1: دستورالعمل گزارش اسمیر برای باسیل اسید- فست
| تعداد باسیل اسید- فست با رنگآمیزی فلوروکروم (X450) | تعداد باسیل اسید- فست با رنگآمیزی کربول فوشین (X1000) | گزارش |
| 0 | 0 | عدم مشاهده باسیل اسیدفست |
| 1-2 باسیل در 70 میدان میکروسکوپی (فیلد) در 2 بار رفت و برگشت | 1-2باسیل در 300 میدان میکروسکوپی (فیلد) در 3 بار رفت و برگشت | مشکوک، بررسی مجدد و نتیجه گزارش شود |
| 2-18 باسیل در 50 میدان میکروسکوپی (فیلد) در 1 بار رفت و برگشت | 1-9 باسیل در 100 میدان میکروسکوپی (فیلد) | نادر، +1 |
| 4-36 باسیل در 10 میدان میکروسکوپی (فیلد) | 1-9 باسیل در 10 میدان میکروسکوپی (فیلد) | کمی، +2 |
| 4-36 باسیل در 1 میدان میکروسکوپی (فیلد) | 1-9 باسیل در 1 میدان میکروسکوپی (فیلد) | تعدادی، +3 |
| 36 باسیل یا بیشتر در 1 میدان میکروسکوپی | 9 باسیل یا بیشتر در 1 میدان میکروسکوپی | +4 |
- اگر تعداد غیرواقعی از باسیل در اسمیر مشاهده شد و کشت منفی بود باید موارد زیر مجدداً چک شوند:
- آلودگی مخازن آبی با ارگانیسمهای اسید فست.
- تمیزی اسلایدها.
- آلودگی احتمالی روغن امرسیون
- رنگ زمینه قوی ممکن است وجود باسیل را مخفی نماید، لذا بهتر است بجای متیلنبلو از برلیان گرین که زمینه را شفافتر میکند، استفاده شود.
- ارگانیسمهای اسیدفست عبارتند از: ردوکوکوس، نوکاردیا، لژیونلا میکددی، کیست کریپتوسپوردیوم
- نمیتوان تنها به نتایج اسمیر تکیه کرد و گزارش داد، بلکه علاوه بر نتایج اسمیر نتایج کشت هم بایستی گزارش گردد.
فاکتورهایی که حساسیت نتایج اسمیر را تحت تأثیر قرار میدهند شامل:
- جمعیت بیمار (بیماران با لزیونهای غاری شکل از افراد بدون غار احتمال بیشتری برای خلط اسمیر مثبت دارند).
- نوع نمونه (نمونههای تنفسی با احتمال بیشتری نسبت به سایر نمونهها مثبت میشوند).
- تعداد نمونههای آزمایششده
- تعداد باسیل اسیدفست موجود در نمونه (به ازای هر میلیلیتر از نمونه تعداد 10000-5000 ارگانیسم نیاز است تا نتیجه مثبت شود).
- گونههای حاضر در نمونه (نمونههای حاوی مایکوباکتریوم توبرکلوزیس یا مایکوباکتریوم کانزانسی با احتمال بیشتری از سایر مایکوباکتریوم ها مثبت میشوند).
- مهارت کارشناس
- رنگ مورد استفاده (رنگهای فلورکروم نسبت به رنگهای کربول فوشین حساسیت بالایی داشته و راحتتر قابل مشاهده هستند و توسط متخصصان CDC توصیه شدهاند. همچنین CDC پیشنهاد میکند که برای نمونههای تخصصی، نتایج اسمیرهای رنگآمیزی شده در طی 24 ساعت از نمونهگیری بایستی گزارش شوند؛ به این منظور در هر 7 روز هفته پردازش نمونهها صورت گیرد.
کشت:
بهمنظور بازیابی مناسب مایکوباکتریوم از نمونههای بالینی، تلقیح به هر دو محیط مایع و جامد توصیه شده است؛ چرا که محیط مایع خیلی حساستر از محیط جامد است و شناسایی سریع رشد مایکوباکتریومها را فراهم میکند. مهمترین مزیت کشت روی محیطهای جامد این است که این کار امکان تشخیص مورفولوژی کلنیها و تولید پیگمان را میدهد که به تشخیص بهویژه افتراق کلنیهای مایکوباکتریوم توبرکلوزیس از بعضی مایکوباکتریوم غیرسلیکمک میکند.
معایب محیطهای جامد مرسوم شامل مدت زمان طولانی برای رشد مایکوباکتریومها (کلنیها اغلب بر روی محیط جامد لولهای زودتر از 4-3 هفته مشاهده نمیشود) و حساسیت پایین آنها است. تلقیح به محیط جامد میدلبروک بر روی پلیتها که بعداً در زیر میکروسکوپ بررسی میشوند، اجازه شناسایی سریع کلنیها را میدهد، اما این پروسه بهزحمت فراوان و نیروی انسانی زیادی نیاز دارد، بنابراین در بیشتر آزمایشگاههای بالینی منع استفاده دارد.
برای کشت مایکوباکتریومها از پلیت، لولههای درپیچدار و فلاسکها استفاده میشوند. فلاسکها به دلیل ایمنی بیشتر و فراهم کردن سطح بیشتری برای رشد مایکوباکتریومها به لولههای درپیچدار و پلیتها ترجیح داده میشوند.
محیط لازم برای کشت اولیه مایکوباکتریومها باید شامل یک محیط غیرانتخابی و یک محیط انتخابی باشد؛ محیط انتخابی حاوی آنتیبیوتیکهایی مانند ]مالاشیت گرین g/100ml 0/0025، لینکومایسین (μ g/ml2)، سیکلوهگزامید ( μg/ml360)، نالیدیکسیک اسید ( μg/ml20-30)، کربنی سیلین (μg/ml50)، پلیمیکسین B(U/ml200)، تریمتوپریم (μg/ml20)، آمفوتریسین B (μg/ml10)[، جهت جلوگیری از رشد بیش از حد سایر باکتریها و قارچهای آلودهکننده است. سه فرمول عمومی وجود دارند که میتوانند هم برای محیطهای کشت انتخابی و هم غیرانتخابی مورد استفاده قرار گیرند که شامل محیطهای زیر است:
1- محیط آگار نیمهمصنوعی (مانند میدلبروک 7H10 و 7Hll). از این محیطها بیشتر برای مشاهده شکل کلنیها و آزمایش حساسیت باکتریها استفاده شده و با افزودن برخی آنتیبیوتیکهای فوق انتخابی میشوند.
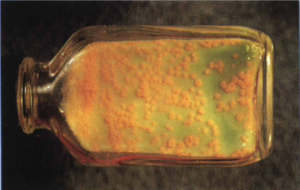
2- محیط تخممرغ سفتشده (مانند لوین اشتاین جانسون). این محیط که بیشتر در کشور ما ایران استفاده میشود، با افزودن آنتیبیوتیک انتخابی میگردد. اگر نمونه کم باشد، 6-3 هفته وقت لازم است تا روی آن کلنی ظاهر شود (شکل 3).
3- محیط آبگوشتی (میدلبروک 7H9 و 7H12). محیطهای انتخابی آبگوشتی حساسترین روش جهت جداسازی است و خیلی سریعتر از سایر محیطها جواب میدهد (شکل 4).
نمونهها را میتوان با لوپ، سواب، پیپت پاستور، نوک سمپلر و غیره کشت داد. پس از محکم کردن درب لوله کشت، باید یک هفته در وضعیت شیبدار قرار گیرد تا با پخش شدن ماده تلقیحشده، رشد یکنواختی حاصل شود. انکوباسیون بایستی در 11-3% CO2 انجام شود. گرچه در آزمایشگاههای ما معمولاً این اصل به علت کمبود انکوباتورهای CO2دار نادیده گرفته میشود، ولی میتوان از جارهای بیهوازی با روشن کردن شمع، محیط CO2 موردنظر را تأمین کرد. هرچند که بدلالیل نامعلوم، مایکوباکتریها در جارهای بیهوازی بهخوبی رشد نمیکنند.
شکل (3): کلنیهای مایکوباکتریوم توبركلوزيس روی محیط لوين اشتاين جانسون بعد از 22 روز انکوباسیون در 35 درجه سانتیگراد در حضور 10% CO2
شکل (4): کلنیهای مایکوباکتریوم توبركلوزيس روی محیط میدل بروک 7H10 بعد از 25 روز انکوباسیون در 35 درجه سانتیگراد در حضور 10% CO2
بررسی لولههای کشت: حداقل هر هفت روز یکبار به مدت هشت هفته باید محیط کشت را از نظر کلنی بهوسیله ذرهبین بررسی نمود.
گزارش رشد
| تعداد کلنی مشاهدهشده | گزارش |
| هیچ کلنی | منفی |
| کمتر از 50 کلنی | ذکر تعداد کلنی |
| 50-100 کلنی | +1 |
| 100-200 کلنی | +2 |
| 200-500 کلنی | +3 |
| بیش از 500 کلنی | +4 |
نکته: در سالهای اخیر بدلیل استعمال نادرست از داروهای ضدسلی، متابولیسم باکتری عامل سل، طوري تغییر یافته است که پس از هموژنیزه کردن خیلی کند رشد میکنند و گاهی هم رشد نمیکنند، به همین جهت توصیه میشود که:
اولاً هرقدر ممکن است زمان هموژنیزاسیون را کوتاه کنید و ثانیاً لولههای کشت را مدت زیادتری در انکوباتور قرار دهید.
كشت و تشخیص سریع:
معمولترين سيستم كشت سريع مایکوباکتریوم توبركلوزيس که اغلب امروزه مورد استفاده قرار میگیرد، سيستم BACTEC است که چندین نوع از این سیستمهای جداسازی اتوماتیک در دسترس است. در یک سیستم BACTEC از ميدلبروك 7H12 حاوي سوبستراي اسيد پالمتيات نشاندار با C14 استفاده میشود. مایکوباکتریوم توبركلوزيس در عرض 10 تا 14 روز در اين سيستم رشد میکند، سپس از روي اين سيستم به محيط BACTEC-NAP براي 7 روز بعد منتقل میشود (مجموعاً 7 تا 21 روز). در اين محيط با استفاده از ρ- نيترو-a- استيل آمينو- b-هيدروكسي پروپيوفنون (NAP) رشد باكتري متوقف میشود.
از طرف ديگر، با DNA پروب، نمونههاي محيط BACTEC كه بهعنوان مایکوباکتریوم توبركلوزيس شناخته شدهاند را بهسرعت میتوان در 2 تا 8 ساعت بعد از جداسازي تشخيص داد (جمعاً 10 تا 15 روز). استفاده از BACTEC، زمان لازم براي شناسايي مایکوباکتریوم توبركلوزيس را از 3 تا 5 هفتهاي كه براي روشهاي معمول (مثل محيط لوين اشتاين جانسون) لازم است، كاهش میدهد، همچنین از این سیستمها میتوان برای تعیین حساسیت سریع مایکوباکتریومها اقدام کرد. معایب این سیستمها گرانقیمت بودن آنها، مشاهده نکردن کلنیها روی آن، آلودگی با سایر مایکوباکتریومها، مشکلات در دفع مواد رادیواکتیو و استفاده زیاد از سوزن است.
در آزمایشگاههایی که برای کشت مایکوباکتریومها تعداد درخواستهای کمی دارند، لولههای MGIT که بهصورت دستی خوانش میشوند و میتوانند برای کشت همه انواع نمونهها بهاستثنای خون و ادرار مورد استفاده قرار گیرند، ممکن است مقرون بهصرفهترین انتخاب برای آزمایش باشند. این لولهها حاوی محیط مایع 7H9 اصلاحشده و یک اندیکاتور فلوئورسنت تعبیهشده در یک سیلیکون در ته لوله هستند. لولهها با نمونهها تلقیح شده و یک مخلوط آنتیبیوتیکی برای مهار رشد باکتریهای آلودهکننده و یک غنیکننده رشد مایکوباکتریومی نیز افزوده میشود و در لولهها بسته شده و در دمای 37 درجه سلسیوس به مدت 6 هفته انکوبه میشوند.
برای ردیابی رشد مایکوباکتریومها، لولهها بهصورت دستی بر روی ترانسلومیناتور با طولموج 365 نانومتر فرابنفش یا در مقابل لامپ WOOD قرار داده میشوند. ظاهر شدن فلوئورسنس قوی در سنسور (یک رنگ نارنجی روشن در ته لوله و هلال سطحی) نشاندهنده رشد باکتری است. برای آزمایشگاههایی با تعداد بالای درخواستها، دستگاه تمام اتوماتیک BACTEC960 که بهصورت پیوسته لولهها را از نظر فلوئورسانس و سیگنال برای آنهایی که مثبت شدهاند چک میکند، مناسب است.
علاوه برBACTEC و MGIT، دو سیستم دیگر اتوماتیک برای رشد و شناسایی مایکوباکتریومها در نمونهها بهصورت تجاری در دسترس هستند؛ سیستم MB/BACT و VersaTrek. در هرکدام از سیستمها بطریها با نمونهها، یک مخلوط آنتیبیوتیکی و یک مکمل رشد تلقیح میشوند. سیستم MB/BACT یک نوع بطری برای نمونه خون و یک نوع بطری دیگر برای سایر نمونهها دارد.
در مرحله بعد بطریها در دستگاه مربوطه انکوبه میشوند؛ جایی که بطریها از نظر تغییرات در تولید و مصرف گازهای مختلف که نشاندهنده رشد مایکوباکتریومها یا سایر ارگانیسمها است، کنترل میشوند. بهطور کلی این سیستمها بهطور قابلمقایسهای با BACTEC960 عمل میکنند و مزیتهای تمام اتوماتیک بودن را دارا هستند؛ بنابراین زحمت کمتر و نیروی انسانی کمتری نیاز دارند و غیررادیومتریک نیز هستند.
جداسازی و شناسایی مایکوباکتریوم ها بهوسیله روشهای مولکولی، سرولوژی و شناسایی آنتیژن:
چهار کاربرد مهم برای استفاده تکنیکهای مولکولی جهت شناسایی مایکوباکتریومها در آزمایشگاههای کلینیکی وجود دارد:
1- تأیید کشت با استفاده از نشانگرهای DNA
2- شناسایی مایکوباکتریها از طریق سکانس کردن DNA
3- جداسازی مستقیم مایکوباکتریوم توبركلوزيس از خلط و سایر نمونههای کلینیکی
4- تایپینگ گونههای مایکوباکتریها
امروزه روش PCR، در تشخیص سریع و مستقیم مایکوباکتریوم توبرکلوزیس در نمونههای بالینی کمک بسیاری میکند. حساسیت کلی آن 55 تا 90% و میزان ویژگی آن حدود 99% است. بیشترین میزان حساسیت هنگامی است که این آزمون برای نمونههایی بکار رود که گسترههای حاصل از آنها حاوی باسیلهای اسید– فست بودهاند. استفاده از آزمون PCR برای این امر تأیید شده است. در شکل (5) توصیه CDC جهت نحوه گزارشدهی تستهای مولکولی ارائه شده است.
سریعترین روش شناسایی سل ریوی استفاده از تست تکثیر اسید نوکلئیک[1] برای ردیابی مستقیم مایکوباکتریوم توبرکلوزیس در نمونههای بالینی است. امروزه سه نوع تست NAAT برای بررسی نمونههای تنفسی در جهان در دسترس است:
- تست مستقیم مایکوباکتریوم توبرکلوزیس تکثیرشده[2]،
- تست امپلیکور مایکوباکتریوم توبرکلوزیس[3]
- تست Xpert MTB/RIF assay که از سال 2012 در امریکا در دسترس قرار گرفت.
سیستم BDProbtect (BD Biosciences) تنها در ایالات متحده در دسترس است.
روشهای XPERT و AMPLICOR توسط سازمان غذا و دارو تنها برای آزمایش نمونههای تنفسی که از نظر اسمیر باسیل اسیدفست مثبت هستند، تأیید شدهاند.
تست Geneprobe برای آزمایش نمونههای تنفسی اسمیر مثبت و اسمیر منفی مورد تأیید قرا گرفته است.
با مطالعات انجامگرفته، حدس بر این است که تستهای تکثیر اسید نوکلئیک ممکن است در بیماران با اسمیر باسیل اسیدفست منفی که ریسک بالایی برای ابتلا به سل را دارند (مانند افراد آلوده به HIV، افراد زندانی در اماکن تأدیبی) مفید باشد که اجازه تشخیص خیلی سریع و شروع زودتر درمان را میدهد. معمولاً حساسیت تستهای تکثیر اسید نوکلئیک برای نمونههای غیرتنفسی کمی پائینتر است، اما ممکن است برای تشخیص بعضی از موارد خارج ریوی بهخصوص زمانی که اسمیر باسیل اسیدفست مثبت است، مفید واقع شود.
روش+ Xpert MTB/RIF assay به جهت شناسایی ریفامپین دارای مزیت بیشتری است، ولی از نظر فنی و تکنیکی نسبت به سایر تستهای تکثیر اسید نوکلئیک خواهان کمتری دارد. این روش دو جزء دارد:
1- یک کاتریج پلاستیکی حاوی بافرهای مایع پردازش نمونه و PCR و معرفهای لئوفیلیزه Real Time PCR
2- دستگاه GeneXpert که پردازش نمونه و Real Time PCR را در یک مرحله بدون دخالت دست انجام میدهد.
این روش بهطور همزمان مایکوباکتریوم توبرکلوزیس و مقاومت به ریفامپین را بهوسیله تکثیر قطعه 81-bp از ژن ropB مایکوباکتریوم توبرکلوزیس و متعاقباً اتصال پروب برای ردیابی جهشهای مرتبط با مقاومت به ریفامپین شناسایی میکند. انواع نمونههای تأییدشده برای این منظور نمونه خلط تیمارنشده و رسوب خلط تغلیظشده هستند. کل زمان کار با دست به ازای هر نمونه کمتر از 5 دقیقه است و نتایج در کمتر از 2 ساعت در دسترس قرار میگیرد. حساسیت این روش نسبت به کشت برای شناسایی مایکوباکتریوم توبرکلوزیس در نمونههای خلط برای موارد اسمیر مثبت بین 98 تا 100 درصد و برای اسمیرمنفی 69 تا 72 درصد است، اختصاصیت تکنیک 100 درصد است.
پروبهای DNA کمیلومینانس که اجازه شناسایی تعداد کمی از گونهها یا کمپلکسی از گونههای مایکوباکتریوم را در عرض 1 تا 2 ساعت بعد از رشد بر روی محیطهای جامد و مایع کافی میدهند، موجود است. پروبهای تجاری اختصاصی مایکوباکتریوم توبرکلوزیس، مایکوباکتریوم اویوم، مایکوباکتریوم انتراسلولار، مایکوباکتریوم گوردونه و مایکوباکتریوم کانزانسی در دسترس است. تکنیک به حداقل تجهیزات مانند لومینومتر، سونیکاتور و بلوک حرارتی نیاز دارد.
معایب این روش این است که در تشخیص بعضی از ایزولههای مایکوباکتریوم اویوم بهخصوص زمانی که آزمایش رشد از روی یک محیط مایع باشد ناتوان است. نتایج مثبت کاذب با پروب مایکوباکتریوم آویوم زمانی است که نمونههای همحجم aliquots از کشت مایع انجام میشود و بهندرت نتایج مثبت کاذب با پروب مایکوباکتریوم توبرکلوزیس رخ میدهد.
یک تکنیک نسبتاً تازه معرفیشده تحت عنوان Matrix-assisted laser desorption ionization-time of flight mass spectrometry است که شناسایی سریع رشد بر روی محیط جامد را فراهم میکند. این روش که توسط FDA تأیید نشده است، نیاز به دستگاه گرانی دارد، اما هزینههای معرفهای آن برای شناسایی خیلی پائین است، علاوه بر این، این دستگاه میتواند برای شناسایی باکتریها و قارچها در کنار مایکوباکتریومها مورد استفاده قرار گیرد و از نظر تکنیکی برای استفاده آسان است. کروماتوگرافی مایع با کارایی بالا اجازه شناسایی کلنیهای مایکوباکتریومها بر روی محیط جامد یا رشد در محیط مایع را در طی 4-2 ساعت میدهد.
این تکنیک در خدمات بهداشت عمومی و بعضی آزمایشگاههای مرجع برای سالهای زیادی استفاده شده است، اما اخیراً به دلیل در دسترس بودن سایر تکنیکهای خیلی دقیق و کمتر وقتگیر مثل هیبریداسیون معکوس (Line-probe)، محبوبیت خود را از دست داده است. آزمایش Line-probe با استفاده از کلنیهای یک محیط جامد یا مایع از کشت مایع مثبت بهخوبی انجام میشود، اگرچه آزمایش مستقیم نمونههای اسمیر باسیل اسیدفست مثبت امکانپذیر است. در آزمایش line-probe سکانس هدف در ابتدا توسط پرایمرهای بیوتینه شده با انجام PCR تکثیر میشوند، سپس محصول تکثیرشده بر روی یک نوار نیتروسلولز که چندین پروب بر روی آن فیکس شدهاند، بهکار گرفته میشود. خطها در محل هیبریداسیون محصول– پروب شکل میگیرند و الگوی ایجادشده با یک کلید مقایسه شده و اجازه تفسیر نتایج و شناسایی ایزوله را میدهد.
دقیقترین روش شناسایی، تعیین توالی است که اغلب نواحی متغیر ژن ریبوزومی 16SrRNA استفاده میشود. روشهای تایپینگ مولکولی مثل اسپولیگوتایپینگ برای شناسایی اعضای مایکوباکتریوم توبرکلوزیس در سطح گونه مفید هستند. این تکنیکها که به لحاظ تکنیکی پیچیده هستند و نیاز به تجهیزات گرانقیمت دارند، اغلب در زمینه تحقیقات و آزمایشگاههای مرجع بزرگ انجام میشوند.
بررسی ایمنی با استفاده از آنزیم را میتوان برای تشخیص آنتیژنهای مایکوباکتریایی بکار برد، اما حساسیت و میزان ویژگی آن کمتر از سایر روشها است. همین مشکل در استفاده از روش EIA برای تشخیص آنتیبادیهای ضدآنتیژنهای مایکوباکتریوم توبرکلوزیس نیز وجود دارد. هیچکدام از این روشها برای استفادههای تشخیصی عادی کافی نیستند.
شکل (5): توصیههای CDC جهت گزارشدهی تستهای مولکولی (NAA= آمیلی فیکاسیون اسیدنوکلئیک)
تعیین هویت مایکوباکتریوم ها:
از نظر پزشکی، تشخیص و جدا کردن مایکوباکتریوم توبرکلوزیس از سایر گونههای مایکوباکتریومها دارای اهمیت است، بدین منظور بعد از کشت باید باکتری جداشده توسط تستهای بیوشیمیایی زیر هود کلاس II توسط پرسنل ورزیده تعیین هویت شوند. هرچند که تعیین هویت کردن در توان آزمایشگاههای عمومی نیست و امروزه از روشهای مولکولی مانند PCR بیشتر برای تأیید کشت استفاده میکنند، ولی به کمک چند روش ساده زیر تا حدودی میتوان در آزمایشگاههایی که امکان تشخیص مولکولی وجود ندارد به شناخت مایکوباکتریومها نزدیکتر شد.
1- منشأ نمونه: برخی از مایکوباکتریومها مانند مایکوباکتریوم مارینوم، مایکوباکتریوم اولسرانس و مایکوباکتریوم پاراتوبرکلوزیس هرگز از خلط جدا نمیشوند. مایکوباکتریوم مارینوم و مایکوباکتریوم اولسرانس از ضایعات جلدی و مایکوباکتریوم پاراتوبرکلوزیس از مدفوع افراد دچار بیماری کرون جدا میشوند.
2- مورفولوژی میکروسکوپی: مایکوباکتریوم ها بین 5-0/8 میکرومتر طول دارند، لذا به اشکال کوکوباسیل تا باسیلهای کشیده مشاهده میشوند. باسیل سل انسانی معمولاً دراز، باریک و خمیده است، یکنواخت رنگ نمیگیرد و دانهدار بهنظر میرسد که بهعلت وجود پلیفسفاتها و واکوئل در داخل آنها است. مشاهده پلیمورفیسم و گاهی انشعاب ممکن است مربوط به مایکوباکتریومهای گروه سه رانیون (غیرکروموژن) باشد.
3- سرعت رشد: مایکوباکتریومهای سریعالرشد (Rapid grower) که کلنیهای آن در کمتر از 7 روز ظاهر میشوند، معمولاً ساپروفیت محسوب میشوند. یک نوع آن یعنی مایکوباکتریوم اسمگماتیس که در مرد و زن وجود دارد ممکن است نمونههای ادرار و مدفوع را آلوده ساخته و سبب اشتباه در تشخیص سل گوارشی یا ادراری گردد. مایکوباکتریومهای با رشد آهسته (Slow grower) که ظهور کلنی آنها بیش از یک هفته طول میکشد، شامل گونههای مختلفی از جمله مایکوباکتریوم توبرکلوزیس و مایکوباکتریوم بویس هستند. در هر مرحلهای که کلنی ظاهر شد آن را با رنگآمیزی اسیدفست بررسی کنید.
4- حرارت مناسب: بیشتر مایکوباکتریومها رشد مناسب خود را در دمای 37-35 درجه سلسیوس دارند، ولی برخی در شرایط دیگری بهتر رشد میکنند و یا رشد آنها فقط در آن شرایط ایجاد میشود، مانند مایکوباکتریوم مارینوم که در 33-32 درجه سلسیوس و مایکوباکتریوم آویوم– اینتراسلولار در 41 درجه سلسیوس و مایکوباکتریوم گزنوپی در 45 درجه سلسیوس رشد میکنند.
5- شکل کلنی: بهویژه در محیط میدل بروک و به کمک ذرهبین، دقت در شکل کلنی میتواند راهنمایی خوبی جهت تشخیص باشد که نیاز به مهارت و تجربه بالا دارد.
6- تولید پیگمانت: مایکوباکتریومها از نظر تولید پیگمان به سه دسته تقسیم میشوند:
(a) فوتوکروموژن (Photochromogens): کلنی این دسته اگر در تاریکی رشد کرده باشد سفید یا کرم رنگ (بدون رنگدانه) بوده و پس از اینکه کشت آن به مدت یک ساعت در روشنایی قرار گرفت پیگمان زردرنگی بهوجود میآورند که بتدریج تیرهتر شده و قرمز آجری رنگ میگردند؛ مانند مایکوباکتریوم کانزاسی (بهندرت بهصورت نانکروموژن یا اسکوتوکروموژن نیز دیده میشود) و مایکوباکتریوم مارینوم.
(b) اسکوتوکروموژن (Scotochromogens): در نور یا تاریکی تولید کلنی زرد یا نارنجی میکنند، مانند مایکوباکتریوم اسکروفولاسئوم و مایکوباکتریوم گزنوپی
(c) نانکروموژن (Nonchromogens): چه در نور و چه در تاریکی تولید پیگمان نمیکنند، مانند مایکوباکتریوم آویوم- اینتراسلولار (بعضی از ایزولهها بهطور خفیف پس از انکوباسیون طولانی کلنیهای پیگماندار تولید میکنند که سبب اشتباه احتمالی در طبقهبندی بهعنوان اسکوتوکروموژن میشود).
لازم به ذکر است که مایکوباکتریوم توبرکلوزیس، بویس و اولسرانس جزء هیچکدام از این گروهها قرار نمیگیرند. مایکوباکتریوم زولگانی در 37 درجه سلسیوس اسکوتوکروموژن و در 25 درجه فتوکروموژن است. باید توجه داشت که کشت مایکوباکتریومها در محیط مایع باعث میگردد که اندازهگیری سرعت رشد برای طبقهبندی رانیون کاربرد نداشته باشد.
جدول (2): طبقهبندی مایکوباکتریوم ها
آزمایش تولید پیگمان:
اگر باکتری کندرشد باشد و پیگمان تولید نکند و یا پیگمان تولیدشده مشکوک به تأثیر از نور باشد، باید این آزمایش انجام شود:
کشت شیبدار جوان را که حدود 6-5 روز بیشتر از ظهور کلنیهای آن نگذشته باشد به نحوی با کاغذ آلومینیومی بپوشانید که نیمی از کلنیها پوشیده و نیمه دیگر معلوم باشد، سپس آن را به مدت یک ساعت در معرض نور یک لامپ 60-30 وات از فاصله 25 سانتیمتری قرار دهید. در این هنگام باید درب لوله شل باشد تا تبادل هوا انجام شود. بعد از آن لولهها را به انکوباتور تاریک منتقل کرده روز بعد کلنیهای نوردیده را با کلنیهای نورندیده مقایسه نمایید.
سؤال: چرا باید نسبت به تعیین هویت باکتریهای اسیدفست اقدام کرد؟
جواب:
1- به خاطر اطمینان از اینکه باکتری مذکور پاتوژن بوده و ساپروفیت نیست.
2- چون معمولاً مایکوباکتریومهای آتیپیک که موجب سل ریوی و یا ادم ریوی میشوند، مقاومت بیشتری به داروها دارند، لذا گزارش آنها، پزشک را بیشتر به فکر تعیین حساسیت دارویی میکروب میاندازد.
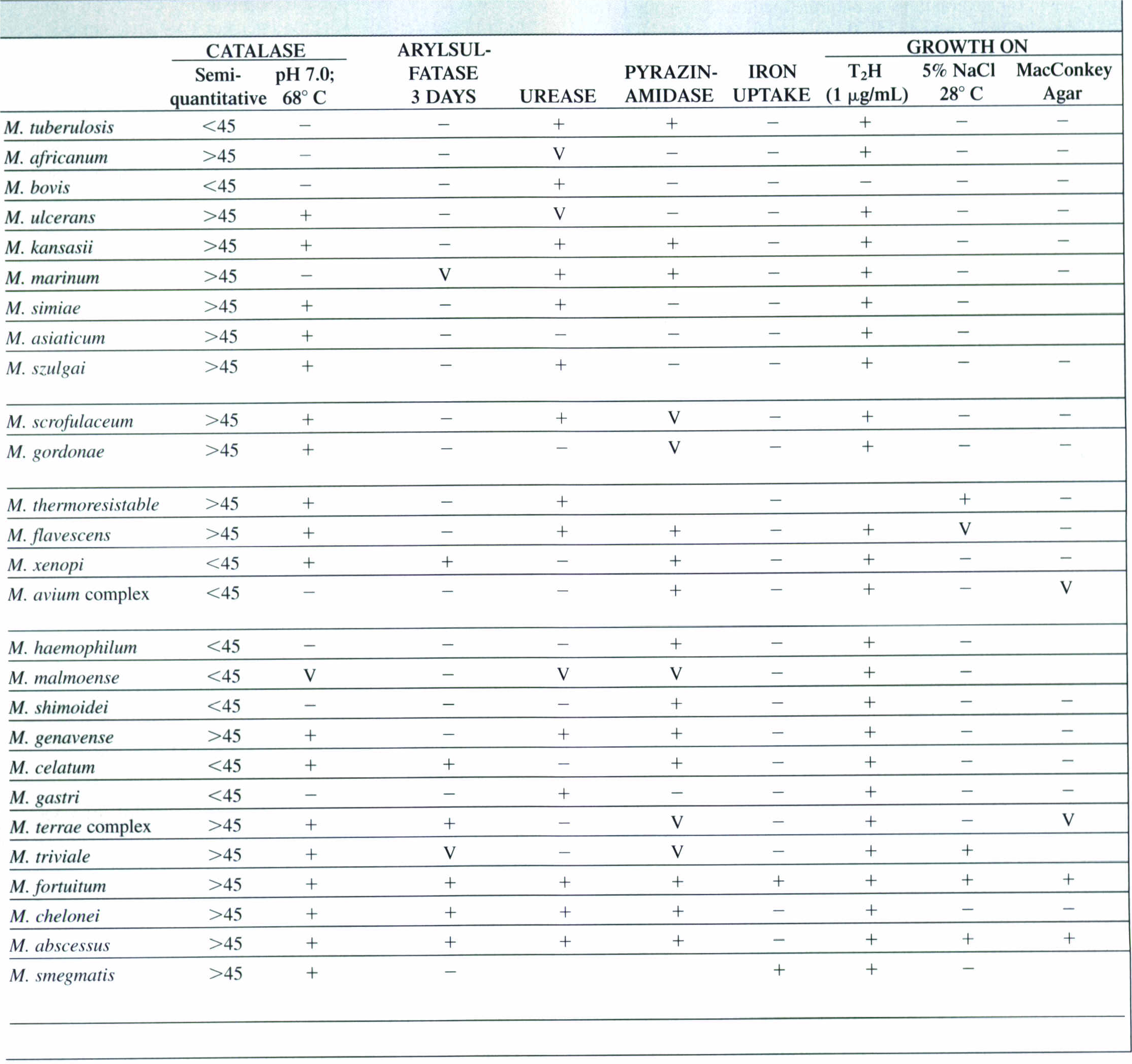
7- واکنشهای بیوشیمیایی: در بین واکنشهای بیوشیمیایی که برای شناسایی و تعیین هویت مایکوباکتریومها بهخصوص مایکوباکتریوم توبرکلوزیس بکار میرود، میتوان به تست نوترال رد، تست نیاسین، تست کاتالاز، تشکیل طناب بعد از رشد روی محیط کشت در زیر لام، حساسیت به T2H (تیوفن)، تولید اورهآز، تست آریل سولفاتاز و رشد روی مکانکی اشاره کرد. در جدول زیر برخی ویژگیهای مختلف گونههای مایکوباکتریوم نشان داده شده است. مایکوباکتریوم توبرکلوزیس از مایکوباکتریـــــــــــوم بویس با اضافه کردن ماده اسید تیوفن (μ g/ml10) به محیط لوین اشتاین جانسون قابل تفکیک است. باسیل سل مقاوم و باسیل گاوی حساس است. مایکوباکتریومهای آتیپیک نیز مقاوم هستند.
جدول (3): ویژگیهای مختلف گونههای مایکوباکتریوم
8- نشانگرهای مولکولی: نشانگرهای مولکولی روشی سریع، حساس و اختصاصی برای تعیین هویت مایکوباکتریوم ها را فراهم میآورند. نشانگرها میتوانند برای مایکوباکتریومهای رشدیافته در محیطهای جامد یا آبگوشتی بهکار روند. نشانگرهای DNA اختصاصی برای توالی rRNA ارگانیسم تحت آزمایش، در روش هیبریداسیون در هر سلول مایکوباکتریایی مورد استفاده قرار میگیرند. در هر سلول مایکوباکتریایی نزدیک به 10000 کپی از rRNA وجود دارد که یک سیستم تکثیر طبیعی را ایجاد کردهاند و در تشخیص کمککننده هستند. هیبریدهای دو رشتهای از نشانگرهای تکرشتهای هیبریدنشده جدا میشوند. نشانگرهای DNA به مواد شیمیایی متصل هستند که در هیبریدهای فعالشده، به کمک درخشش شیمیایی خود شناسایی میشوند.
از نشانگر (پروب) برای شناســــــــــــــــــایی مجموعة مایکوباکتریوم توبرکلوزیس (شـــــــــــــامل M.africanum , M.bovis و M.tuberculosis) مجموعة M.avium (شامل M.intracellulare , M. avium و مایکوباکتریــــومهای بسیـــــــــــــــــــــار شبیه به آن)، M. gordonae ,M. kansasii استفاده میشود. استفاده از این نشانگرها، زمان تشخیص مایکوباکتریومهای مهم از نظر بالینی را از چندین هفته به یک روز تقلیل میدهد.
روش کروماتوگرافی مایع با عملکرد زیاد (HPLC) برای شناسایی مایکوباکتریومها مورد استفاده قرار میگیرد که بر اساس مقدار اسیدمیکولیک (که از گونهای به گونه دیگر متغیر است) استوار است. روش HPLC برای شناسایی مایکوباکتریومها در آزمایشگاههای مرجع در دسترس است.
آنتیبیوگرام:
به علت شیوع بالای ظهور نژادهای چند مقاومتی، باید تمام نمونههای مایکوباکتریم توبرکلوزیس مشکوک را حتماً از نظر حساسیت آنتیبیوتیکی بررسی کرد تا به یافتن درمان انتخابی کمک شود. برای ایزوله اولیه مایکوباکتریوم توبرکلوزیس از هر بیمار باید نسبت بهتمامی داروهای خط اول تست شود. درصورتیکه پس از گذشت 3 ماه از درمان مناسب همچنان کشت نمونهها مثبت بوده یا زودتر از این زمان اگر شواهد بالینی از شکست درمان موجود باشد، تست حساسیت باید تکرار شود. تست ممکن است بر روی کلنیهای ایزولهشده یا یک کشت مایع مثبت (تست غیرمستقیم) و یا بر روی نمونههای خلطی که اسمیر باسیل اسیدفست هستند (تست مستقیم) انجام شود.
در جدول 4 داروهای توصیهشده برای آزمایش حساسیت دارویی مایکوباکتریوم ها ارائه شده است. زمانی که یک ایزوله بهتنهائی به ریفامپین یا هرکدام از دو داروی خط اول بهطور همزمان مقاوم باشد، حساسیت به خط دوم داروها بایستی چک شود. ایزولههایی که تنها به غلطتهای بحرانی از ایزونیازید مقاوم باشند نیز باید نسبت به داروهای خط دوم تعیین حساسیت شوند، اگر رژیمدرمانی برنامهریزیشده شامل یک فلوروکینولون باشد. از تکنیک “محیط آبگوشتی رادیومتریک استاندارد شده” (BACTEC 460، BACTEC 12B) میتوان برای سنجش حساسیت نسبت به داروهای خط مقدم درمان استفاده کرد.
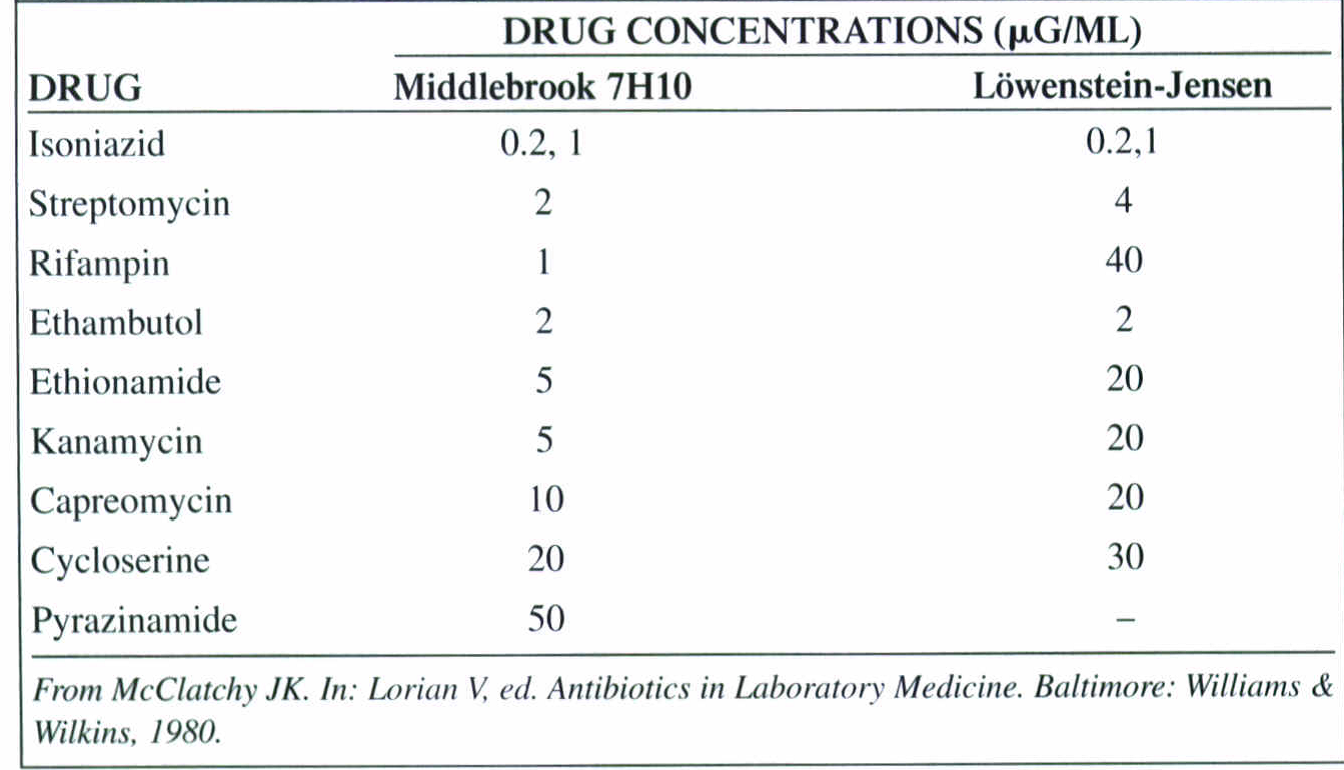
تکنیک متداول، پیچیده و دشوارتر متکی به agar dilution، معمولاً در آزمایشگاههای مرجع بهکار میرود. بهوسیلة این روش داروهای خط اول و دوم درمان را میتوان آزمایش کرد. در جدول 5 غلظتهای دارویی مورد استفاده جهت سنجش حساسیت با محیط کشتهای متفاوت نشان داده شده است.
جدول 4: داروهای خط اول و دوم جهت تعیین حساسیت دارویی مایکوباکتریومها
| گونههای مایکوباکتریوم | بروز | داروها |
| کمپلکس مایکوباکتریوم توبرکلوزیس | اولیه | ایزونیازید، ریفامپین، اتامبوتول، پیرازینامید |
| ثانویه | کاپرومایسین، اتیونامید، اتامبوتول (غلظتهای بالا)، آمیکاسین، کانامایسین، لووفلوکساسین، افلوکساسین، موکسی فلوکساسین، ایزونیازید (غلظتهای بالا)، پاراآمینوسالیسیک اسید، ریفابوتین، استرپتومایسین | |
| کمپلکس مایکوباکتریوم آویوم | اولیه | کلاریترومایسین |
| ثانویه | موکسی فلوکساسین، لینرولاید | |
| مایکوباکتریوم کانزانسی (باسیل زرد) | اولیه | ریفامپین |
| ثانویه | آمیکاسین، سیپروفلوکساسین، کلاریترومایسین، اتامبوتول، ایزونیازید، موکسی فلوکساسین، ریفابوتین، ریفامپین، تریمتوپریم، سولفومتوکسازول | |
| مایکوباکتریوم مارینوم | اولیه | آمیکاسین، سیپروفلوکساسین، کلاریترومایسین، داکسی سیکلین، مینوسیکلین، موکسی فلوکساسین، ریفابوتین، ریفامپین، تریمتوپریم، سولفومتوکسازول |
| مایکوباکتریومهای سریعالرشد | اولیه | آمیکاسین، سفوکسیتین، سیپروفلوکساسین، کلاریترومایسین، داکسیسیکلین، یا مینوسیکلین، ایمیپنم، لینزولاید، مروپنم، موکسی فلوکساسین، توبرامایسین، تریمتوپریم، سولفومتوکسازول |
جدول 5: غلظتهای دارویی مورد استفاده جهت سنجش حساسیت با محیط کشتهای متفاوت
با توجه به اهمیت شناسایی سریع سل مقاوم به دارو برای آزمایش کشتهای مایع یا جامد یا نمونههای اسمیر مثبت بهمنظور ردیابی جهشهای مرتبط با مقاومت مورد استفاده قرار گیرند، روشهای مولکولی توسعه یافته است. استفاده از روشهای مولکولی برای شناسایی مستقیم مقاومت دارویی در نمونههای اسمیر مثبت ممکن است در بیمارانی که سابقه درمان قبلی سل را دارند، آنهایی که از کشور با میزان بالای مقاومت دارویی هستند و وارد کشورمان شدهاند و یا کسانی که نتوانند پاسخ مناسبی به درمان استاندارد داشته باشند، در نظر گرفته شود.
روشهای مولکولی مبتنی بر پروب حضور یا عدم جهشها را ردیابی میکنند. روشهای بر پایه تعیین توالی (مثل روش تعیین توالی سانجر و پیروسکانسینگ) توالیهایی از هر دو ایزوله وحشی و سویه موتانت را فراهم میکنند. نواحی ژنتیکی رایجی که آزمایش میشوند شامل نواحی مشخص مقاومت دارویی از ژنهای embB، gyrA، پروموتورهای inhA و eis، نواحیی از KatG و rrs با جهشهای شناختهشده و orf کامل ژنهای tlyA و pncA هستند. باید به این نکته توجه داشت که بدلیل اینکه مقاومت دارویی میتواند توسط جهشهای دیگری غیر از آنهایی که با این روشها شناسایی میشوند، ایجاد شود؛ بنابراین عدم شناسایی یک جهش الزاماً به معنی حساسیت فنوتیپی نیست.
همچنین به دلیل اینکه حضور جهشها همیشه با مقاومت دارویی مرتبط نیستند، بنابراین شناسایی یک جهش توسط یک روش بر پایه پروب ممکن است در همه موارد مساوی با مقاومت فنوتیپی نباشد، علاوه بر این، از آنجایی که تمامی مکانیسمهای مقاومت شناختهشده نیستند، ناتوانی در شناسایی یک جهش بهروشنی وجود مقاومت را رد نمیکند، ازاینرو اگرچه روشهای مولکولی اطلاعات مفیدی را بهسرعت در اختیار ما قرار میدهند، اما بهعنوان روش کمکی نه جایگزینی برای تست حساسیت دارویی فنوتیپی هستند.
تعیین حساسیت برای مایکوباکتریوم های غیرسلی بایستی بر روی ایزولههای با اهمیت بالینی انجام شود. با این حال برای بعضی گونهها ارتباط کمی میان نتایج تست حساسیت برای بعضی داروها و پاسخ بالینی به درمان یافت شده است. این حالت بهخصوص در مورد کمپلکس مایکوباکتریوم اویوم و نتایج برای اتامبوتول و ریفاماپیسین مناسب است. روش توصیهشده توسط CLSI جهت تست حساسیت مایکوباکتریوم های غیرسلی کندرشد و سریعالرشد، روش میکروبراث دایلوشن است.
ایمنی:
1- پرسنل: باید تعداد آنها کافی و آموزش لازم را دیده باشند، چرا که بیتوجهی پرسنل معمولاً موجب آلودگی میگردد. قبل از شروع به کار در بخش سل، تست PPD را انجام داده و در صورت منفی بودن واکسن BCG تلقیح گردد. در فواصل یکساله از آنها رادیوگرافی سینه بهعمل آید.
2- ساختمان آزمایشگاه: قابلیت شستشوی کف (کفپوشهای پلاستیکی بهتر است) و نبودن خلل و فرج در دیوارها، وجود کافی روشنایی و نورگیر بودن و جدا بودن محل آزمایش از محل کارهای دفتری از ضروریات است.
3- تجهیزات: جهت انجام کشت حتماً یک هود بیولوژیک با فیلتر هپا ضروری است. استفاده از تعدادی لامپ UV در داخل هود و همچنین فضای آزمایشگاه نیز ضروری است. لامپهای UV هرچند ماه یکبار باید تعویض شوند.
4- استفاده از روپوشهای بلند (گان) و ماسک
5- اجتناب از ایجاد آئروسل: بیشتر آلودگیها در آزمایشگاه سل بهواسطه ایجاد آئروسل پدید میآید. اعمالی چون باز کردن ظرف نمونه، پیپت کردن، سوزاندن لوپ، استفاده از شیکر و سانتریفوژ از جمله کارهایی هستند که ایجاد آئروسل مینمایند و بهتر است همگی در زیر هود انجام شده و نهایت احتیاط بهعمل آید. جهت سوزاندن لوپ قبلاً آن را در یک ارلن مایر (با حجم 500-250 میلیلیتر) که نیمی از آن از شن تمیز و نیمی با اتانول 95% پرشده، فروکرده، پس از یک دقیقه بیرون آورده و روی شعله بسوزانید. بقایای دورریختنی خلط را باید در ظروف درپیچدار (و نه فشاری) تخلیه نمود، تا باز کردن آنها موجب ترشح محتویات آنها نگردد.
6- استفاده از ماده ضدعفونیکننده مناسب: مثل فنل 5% جهت تمیز کردن سطوح مشکوک به آلودگی.
7- زبالهها باید داخل ظروف بدون درز ابتدا اتوکلاو شده و سپس در کیسههای دربسته تحویل رفتگر شوند. لوازم شیشهای نیز پس از اتوکلاو شستشو شده و سپس با فور استریل گردند. بهتر است یک اتوکلاو در خود آزمایشگاه سل موجود باشد.
مایکوباکتریوم لپره
مایکوباکتریوم لپره (M. leprae) در سال 1873 توسط هانسن توصیف شد (9 سال قبل از کشف باسیل سل توسط کخ). این باکتری عامل بیماری جذام است که امروزه در سراسر دنيا 12 تا 13 ميليون نفر به آن آلوده هستند و موارد جدید در هرسال بین 500 تا 700 هزار نفر گزارش میشود. بيماري بيشتر در آفريقا، هند، آمريكاي جنوبي، فيليپين، آسياي جنوب شرقي و پاسيفيك جنوبي ديده میشود. اين باكتري در هيچ محيط مصنوعي و كشت سلولي رشد نکرده و فقط در حيوانات، خصوصاً كف پاي موش و آرماديلو قابل رشد است.
این بیماری از طریق آئروسل، راه پوست سالم و یا زخمهای عمقی مانند خار یا گزش بندپا، انتقال توسط شیر مادر و انتقال از راه جفت به انسان منتقل میگردد. مواردی از بیماری جذام انسانی به دنبال تماس با آرمادیلو گزارش شده است. افزون بر این با کشف یک بیماری شبهجذام طبیعی در میان آرمادیلوها و این واقعیت که موارد تکگیر از جذام در افرادی که تماس مشخصی با فرد جذامی نداشتهاند، اتفاق میافتد، پیشنهاد میگردد که منابع غیرانسانی مایکوباکتریوم لپره ممکن است وجود داشته باشد. بیماری جذام به دو شکل مشخص (لپروماتوز و توبرکلوئیدی) به همراه چند مرحله بینابینی دیده میشود.
تشخيص آزمايشگاهي
1- پوستههاي حاصل از تراشيدن پوست يا مخاط بيني توسط اسكالپل يا بيوپسي نرمه گوش و مناطق دیگر را بر روي يك لام گسترده و با تكنيك ذيل– نلسون رنگآميزي نمایید.
2- گسترش تهیهشده و یا بيوپسي پوست و عصب ضخيمشده را میتوان توسط رودامین- اورامین رنگآمیزی نمود و با استفاده از میکروسکپ فلوئورسانس بهآسانی تشخیص داد.
3- اگرچه بررسی هیستولوژیک بيوپسي پوست يا عصب ضخيمشده، تصوير بافتشناسي تيپيك را نشان ميدهد، ولی هيچ آزمون سرولوژيك باارزشی وجود ندارد. تستهاي سرولوژيك غيرترپونمائي که براي تشخيص سيفليس بکار ميروند در بيماران مبتلا به جذام اغلب نتايج مثبت كاذب ايجاد ميکنند.
4- از تستهای ایمونولوژیک مانند لپرومین (بهصورت دو واکنش فرناندز و میتسودا) میتوان جهت بررسی بیماری استفاده کرد.
5- با تلقیح ضایعات جذامی به کف پای موش میتوان به بررسی روند درمان بیماری و تأیید تشخیصهای مشکوک پرداخت، همچنین با تلقیح این ضایعات به آرمادیلو میتوان باکتری را به میزان زیادی تکثیر نموده و برای مطالعات بیشتر استفاده نمود.
مايكوباكتري هاي آتيپيك
مطالعات اوليه مايكوباكتريها بر روي مایکوباکتریوم توبركلوزيس و تظاهرات معمول بيماري آن متمركز شد. ساير باكتريهايي كه گاهگاهی بهعنوان عامل بیماریهای شبهسل شناخته میشدند، از نظر مراحل، آتيپيك بودند و در تست توبرکولین نيز نتايج آتيپيكي بهوجود میآوردند. اين باكتريها را مايكوباكتريهاي آتيپيك ناميدند. اين باكتريها اعضای گروه بزرگ مايكوباكتريها هستند كه در خاك و آب يافت میشوند و اهمیتشان اين است كه جزو باکتریهای فرصتطلب محيطي هستند. مهمترین بيماريزاها در انسان عبارتند از: مايكوباكتريوم آويوم اينتراسلولار، مايكوباكتريوم كانزاسي، مايكوباكتريوم مارينوم و مايكوباكتريوم اسكروفولاسئوم. در جدول (3) بعضی از ویژگیهای مختلف این باکتریها و راههای افتراق آنها از باسیل کخ نشان داده شده است.
امروزه کمپلکس مایکوباکتریوم اویوم در ایالات متحده امریکا فراوانترین مایکوباکتریوم ایزولهشده است. این کمپلکس یک گروه هتروژنی از مایکوباکتریومها است که بهطور کلاسیک از دو گونه مایکوباکتریوم اویوم (مهمترین پاتوژن بالینی در بیماری منتشره بهصورت گسترده در افراد دچار نقص سیستم ایمنی اکتسابی با ناهنجاریهای مهم آزمایشگاهی شامل آنمی و افزایش آلکالین فسفاتاز) و مایکوباکتریوم اینتراسلولار (در بیماری ریوی اهمیت دارد) تشکیل شده است. بهتازگی مایکوباکتریوم اویوم به سه زیرگونه اویوم، سیلواتیکوم و پاراتوبرکلوزیس طبقهبندی میگردد. مایکوباکتریوم کایمرا[4]، مایکوباکتریوم کلمبینس[5]، مایکوباکتریوم وولنریس[6]، مایکوباکتریوم مارسیلنس[7]، مایکوباکتریوم تیموننس[8] و مایکوباکتریوم بوچه دوربوننس[9] از گونههای جدیدی هستند که معرفی شدهاند.
این گونهها ویژگیهای رشد و واکنشهای بیوشیمیایی مشابهی دارند و اغلب در آزمایشگاه میکروبشناسی بالینی از هم افتراق داده نشده و ایزولهها معمولاً بهعنوان کمپلکس مایکوباکتریوم اویوم گزارش میشوند.
با تکنیکهای مولکولی، امروزه بیش از 100 گونه مایکوباکتریومهای غیرسلی شناسایی شده است. مایکوباکتریومی که بهتازگی توسط Tortili شرح داده شده است و بهندرت جداسازی میگردد، مایکوباکتریوم گوردونه است که بهعنوان باسیل شیر آب[10] شناخته میشود و از آلودهکنندههای رایج آزمایشگاهی است و بهندرت در انسان بیماریزا است.
مایکوباکتریوم زنوپی[11] از شیرهای آب سرد و گرم، ژنراتور آب گرم بیمارستان و تانکهای ذخیره و سایر منابع محیطی کشت داده شده است. پرندگان یک مخزن طبیعی احتمالی هستند. بیماری ریوی و خارج ریوی که توسط این مایکوباکتریوم ایجاد شده، از نقاط مختلف جهان گزارش شده است.
مایکوباکتریوم مارینوم سبب عفونتهای گرانولوماتوز مزمن با درگیری پوست و بافت نرم شده که اغلب گرانولومای استخر یا گرانولومای حوض ماهی[12] نامیده میشود. علائم بیماری مشابه بیماری اسپورتریکوزیس است.
مایکوباکتریوم هموفیلوم[13] بدلیل نیاز به هموگلوبین و هِمین برای رشد خود در میان مایکوباکتریوم ها استثنا است. این باکتریها مشابه سایر مایکوباکتریومها سبب لنفادنیت میشوند.
مایکوباکتریوم اولسرانس سبب زخمهای بیرنسدال[14] در نواحی استرالیا و زخم بورولی[15] در نواحی اوگاندا است که با لنفادنیت و ندولهای ماهوارهای و زخمها ظاهر میگردد.
[1] Nucleic acid amplification test (NAAT)
[2] Amplified Mycobacterium tuberculosis Direct Test
[3] AMPLICOR Mycobacterium tuberculosis Test
[4] M. chimaera
[5] M. colombiense
[6] M. vulneris
[7] M. marseillense
[8] M. timonense
[9] M. bouchedurhonense
[10] Tap water bacillus
[11] M. xenopi
[12] Fish tank granuloma
[13] M. haemophilum
[14] Bairnsdale ulcer
[15] Buruli ulcer
روشهای تایپینگ مولکولی روی مایکوباکتریوم توبرکلوزیس
مقاومتهای دارویی در مایکوباکتریومها و ژنهای مرتبط با آن
تایپینگ سوشهای مایکوباکتریوم توبرکلوزیس جدا شده در بیماران با استفاده از Spoligotyping
ارزیابی روشهای تشخیص بیماری سل ریوی
https://www.sciencedirect.com/topics/medicine-and-dentistry/mycobacterium
برای دانلود پی دی اف برروی لینک زیر کلیک کنید
ورود / ثبت نام