میکرواری (ريزآرايه)
(قسمت اول)
دکتر مهدی فصیحی رامندی (استادیار دانشگاه)
زهرا کریمی (مرکز تحقیقات زیست سلول پژوهان گستر)
تعیین ترادف ژنوم جانداران مختلف، اطلاعات بسیار زیادی را در اختیار محققان قرار داده است، با وجود این هنوز نمیتوان با دقت لازم ژنها را شناسایی کرد و از طرفی پیشبینی نحوه بیان ژن از طریق اطلاعات ردیف DNA هنوز کاری مشکل و همراه با خطا است، بنابراین با دسترسی به اطلاعات ژنوم، نیاز به فناوریهایی بوده که بتوانند حجم بسیار زیادی از ژنها را تجزیهوتحلیل کنند. فناوری ریزآرایه نیز بر همین اساس توسعه یافت. فنآوری میکرواری در اوایل دهه 1990 توانست انقلاب بزرگی را در ژنومیکس ایجاد نماید و بررسی پروفایل ژنی هزاران ژن را بهصورت همزمان امکانپذیر نمود. این فناوری از ترکیب علومی مانند میکروالکترونیک، میکروفلوئیدیک، زیستشناسی مولکولی و بیوانفورماتیک حاصل شده است. میکرواری براساس نوع مولکولی که شناسایی میشود نامگذاری میگردد.
انواع میکرواریها عبارتند از: DNA array، RNA میکرواری، پروتئین میکرواری و میکرواری بافتی.
یکی از پرکاربردترین انواع میکرواری، DNA array نامیده میشود که این روش از اطلاعات توالی ژنوم و ترانسکریپتوم برای آنالیز ساختار و عملکرد ژنها استفاده میکند.
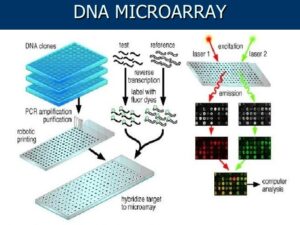
میکرواری، تکامل یافته روش بلاتینگ است. در روش بلاتینگ قطعاتی از مولکول (DNA) به یک غشا نیتروسلولزی متصل گشته و سپس توسط یک ژن مشخص، شناسایی میگردد. در میکرواری با استفاده از یک توالی تکرشته به نام پروب که بر روی یک سطح جامد (شیشه) قرار گرفتهاند، ژن را شناسایی میکنند. اختصاصیترین کاربرد میکرواری در اندازهگیری بیان ژنها است که امکان بررسی تعداد زیادی از ژنها را بهطور همزمان امکانپذیر کرده است.
پروب یک توالی اولیگونوکلئوتیدی تکرشته با اندازه 30- 18 نوکلئوتید است که میتواند بخشی از DNA، محصول PCR و mRNA باشد. در DNA array هر نقطه روی اسلاید نشاندهنده یک ژن میباشد.
میکرواری به دو مرحله اصلی تقسیم میشود:
1) آزمایشگاه مرطوب که شامل مراحل آزمایشگاهی میباشد.
2) آزمایشگاه خشک که مربوط به آنالیز دادههای میکرواری میباشد. این دو مرحله برای همدیگر لازم و ضروری هستند (شکل 1).
آزمایشگاه مرطوب شامل مراحل زیر میباشد:
1) آمادهسازی نمونه و استخراج ژنوم و یا RNA
2) تهیه چیپ میکرواری با استفاده از پروب سنتز شده
3) نشاندار کردن بهطور مستقیم یا غیرمستقیم cDNA با رنگ فلورسانس
4) هیبریداسیون شامل اتصال پروب با مولکول هدف
5) شستشو و اسکن نمونه
شکل 1: مراحل مختلف میکرواری بهصورت شماتیک
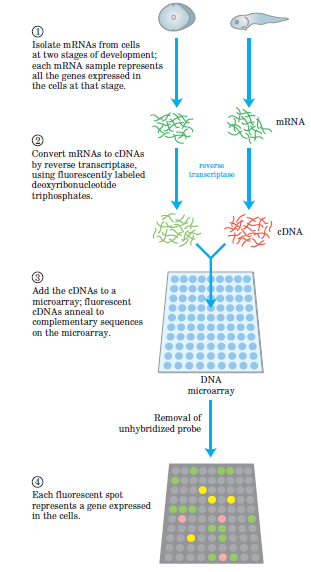
آمادهسازی مولکول هدف و نشاندار کردن آن
با توجه به هدف آزمایش، نوع مولکول استخراج شده و نحوه آمادهسازی آن متفاوت است. در واقع مولکول هدف همان نمونه مجهول در آزمایش میباشد. چنانچه قرار باشد شناسایی بر روی سطح ژنوم قرار گیرد مولکول هدف DNA و اگر میزان بیان ژن موردنظر باشد، مولکول هدف mRNA میباشد.
استخراج DNA با روشهایی از قبیل جوشاندن، الکالین فسفاتاز و کیتهای موجود در بازار امکانپذیر است و برای استخراج RNA از پروتکل مربوط به آن استفاده میشود. هردوی این مواد بر روی هیبریداسیون (دورگهسازی) اثر میگذارند، پس لازم است قبل از انجام آزمایش از لحاظ کمی کیفیت آنها بررسی شود. این کار با دستگاه اسپکتروفتومتری (نانو دراپ) انجام میشود. جذب 280/260 مربوط به DNA است که بیــــن 1/8-1/5 و جذب 230/260 مربوط به RNA میباشد که باید بین 2-1/8 باشد. برای بررسی کیفی میتوان از الکتروفورز استفاده کرد. بعد از اینکه از خلوص و غلظت نمونه استخراج شده اطمینان حاصل شد، نوبت به نشاندار کردن آن با مواد فلورسانس است. در میکرواری دو نمونه با همدیگر روی یک اسلاید مقایسه میگردند (نمونه سالم و بیمار). پس باید هرکدام از نمونهها با یک رنگ متفاوت نشاندار شود، به این مکانیسم میکرواری دو کاناله میگویند. رنگ رایجی که بیشتر مورد استفاده است Cy3 و Cy5 میباشد. رنگ Cy3 در طولموج nm 523 به رنگ سبز و رنگ Cy5 در طولموج nm 635 به رنگ قرمز دیده میشود.
نشاندار کردن به روش مستقیم بیشتر استفاده میشود که در حین سنتز رنگ به محیط اضافه شده و وارد زنجیره در حال سنتز میشود، تمایل رنگ Cy3 نسبت به Cy5 بیشتر است. در روش غیرمستقیم از dUTP که آنالوگ dTTP است و دارای یک گروه آمین است، استفاده میگردد. در رشته cDNA سنتز شده dUTP وارد میشود، پس بهجای باز تیمین، یوراسیل قرار میگیرد. در مرحله بعد رشته با رنگ فلورسانس کوپل میگردد. بعد رنگهای کوپل نشده حذف و در نهایت یک رشته فلورسانس بدست میآید (شکل 2).
در نمونههایی که بهصورت mRNA میباشند باید برای انجام آزمایش به cDNA تبدیل شوند که این کار به دو علت است:
1) پایداری cDNA از mRNA بیشتر است و کار کردن با آن راحتتر است.
2) نشاندار کردن cDNA راحت است و این کار را میتوان با dNTP نشاندار در حین سنتز انجام داد.
شکل 2: مراحل آمادهسازی نمونه و تبدیل به cDNA و نشاندار کردن آن که بهصورت شماتیک نمایش داده شده است
ساخت و طراحی چیپ میکرواری
اسلاید یا چیپ یکی از اجزای اصلی آزمایش میباشد. برای ساخت اسلاید علاوه بر دستگاه اسپاتر برای قرار دادن توالی نوکلئوتیدی نیاز به بستر شیشهای از جنسهای مختلف مانند الومینوسیلیکات، زینک تیتانیوم، سیلیکا و بروسیلیکات برای اتصال پروب داریم.
امروزه از سه فناوری برای ساخت چیپها استفاده میشود:
- فتولیتوگرافی
- نقطهگذاری مکانیکی
- Ink jet
روش فتولیتوگرافی
فتولیتوگرافی در صنایع تراشههای رایانهای بهخوبی شناخته شده است. یکی از شرکتهای اصلی در این زمینه Affymetrix میباشد. روش ساخت چیپ میکرواری با استفاده از این روش به این گونه است که پروب نوکلئوتید به نوکلئوتید سنتز میشود. تمام چیپ با یک ماده حساس به نور پوشانده میشود که از اتصال نوکلئوتید به شیشه جلوگیری کند. در این روش، از یک منبع نور فرابنفش برای ساخت اولیگونوکلئوتیدها بر روی یک سطح شیشهای سیلیکونی استفاده میگردد. با استفاده از این فناوری میتوان هزاران اولیگونوکلئوتید به طول 50-25 نوکلئوتید را بر سطحی با اندازه یک سانتیمتر مربع قرار داد.
روش نقطهگذاری مکانیکی
بر پایه قرار دادن فیزیکی مواد بر روی سطوح با استفاده از سوزنهای مخصوص استوار است. با توجه به اینکه مبنای کار این سیستم بر اساس تماس مستقیم سوزن با مقادیر کم مایع و انتقال آن بر اسلایدهای شیشهای است، امکان آلودگی به ذرات گردوخاک و تبخیر مایعات وجود دارد. برای ایجاد شبکهای از DNA بر روی اسلایدها، از بازوی روباتی استفاده میشود که در سه محور Y ,X و Z حرکت میکند. مواد که حاوی DNA هستند در ظرفی که 96 یا 384 چاهک دارد، قرار داده شده و سپس این بازو با توجه به الگویی که به آن داده میشود، نقاط را بر روی اسلاید شیشهای قرار میدهد. شایان ذکر است که در نخستین مقالهای که در زمینه ریزآرایه منتشر شد، عبارت هدف برای توصیف DNA روی اسلاید (محصول PCR) و عبارت کاوشگر برای توصیف نمونه mRNA که با مواد فلورسانس نشاندار شده، به کار رفت. البته در کاربرد این لغات کمی اختلافنظر بین محققان وجود دارد و برخی از آنها عکس این حالت را به کار میبرند.
در این روش از یک بازوی روباتیک برای انتقال پروب استفاده میشود. در این روش پروب قبلاً سنتز شده و توسط اسپاتر منتقل میگردد. این روش به دلیل ساده بودن بیشتر در آزمایشگاههای تحقیقاتی استفاده میشود. با استفاده از این روش میتوان پروبهایی با طول 60 نوکلئوتید ایجاد کرد.
روش Ink jet
این روش مشابه روش قبل است، با این تفاوت که پروب مستقیم به سطح اسلاید متصل نمیشود. در این روش یک ترکیب شیمیایی به سطح متصل میشود و پروب به آن متصل میگردد.
در کل دو حالت برای اتصال وجود دارد:
1) پروب بهصورت غیرکووالانسی یا با برقراری پل نمکی با پوشش شیمیایی سطح اسلاید پیوند برقرار کند. 2) پروب بهصورت کووالانسی به پوشش شیمیایی سطح اسلاید متصل شود که این اتصال محکمتر بوده و پیوند قویتری ایجاد میکند. برای این کار در انتهای 5 پروب توالی آمینی قرار میدهند و برای اینکه ممانعت فضایی ایجاد نشود از یک ترکیب 6 کربنه برای ارتباط بین پروب و شیشه استفاده میکنند. معمولاً برای پروبهایی با طول حدود 60 نوکلئوتید میتوان از پوشش الدهیدی یا اپوکسی استفاده کرد و برای پروبهایی که محصول PCR و یا ژنوم که طویل هستند از پوشش آمینی استفاده میشود.
تکنیکهای تکثیر چندگانه کاوشگر
الایزا ، روشهای کنترل کیفی و مدیریت خطا
برای دانلود پی دی اف بر روی لینک زیر کلیک کنید
ورود / ثبت نام