آنالیز نمودار منحنی ذوب با دمای بالا
(High Resolution Melt Curve Analysis)
دکتر رضا میرنژاد (دانشیار دانشگاه) – وهاب پیرانفر (محقق)
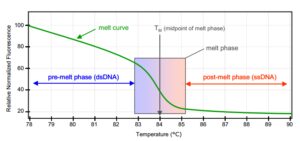
آنالیز نمودار منحنی ذوب با دمای بالا (High resolution melt curve analysis (HRM)) یک روش ساده و مقرون به صرفه و Post-PCR است که با اسکن کردن موتاسیونها و ژنوتایپینگ معروف شده است. این تکنیک بر اساس دمای ذوب محصولات PCR شکل گرفته است؛ یعنی دمای ذوب یک DNA دو رشتهای. دماي ذوب DNA يك شاخص ويژه براي اين مولكولها است كه به ساختمان آن و عواملي نظير طول و تعداد نوكلئوتيد، غلظت پروب، ميزان غلظت نمك محيط و درصد نوكلئوتيدهاي سيتوزين وگوانين بستگي دارد. از مزاياي Real-Time PCR تعيين منحني ذوب (High Resolution Melt Curve Analysis) است كه پس از پايانPCR ترسيم و براي تأييد نتايج استفاده ميشود و در صورت وجود اختلاف در ترادف نوكلئوتيدي دو نمونه شكل منحني تفاوت ميكند. با مقايسه منحني نمونهها با هم يا نمونههاي نرمال (توالييابي شده) موارد متفاوت مشخص ميشوند و در اين منحني هر يك از قلهها نمايانگر نقطه ذوب يك محصول PCR است كه اين كار بوسيله اندازهگيري تغييرات فلورسانس در دماهاي مختلف صورت ميگيرد. از ديگر مزاياي منحصر به فرد اين تست سرعت و دقت بالا و هزينه پايين آن ميباشد. در شکل زیر اصول منحنی HRM در شکل شماتیک نشان داده شده است. منحنی ذوب (سبز رنگ) دارای فلورسانس بالایی است که مرحله پیش از ذوب میباشد، سپس با افزایش دما و باز شدن دو رشته DNA کاهش شدید فلورسانس را میتوان مشاهده کرد. این کاهش تا زمان تک رشتهای شدن DNA ادامه پیدا میکند. بر اساس این کاهش رنگ فلورسانس و رابطه آن با دما، میتوان به تجزیه و تحلیل دادهها و اسکن قطعه ژنی پرداخت.
شکل 1: شکل شماتیک از منحنی HRM
رنگهای مورد استفاده در HRM
راز آنالیز منحنی ذوب با قدرت تفکیک بالا در بررسی نمودار در زمان واقعی است؛ یعنی آنالیز و تفسیر در حین انجام آن. این اتفاق با استفاده از رنگهای فلورسانس اشباع ممکن شده است. این رنگها به عنوان رنگهای intercalating dyes شناخته شدهاند که میتوانند به طور تخصصی و در مقدار بالا بهDNA دو رشتهای متصل شوند. اگر DNA دو رشتهای در محیط وجود نداشته باشد آنها قابلیت نشر نور در سطح بسیار پایین در حد 1 تا 4 درصد را بیشتر نداشته و پارازیت دستگاه محسوب میشوند. برای شروع تجزیه و تحلیل HRM پس از پایان رساندن PCR سطح بالایی از فلورسانس به دلیل باند شدن رنگ، میلیونها نسخه از محصول PCR را شاهد هستیم، بنابراین با حرارت دادن نمونه از حالت دو رشته به تک رشتهای میزان فلورسانس محیط با آزاد شدن رنگهای اشباع از زنجیره دو رشتهای DNA، کاهش فلورسانس را انتظار داریم. این اساس کار HRM به سادگی است. رنگهای زیر برای HRM در حال حاضر موجود هستند که برای مصارف گوناگون این تکنیک به کار میروند.
LC Green
SYTO9
Eva Green
Chromofy
BEBO
SYBR Green
HRM assays with raZor probe
یکی از رنگهائی که کاربرد وسیع دارد رنگ Eva Green است که در اینجا به شرح تفصیل رنگ پرداخته میشود.
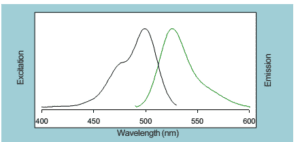
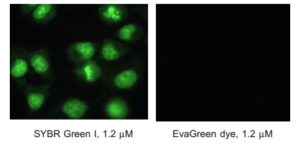
Eva Green یــک رنگ سبـــــز فلورســـانس بــرای اسیــد نــوکلئیــک اســـت کــه در بیــــولوژی مولــکولی کاربــردهای فراوان دارد. از ایــــــــن رنگ در qPCR، HRM، Real-time monitoring و Routine solution DNA Quantification and capillary gel electrophoresis استفاده میشود. این رنگ میتواند به DNA متصل شده و نشر نزدیکی به رنگهایی چون FAM و SYBR® dye Green I دارد. سمیت این رنگ البته از دو رنگ مذکور بسیار کمتر است. این رنگ در طول موج 488 نانومتر قابل شناسایی است (شکل 2). رنگ ایواگرین به حرارت و هیدرولیزشدن مقاوم بوده و بسیار پایدار است. البته باید گفته شود که رنگ به صورت ذاتی نشر فلورسانس نداشته، اما پس از اتصال به DNA دو رشته، میتواند به شدت فلورسانس شود. Eva Green سمی نبوده و موتاژن نیست و نسبت به غشاء نفوذ ناپذیر است (شکل 3).
شکل 2: طیف طول موجهای تحریک و برانگیختگی (چپ) و انتشار (راست) رنــــــگ Eva Green
شکل 3: عدم نفوذپذیری در غشاء. سمت راست رنگ ایواگرین و سمت چپ رنگ سایبرگرین
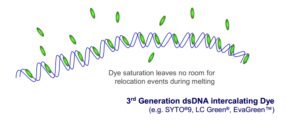
درک HRM میــسر نیــست مــگر اینــکه مکانیــسـم رنــــگهــا و اتــصـــال بــــه DNA دو رشــــتهای را در تصــور خـــود داشـــته باشــــــید. نســــل ســــوم رنــــگهای اشــــباع مــانــند SYTO 9 (Invitrogen Corp, Carlsbad, CA), LC Green (Idaho Technologies, Salt Lake City, UT) and Eva Green (Biotium Inc, Hayward, CA)به خوبی در شکل 4 نشان داده شدهاند.
شکل 4 : نحوه عملکرد نسل سوم رنگهای اشباع در مواجهه با DNA
نرمال کردن منحنی تفکیک (HRM data normalizationshape & shift)
دو روش وجود دارد که میتوان منحنی HRM را بر اساس آن تفسیر کرد:
الف) شکل (Shape): به عنوان مثال مقایسه شکل دو نمودار یک نمونه با یکدیگر
ب) شیفت (Shift): بررسی و مقایسه نمودار بر اساس سلسلههای دمایی
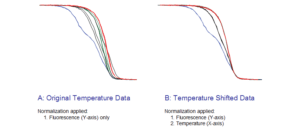
قبل از رسم منحنی اصلی HRM دادهها باید به صورت نرمال شده درآیند. این نرمال شدن بر اساس نشر فلورسانس روی محور Y و درجه حرارت روی محور X میباشد. این کار شبیه در نظر گرفتن زمان واقعی تکثیر قطعه PCR است، منتهی این بار به جای تعداد چرخه، دما در نمودار لحاظ میشود. این عمل در یک مقیاس 0% تا 100% صورت میگیرد (شکل 5).
شکل 5: نرمال کردن منحنی با استفاده از اطلاعات خام
همانطورکه در بالا اشاره شد تکنیک HRM یک روش قدرتمند در میان تکنیکهای مولکولی است. از این روش برای شناسائی موتاسیونها، پلیمورفیسم (چند ریختیهای ژنی) و اپیژنتیک استفاده میکنند. این تکنیک بر پایه تفکیک تفاوتهای دو رشتهایDNA نمونهها ساخته شده است. این تکنولوژی در دانشگاه اوتای آمریکا و در آزمایشگاه ایداهو اختراع و بهرهبرداری شده است. دانشمندان از این تکنیک برای ژنوتایپینگ گونهها نیز استفاده میکنند.
مقرون به صرفه بودن، سریع بودن و دقت به اندازه توالی یابی، باعت شده است که از این تکنیک در ژنوتایپینگ به سرعت استفاده شود، همین طور برای پیدا کردن SNPها. این تکنیک توانایی اسکن کردن ژنوم با دقت فراوان را داراست. در جدول زیر کاربردهای مختلف این روش نشان داده شده است.
جدول 1: کاربردهای مختلف تکنیک HRM
|
|
با استفاده از HRM در برنامههای بالا، کاهش هزینه نسبت به روشهای مانند ® TaqMan و طراحی پروب قابل توجه است، چرا که در این روش از رنگهای اشباع مانند سایبرگرین استفاده میکنند. همین طور به نظر میرسد که این روش بسیار سادهتر و نتایج حاصل از آن قابل تفسیرتر است. به عنوان مثال چندین نمودار زیر انواع نمودار HRM را نشان میدهند که قابل تفسیر هستند.
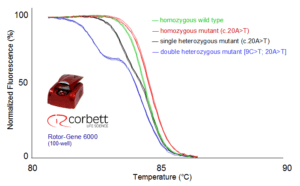
شکل 6: با استفاده از دستگاه Rotor-Gene همه چهار ژنوتیپ به خوبی از هم قابل افتراق هستند.
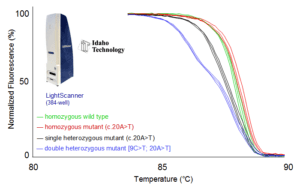
شکل 7: با استفاده از دستگاه LightScanner تنها هتروزیگوتها را میتوان از هم افتراق داد.
برای دانلود پی دی اف بر روی لینک زیر کلیک کنید
ورود / ثبت نام