ادامه مبحث تایپینگ مولکولی
REP-PCR
اساس کار Rep-PCR قطعات تکراری DNA در ژنوم پروکاریوت میباشد. این قطعات تکراری محلی برای هیبریداسیون پرایمرها بکار رفته در Rep -PCR هستند. پرایمرها که مکمل با سکانسهای تکراری منتشر میباشند بعد از آمپلیفیکاسیون با PCR، قطعات DNA در اندازههای متفاوت ایجاد میکنند که در الکترفورز از همدیگر جدا میگردند. الگوهای الکترفوروتیک برای تعیین ارتباط سویههای باکتریها با یکدیگر مورد استفاده قرار میگیرند. یک مجموعه پرایمر برای قطعات تکراری متفاوت استـــــــفاده ميشود. Rep-PCRدر انواع ایزولههای اشریشیاکلی انتروهموراژیک (EHEC) جهت اثبات انتقال آنها از حیوان به انسان مورد استفاده قرار گرفته است. این تکنیک همانند سایر تکنیکها در تیپ بندی باکتریها، برای پیگیری باکتریهای گرم منفی و گرم مثبت که مقاوم به عوامل ضد میکروبی میباشند و در اپیدمیولوژی عفونت بیمارستانی مهم هستند، كاربرد دارد، همچنین اطلاعات بدست آمده از Rep-PCR جهت شناسائی میکروارگانیزمها مورد استفاده قرار میگیرد. برای مثال این تکنولوژی در شناسائی ارگانیزمها و ارزیابی تنوع ژنهای استرپتومایسس و شناسائی گونههای بوردتلا بكار رفته است. همچنین برای انواع پاتوژنهای بیمارستانی مانند اشریشیاکلی، استافیلوکک اورئوس و انتروکوکها مورد استفاده قرار میگیرد.
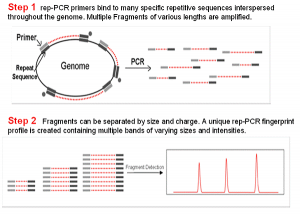
در این تکنیک نواحيی که بین قطعات تکراری قرار گرفته است از نظر اندازه متفاوت هستند، لذا بعد از انجام PCR و الکترفورز الگوهای متفاوت ایجاد میکنند که براحتی با مقایسه این الگوها میتوان سویهها را از یکدیگر تمایز و تشخیص داد. (شکل 1)
شکل 1: مراحل مختلف اجرای Rrp- PCR
Variable Number Tandem Repeat (VNTR):
تیپ بندی توالی انتهائی با تعداد متغیر (VNTR) به همراه اسپولیگوتایپینگ (Spoligotyping) برای مایکوباکتریم توبرکلوزیس مورد استفاده قرار میگیرد. در ژنوم این باکتری، نواحی غنی از توالی مستقیم با فاصله تکراری و سکانسهای غیرتکراری یا الیگونوکلئوتیدهای فاصله انداز (Speacer) قرار دارند (شکل 2). در اسپولیگوتایپینگ نواحی تکراری محافظت شده به عنوان اهداف پرایمرهای PCR جهت آمپلی کردن سکانسهای فاصله انداز بكار ميروند. محصولاتي که با پرایمرهای بیوتیندار آمپلی میشوند برای هیبریداسیون با یک پانل حاوی الیگونوکلئوتیدهای سنتیتیک (که معرف سکانسهای فاصله انداز میباشد) متصل به غشاء استفاده میگردند. محصولات PCR به مکمل خود با سکانسهای الیگونوکلئوتید باند میکنند و پروفایل هیبریداسیون متفاوتي ایجاد میکنند که برای ژنوتایپینگ ارگانیزمهای مختلف مورد استفاده قرار میگیرند.
شکل2: نمائی از VNTR در 4 الل
VNTR تایپینگ، شکلی از ژنومهای باکتری که دارای سکانس کوتاه، تکراری و tandem میباشند را مورد استفاده قرار میدهد. تعداد کپی از سکانسهای VNTR اغلب در میان سویههای غیروابسته متفاوت میباشد که از روی این خصوصیات میتوان براي ژنوتایپینگ ارگانیزمها استفاده کرد. اغلب پرایمرهای نشاندار با فلورسنت برعلیه این نواحی تکراری کامل طراحی میشود. بدنبال آمپلی فیکاسیون، محصولات PCR اغلب با سکانسهای اتوماتیک جدا میشوند و جهت تعیین تعداد نواحیهای تکراری آنالیز و ارزیابی میگردند. بطور تیپیک نواحی با توالی زیاد جهت تعیین ژنوتیپ آنالیز میشوند. تعدادی پاتوژنهای بیمارستانی وجود دارند که میتوان آنها را با تکنیک VNTR تیپ بندی کرد. (شکل 3)
شکل 3: نمای کلی از اجرای تکنیک VNTR
IS-PCR, BOX-PCR, ERIC-PCR:
Enterobacterial Repetitive Intergenic Consensus و سکانسهای داخل شــــــونده روشهای تیپ بندی بر اساس PCR هستند که در آنها سکانسهای حفظ شده و توالیهای تکراری در باکتری یا در بعضی موارد در قارچها مشاهده میشود. در سال 1991، Versalovic و همکارانش به حضور سکانسهای تکراری در رنج وسیعی از گونههای باکتری اشاره کردند که میتوانند مستقیماً جهت انگشت نگاری ژنوم باکتری مورد استفاده قرار گیرند. سکانسهای تکراری خاصی شامل سکانس ERIC با 127-124 جفت باز و سکانسهای BOX با 154 جفت باز میباشند. این سکانسها عموماً در سراسر کروموزم به صورت داخل ژنی وجود دارند. بعضی سکانسهای تکراری به سکانسهای جدیدی در ژنوم نقل مکان میکنند که به آنها ســکانسهــای داخـل شـونده (IS) یـا تـرانســپوزون میگــــــــــــــــــــــویند. اصــول تیپ بندی IS-PCR, BOX-PCR, ERIC-PCR همانند VNTR میباشد که قبلاً توضیح داده شد. در همه این تکنیکها تیپ بندی براساس توزیع راندم قطعات داخل ژنوم است که بر اساس آنها میتوان ارگانیزمها را از یکدیگر متمایز کرد. یعنی اینکه باکتریها در میزان فراوانی توالی تکراری مختلف هستند و لذا الگوهای متفاوت بعد از انجام PCR با پرایمرهای خاصی در الکترفورز ایجاد میکنند. در این تکنیکها پرایمرها طوری طراحی میشوند که به قطعات تکراری متصل شوند و فاصله بین دو قطعه تکراری که پرایمرها به آنها متصل شده است را آمپلی میکنند، لذا تغییر در ارگانیزمهای غیروابسته ناشی از تعداد و مکانهای راندم این عناصر در ژنوم میباشد. در جدول زیر بخشهای تکراری در برخی باکتریها ارائه شده است.
جدول 1: موتیفهای تکراری در باکتریها
| نام | طول (bp) | باکتری |
| پالیندروم تکراری خارج ژنی (REP) | 38 | اشریشیاکلی، سالمونلا تیفی موریم |
| Consensus تکراری داخل ژنی انتروباکتریا | 127-124 | اشریشیاکلی، سالمونلا تیفی موریم |
| NgREP | 26 | نایسریا گونوره |
| DNREP | 192-150 | دینیکوکوس رادیودورانس |
| MXREP | 87 | میکسوکوکوس گزانتوس |
| REPMP1 | 300 | مایکوپلاسما پنومونیه |
| SDC1
|
400 | مایکوپلاسما پنومونیه |
مقایسه و انتخاب یک تکنیک:
هنگامی که میخواهیم یک تکنیک مولکولی جهت تایپینگ یک ارگانیزم بکار ببریم بایستی به این نکته توجه داشته باشیم که آیا این تکنیک به رسیدن به اهداف ما کمک میکند و آیا به سؤالات ما پاسخ میدهد؟ در یک بیمار خاص، کاربرد ویژگیهای مولکولی در جداسازی عود از آلودگی مجدد یا در موارد باکتریمی (آیا این ناشی از آلودگی میباشد یا ناشی از عفونت سایر نقاط بدن) به ما کمک زیادی میکنند. مثلاً PFGE میتواند در یک گروه در اثبات ارتباط کلونی که در ارزیابی انتشار ارگانیزم از یک فرد به فرد دیگر مهم است، کمک کننده باشد. هنگامی که به انتشار ژن یا ژنهای مقاومت خاص مشکوک هستیم، آنالیز پلاسمید یا ترانسپوزون موجود در سویهها کمک کننده هستند. از آنجائیکه استفاده از تکنیکهای مولکولی جهت تیپ بندی باکتریها هزینهبر و وقتگیر میباشند، لذا بایستی همیشه یک دید مناسب در محدودیتهای روشهای تایپینگ مولکولی مختلف وجود داشته باشد تا بتوان به طور کامل و مناسب باکتریها را تیپ بندی کرد. هنگامی که روشهای مولکولی متفاوت را با همدیگر مقایسه میکنیم، بایستی این نکته مهم را توجه داشت که هر روش واقعاً چه چیز را ارزیابی میکند؛ مثلاً الگوهای قطعات برش خورده در PFGE نشان دهنده تفاوت بین فراوانی سایتهای برش در طول کروموزم میباشند، بنابراین هرگونه تغییر در کروموزوم که روی این سایتهای برش اثر میگذارند سبب بوجود آمدن الگوهای متفاوت میشوند و چون در ارگانیزمهای مختلف این سایتها متفاوت هستند به راحتی ایزولهها را میتوان از همدیگر افتراق داد. PFGE که حدود 90% از کرموزوم را اسکن میکند (مجموعه اندازههای قطعات برش خورده) دارای حساسیت متوسط میباشد، چرا که تغییرات ژنتیکی کوچک ممکن است در آن قابل جداسازی نباشد. برعکس روشهای مبتنی بر PCR عموماً نواحی نسبتاًًًً محدودی (کمتر از 10% کروموزمی) را شامل میشوند و مجموعه اندازه قطعات آمپلی شده را نشان میدهند. از آنجا که محصولات PCR عموماً کوچک هستند (برابر یا کمتر از 5 کیلوباز) آنالیز الکتروفوروتیک آنها حتی تغییرات ژنتیکی کوچک را که در اندازه محصول PCR اثر میگذارند، جداسازی مینمایند. باید توجه داشت اگر تغییرات در کروموزم خارج از قطعه مورد نظر باشد، قابل جداسازی نيست.
بعضی از قطعات ژنتیکی در پاتوژنهای بیمارستانی از یک فرد بیمار به فرد بیمار (سالم يا ديگر؟) منتقل میشود. با PFGE میتوان تغییرات کروموزمی قابل جداسازی تکی که به صورت تغییر در حداقل دو جای باند مشاهده میشود را جداسازی کرد. روشهای PCR حداقل تفاوت یک جا را (مانند حذف یا از دست دادن یا شیف در حرکت الکترفوروتیک یک باند) جداسازی می کنند. اگر اختلاف ژنتیکی با يكدیگر تفاوت زيادي داشته باشند میتوان نتیجه گرفت که این ایزولهها از همدیگر متفاوت هستند.
امروزه بانکهای اطلاعات مختلفی در دسترس میباشد که تغییرات بسیار جزئی در ژن یا پروتئینها را در خود دارند و میتوان در بررسی اپیدمیولوژی عفونتها به خصوص عفونتهای بیمارستانی از آنها استفاده کرد.
برای دانلود پی دی اف بر روی لینک زیر کلیک کنید
ورود / ثبت نام