نشانگرهای مولکولی و تكنيكهاي نقشهبرداري ژنومي و انگشتنگاري DNA
دکتر مهدی فصیحی رامندی (عضو هیئت علمی دانشگاه علوم پزشکی بقیها… (عج))
زهرا کریمی (مرکز تحقیقاتی زیست سلول پژوهان تدبیر)
دکتر رضا میرنژاد (استاد دانشگاه)
بر اساس تعریفی که استنسفلد در 1986 ارائه داد، واژه نشانگر معمولاً برای نشانگرهای لوکوسی بهکار میرود. هر ژنی جایی در طول کروموزوم به نام لوکوس دارد. ژنها میتوانند از طریق جهش به چندین شکل متفاوت تبدیل شوند که آلل (یا شکلهای آللی) نامیده میشوند. تمامی شکلهای آللی یک ژن در یک جایگاه در کروموزومهای هومولوگ قرار میگیرند. هنگامی که شکلهای آللی یک لوکوس یکسان باشند، گفته میشود که ژنوتیپ، هموزیگوت (در این لوکوس) است؛ در حالی که شکلهای آللی متفاوت، هتروزیگوت را ایجاد مینمایند. در موجودات دیپلوئید، ژنوتیپ با دو شکل آللی کروموزومهای همولوگ ایجاد میگردد؛ بنابراین نشانگرهای مولکولی عبارتند از تمامی نشانگرهای لوکوسهای مربوط به DNA (نشانگرها میتوانند بیوشیمیایی یا مورفولوژیک نیز باشند).
نشانگر مولکولی مناسب برای ژنتیک جمعیت
یک نشانگر مولکولی مناسب باید:
- توارث مندلی داشته باشد: یعنی از نسلی به نسل دیگر منتقل شود.
- پلیمورفیک باشد: یعنی آللهای متعددی را در لوکوس مورد بررسی نشان دهد.
- همبارز باشد: یعنی بتوان هموزیگوت و هتروزیگوت را از هم تشخیص داد.
- خنثی باشد: یعنی همه آللها شایستگی یکسانی داشته باشند.
- اپیستازی نداشته باشد: یعنی بتوان بدون توجه به سایر لوکوسها ژنوتیپ هر فنوتیپی را تعیین نمود.
- مستقل از محیط باشد: یعنی فنوتیپ از محیط تأثیری نپذیرد.
- در طول ژنوم زیاد تکرار شود.
- تکرارپذیری بالایی داشته باشد.
انتخاب یک نشانگر مولکولی به مناسب بودن آن برای پاسخ دادن به یک پرسش اکولوژیک بستگی دارد؛ بنابراین آنها برای تخمین جریان ژنها بین جمعیتها مثلاً بهمنظور بررسی تنوع مناسبتر هستند یا ترجیح داده میشوند. از طرف دیگر نشانگرهای غالب میتوانند ژنوتیپ را تخمین بزنند، اما توانایی تعیین فراوانیهای آللی را ندارند. نشانگرهای غالب ترجیحاً برای انگشتنگاری بهکار رفته و میتوانند در شناسایی کلونها مفید باشند.
نشانگرهاي مولكولي در نقشهبرداري ژنتيكي كاربرد زيادي پيدا كردهاند. اين نشانگرها در سيستمهاي مختلفي بهكار گرفته شدهاند و همين امر اساس تكنيكهاي تشخيصي متعددي شده است كه از بين آنها ميتوان به موارد زير اشاره نمود:
- Restriction Fragment Length Polymorphism (RFLP)
- Random Amplified Polymorphic DNA (RAPD)
- Simple Sequence Repeat or microsatellite (SSR)
- Amplified Fragment Length Polymorphism (AFLP)
- Single-nucleotide polymorphism (SNP)
- Sequence-Tagged Sites (STS)
- Sequence-characterized amplified region (SCAR)
- Cleaved amplified polymorphic sequences (CAPS)
همچنین میتوان به نشانگرهای آلوزیمی، انگشتنگاری مینیساتلیتها و مایکروساتلیتها اشاره نمود.
پليمورفيسم موجود در نوكلئوتيدهاي يك لوكوس، پايهي توسعه تكنيكهاي نقشهبرداري ژنومي و انگشتنگاري DNA است. اين پليمورفيسمها توسط روشهاي مولكولي تعيين ميشوند كه اين روشها به دو دسته عمده بر پايه هيبريداسيون و بر پايه PCR تقسيم ميگردند. در بهکارگیری اين روشها بايد به مواردي از قبيل سهولت انجام كار، هزينه، دقت و صحت آزمايش توجه كرد.
تعيين ژنوتيپ SNP چندگانه
تخمين زده ميشود كه بيش از 10 ميليون SNP با فراواني بيش از 1% در ژنوم انسان وجود دارد. بعضي از اين چندشكليها، به همراه فاكتورهاي محيطي، مستعدكنندۀ ابتلا به بيماريهاي مختلف از قبيل ديابت، بيماريهاي قلبي عروقي و سرطان ميباشند. بهمنظور ساخت يك منبع اطلاعاتي جامع موردنیاز در مطالعات همراهي ژنتيك با بيماريهاي مختلف، پروژه بینالمللی Hap-MAP مجموعۀ كوچكي در حدود 500 هزار برچسبSNP[1] را شناسايي كرده است كه اين چندشكليها براي مطالعه بيماريهاي پيچيده مورد استفاده قرار ميگيرند. امروزه نياز شديد به يك روش مقرون به صرفه و كارآمد براي تعيين گستردۀ ژنوتيپ SNPها در سطح وسيع احساس ميشود.
روش تعيين ژنوتيپ Illumina، با استفاده از تكنيكهاي مختلف، تعيين ژنوتيپ را در سطح گسترده مقدور ساخته است. اين روش حاوي آرايههاي مينياتوري است كه با استفاده از اسكنر داراي قدرت تفكيك بالا، اين آرايهها خوانده شده و تعيين ژنوتيپ SNP چندگانه انجام ميشود. اين تركيب تكنيكها باعث سهولت كار، بهبود كيفيت و كاهش هزينه آزمايش ميشود. در اين بخش به بررسي اين تكنيك ميپردازيم.
تكنيك تعيين ژنوتيپ Illumina:
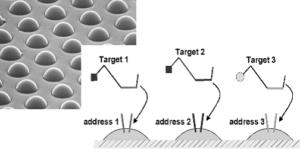
ماتريكس مورد استفاده در اين روش حاوي 96 آرايۀ مينياتوري است كه هر كدام از آنها 1/2 ميليمتر قطر دارند. اين ماتريكس، 8 × 12 بوده و براي جفت شدن با پليتها ميكروتيتر مناسب است. هر كدام از اين آرايههاي مينياتوري حاوي 50 هزار رشته فيبر نوري متصل به هم هستند. اين رشتههاي نوري به هم متصل شده و يك آرايه را تشكيل ميدهند. اين آرايهها را با استفاده از روشهاي شيميايي برش ميدهند تا در انتهاي هر فيبر، چاهكهايي به قطر 3 ميكرون تشكيل شود. هر كدام از اين چاهکها تقریباً 5 ميكرون با هم فاصله دارند. درون اين چاهكها، ذرات ريزي قرار ميگيرند و اوليگونوكلئوتيدها بهطور كووالان به اين ذرات متصل هستند (مطابق شكل زير).
شکل 1: نحوه اتصال اولیگو به میکروپلیتها و تشکیل آدرسها
هر اوليگو به يك نوع از اين ذرات متصل شده و اين ذره يك آدرس را تشكيل ميدهد كه براي اتصال و تشكيل دورگه با محصولات آرايه طراحي شده است (شکل 1). تعداد چاهكها در اين مينياتور، انواع ذرات را تعيين ميكند. بهطور معمول 1624 اوليگونوكلئوتيد منحصر به فرد براي اتصال به ذرات استفاده ميشود، بنابراين بهطور متوسط به ازاي هر نوع ذره، 30 عدد از ذرات قرار ميگيرد، در نتيجه اين تعداد اضافۀ ذرات موجب ميشود كه نتايج حاصل بهتر باشد. اين ذرات بهطور تصادفي بر روي آرايه مينياتوري قرار گرفته، دورگهسازي صورت ميگيرد تا نوع ذرات موجود بر روي آرايه مشخص شود. طي اين فرايند علاوه بر تشخيص نوع ذرات، يك كنترل كيفي از هر ذره صورت ميپذيرد. اندازۀ كوچك ماتريكس، طراحي و ساخت اسكنر ليزري اختصاصي براي خواندن اين آرايههاي چگال را موجب شده است. قدرت تفكيك اين اسكنر 0/8 ميكرون است. اين اسكنر داراي دو ليزر تهييج كننده است (با طولموجهای 535 و 635) و بهطور اتوماتيك عكسهايي را با دو كانال رنگي ميگيرد. نرمافزار اين اسكنر بهطور اتوماتيك از هر 96 آرايۀ مينياتوري عكس گرفته و ثبت ميكند، سپس اطلاعات موجود در اين عكسها با استفاده از نرمافزار مخصوص، استخراج شده و ژنوتيپ نمونهها تعيين ميشود.
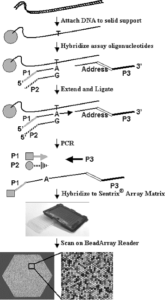
روش Golden Gate براي بررسي همزمان چندين SNP طراحي شده است. تلاشهاي قبلي براي توسعه روشهاي تعيين همزمان ژنوتيپ چندينSNP، بهصورت تكثير افتراقي آلل و يا تكثير آلل و سپس بررسي انواع آللها بود، اما اين روشها داراي محدوديتهايي از قبيل اختصاصي جايگاه[2] و افتراق آللي[3] در تكثير بودند. اين روشها همگي تغيير كرده و بهبود يافته است تا بتواند با موفقيت تعداد زيادي از چندشکلیها را بهطور همزمان شناسايي كند. اين كار با جداسازي فرايندهاي اختصاصي جايگاه و افتراق آللي از فرايند تكثير سيگنال انجام ميشود. جداسازي اين مراحل، اين امكان را به وجود ميآورد تا بتوان هر كدام از مراحل را بهطور مستقل از ديگري تنظيم كرد؛ مثلاً دورگهسازی اوليگو بهطور مستقل از افتراق آللي تنظيم ميشود. از طرف ديگر افتراق آللي نيز مستقل از دورگهسازی و تكثير سيگنال تنظيم شده و شرايط حاكم بر آنها تأثیری بر افتراق آللي نميگذارد.
در روش Golden Gate ابتدا DNA به ذرات مغناطيسي متصل شده و اين ذرات بهعنوان بستري براي خالصسازی مورد استفاده قرار ميگيرند. سه اوليگو براي هر SNP ساخته ميشود: دو اوليگوي اختصاصي آلل [4](ASO) و يك اوليگوي اختصاصي لوكوس [5](LSO). دو اوليگوي اختصاصي آلل در انتهاي ‘3 خود داراي باز مكمل براي ناحيه SNP ميباشند. هر ASO در انتهاي ‘3 خود، باز مكمل يكي از دو آلل SNP را دارد. انتهاي ‘5 هر ASO نيز مكمل يكي از دو آغازگر عمومي PCR (P1, P2) است. LSO نيز در قسمت ‘5 توالي مكمل SNP را دارد. قسمت مياني LSO هدفي براي يكي از 1624 اليگونوكلئوتيد متصل به ذرات مينياتوري است كه در بالا ذكر شد. انتهاي ‘3 اوليگوي اختصاصي لوكوس نيز مكمل آغازگر P3 است. LSO در انتهاي ‘5 فسفريله شده است.
تكنيك Golden Gate براي بررسي همزمان بيش از 1536 لوكوس SNP كاربرد دارد. هر سه اوليگوي لازم براي هر يك از 1536 SNP همه با هم به يك نمونه DNA ژنوميك اضافه ميشود. فرايند دورگهسازي انجام ميشود. اوليگوهاي هيبريد نشده با شستشو حذف ميشوند. DNA پليمراز به واكنش فوق اضافه ميشود و افتراق آللي مستقیماً بر روي DNA متصل به فاز جامد صورت ميپذيرد. انتهاي ‘3 ASO كه مكمل باز موجود در ناحيه SNP است، به DNA ژنومي متصل شده و به كمك آنزيم DNA پليمراز، طويل شده و به سمت ‘5 اوليگوي LSO حركت ميكند، سپس آنزيم ليگاز به محلول فوق اضافه شده، ASO به LSO متصل ميشود و الگوي لازم براي PCR توليد ميشود. حال اين پرسش ممكن است پيش بيايد كه چرا LSO را طوري طراحي نميكنند تا دقیقاً بعد از ناحيۀ SNP قرار گيرد و نيازي به مرحله طويلسازي نباشد؟ جواب اين است كه با اين كار يك درجه آزادي بيشتر براي موقعيت LSO قائل ميشويم و در طراحي LSO آزادي عمل بيشتري داريم. اگر قرار بود LSO دقیقاً بعد از SNP قرار ميگرفت، در اين صورت يك انتخاب براي توالي LSO داشتيم و به احتمال زياد LSO حاصل بهدرستی كار نميكرد، زيرا مشكلاتي از قبيل ايجاد دورگههاي اشتباه، اتصال غیراختصاصی به ديگر نقاط ژنوم، ايجاد ساختمان سنجاقسري و تشكيل دايمرهاي داخلي و غيره ممكن بود بهوجود آيد.
آغازگرهاي P1 ,P2 ,P3 به محصول طويلشده، اضافه ميشود. آغازگر P1 و P2 با مواد فلورسان شامل Cy3 و Cy5 نشاندار شدهاند و در شکل فوق محصول P1-A-P3 تشكيل ميشود. اگر SNP بهصورت هتروزيگوت باشد، هم P1-A-P3 و هم P2-G-P3 تشكيل ميشوند. اگر ژنوتيپ نمونه بهصورت هموزيگوت G باشد، فقط P2-G-P3 تشكيل ميشود. آغازگرهای P1 ,P2 و P3، سيگنال هر 1536 SNP را بهطور خودبهخود تكثير ميكنند. محصولات فلورسنتۀ PCR تکرشتهای شده و با ماتريكس دورگه ميشوند. توالي نشاني[6] موجود بر روي LSO، براي هر لوكوس SNP منحصر به فرد است و با قسمت مكمل اوليگوي موجود بر روي ذرات آرايه دورگه ميشود. واكنشي كه در آن تعداد زيادي SNP بهطور همزمان بررسي شده بودند[7]، در اين مرحله از هم مجزا ميشوند[8] تا قابل خواندن باشند. قسمت پايين شكل فوق، تصويري از يك آرايه است كه با دو رنگ اسكن شده است. ذرات سفيد حاوي سيگنال Cy3 و ذرات خاكستري تيره داراي سيگنال Cy5 است. لكههاي خاكستري روشن نيز هر دو سيگنال Cy3 و Cy5 را دارد.
شکل 2: پلاتفورم Illuminaجهت تعیین ژنوتیپ
[1] Tag SNPs
[2] Site-specificity
[3] Allelic discrimination
[4] Allele specific oligo
[5] Locus-specific oligo
[6] Address
[7] Highly multiplexed
[8] De-multiplexed
بیولومینسانس و کاربرد آن در روشهای ایمونواسی
نقش ترانسپوزونها در پروکاریوتها (3)
مروری برفناوری ریزآرایه (Micro array)
برای دانلود فایل pdf بر روی لینک زیر کلیک کنید
ورود / ثبت نام