برتری استراتژیهای تکثیر HPV
بر اساس استفاده از نواحی E6/E7 نسبت به L1
نادیا شافعی، دکترای تخصصی پزشکی مولکولی، دانشگاه علوم پزشکی کردستان
شادی سایبان، کارشناسی ارشد ژنتیک پزشکی، دانشگاه علوم پزشکی شهید بهشتی
نکات کلیدی:
- E6/E7 جزو نواحی انکوژنیک ویروس است.
- پس از عفونت سلول با ویروس HPV، ناحیه E6 و E7 حفظ میشود در حالی که ناحیهی L1 و E2 حذف میشود.
- برخلاف توالی ناحیه L1، توالی E6 و E7 کاملاً حفاظت شده است.
- تشابه بالا و یکپارچگی ناحیه E6 در هر HPV عاملی است که ریسک عدم اتصال صحیح پرایمر را کاهش میدهد.
- توالی E6 و E7 بین گونههای مختلف پرخطر و کمخطر HPV متفاوت است، در نتیجه امکان تشخیص افتراقی را فراهم میکند.
- با توجه به تمام نکات گفته شده، مشخص میشود که استفاده از توالی E6 و E7 انتخاب بهینه برای تشخیص HPV میباشد.
یکی از علل اصلی سرطان سرویکس، عفونت با سابتایپهای پرخطر HPV میباشد. یکی از رایجترین روشها برای تشخیص عفونتهای ویروسی استفاده از روشهای مبتنی بر PCR است. با استفاده از تکنیک PCR، 100–500-bp تکثیر میشود در حالی که ژنوم HPV، 7.9 kb است و این بدان معنی است که نواحی بسیاری از ژنوم این ویروس میتواند بهعنوان کاندید تکثیر در نظر گرفته شوند؛ اما آنچه اهمیت دارد این است که تفاوت در ویژگیهای بیولوژیک و ویژگیهای توالی موجود در نواحی مختلف ژنوم میتواند تأثیر حیاتی بر بازده PCR داشته باشد. در مقاله حاضر به بررسی مزایا و معایب تکثیر توالیهای رایج در کیتهای موجود در بازار پرداخته شده است.
ویژگی فیزیکی DNAHPV
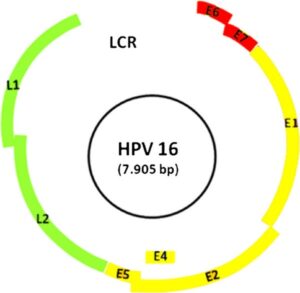
ژنوم HPV دارای یک ناحیه تنظیمی بالادست یا ناحیه کنترل LCR، یک ناحیه برای ژنهای بیان شوندهی اولیه(E1 ,E2 ,E6 and E7) و یک ناحیه برای ژنهای تأخیری (L1 and L2) میباشد. برخی از این نواحی با هم همپوشانی دارند؛ بهعنوان مثال ناحیه E6 که بلندتر از توالی ناحیه E7 میباشد در یک ناحیه قابل توجه با توالی E7 همپوشانی دارد. پس از عفونت سلولهای اپیتلیال سرویکس با ویروس، DNA حلقوی ویروس در سلول آزاد میشود.
در نمونههای کمخطر نظیر سابتایپهای 6 و 11، ژنوم ویروس در سلول بهصورت اپیزومال باقی میماند، این حالت در نمونههای بالینی CIN1، CIN2و گاه در CIN3 که دارای عفونت با سابتایپهای پرخطر هستند نیز مشاهده میشود. با این وجود در سلولهای سرطانی، اغلب مشاهده شده است که DNA ویروس در DNA سلول میزبان اینتگره شده است. دخول DNA ویروس در ژنوم میزبان عامل ترانسفورماسیون و سرطانی شدن سلول میباشد.
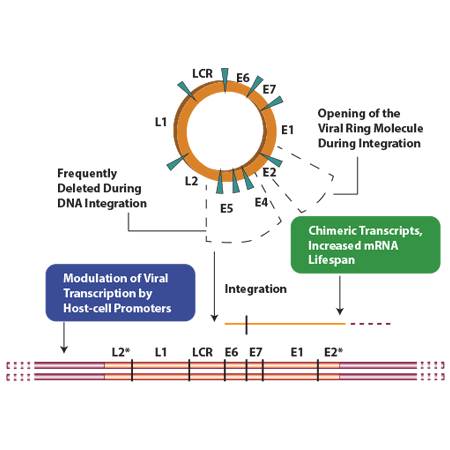
در طی فرآیند اینتگراسیون بخش بزرگی از ژنوم ویروس از جمله ناحیهی E2، E1، L1 و L2 حذف میشود. اهمیت این موضوع زمانی بیشتر مشخص میشود که هدف، غربالگری افراد در معرض خطر برای سرطان سرویکس است چرا که در این سلولها، فقط نواحی خاصی از ویروس که طی فرآیند اینتگراسیون حذف نشدهاند، قابل شناسایی است.
در مطالعات مختلف نشان داده شده است که اینتگره شدن و حذف توالیهای ذکرشده در HPV16 در 86% نمونهها مشاهده شده است. قابل ذکر است که HPV16 نیز یکی از رایجترین سابتایپها در ایجاد سرطان سرویکس است، بنابراین طراحی پرایمر برای تشخیص دقیق این ویروس و طراحی روشهایی با حساسیت بالا در تشخیص عفونت یکی از چالشهای اصلی در این ارتباط میباشد. در کیتهایی که از پرایمر L1 برای تشخیص HPV استفاده میکنند، نرخ نتایج مثبت بسیار کمتر از مقدار واقعی است و علت این نتایج منفی کاذب حذف توالی L1 طی فرآیند اینتگراسیون میباشد. از آنجایی که حذف توالیL1 نهتنها در فازهای تأخیری نئوپلازی بلکه در مراحل اولیه ایجاد ضایعههای سرطانی نیز دیده میشود، میتوان به این نتیجه رسید که احتمالاً این مرحله یکی از مراحل اصلی در سرطانزایی باشد.
علاوه بر این مطالعات اخیر نشان دادهاند که برخی سابتایپهای HPV نظیر 16، 18 و 45 بسیار بیشتر از سابتایپهای پرخطر 31 و 33 به حالت اینتگره درمیآیند.
نواحی ژنومیک دخیل در فرایند اینتگراسیون
اینتگراسیون شامل ایجاد شکست درDNA و حذف قطعهای از ژنوم ویروس میباشد. بر اساس مطالعات انجام شده بر روی لاین سلولی سرطان سرویکس، تغییراتی که در طی فرایند اینتگراسیون مشاهده شده است عبارتند از:
- حذف یک ناحیه 3-2 کیلوبازی که شامل ناحیهی E2 تا L2 است
- امکان شناسایی رونوشتهای E6–E7 در سلولهای آلوده
- بههمریختگی چارچوب خواندن کدونهای ترجمه در توالیهایE1 ,E2 ,E4 و E5 طی فرآیند اینتگراسیون در ژنوم میزبان
- با بررسیهایی که از طریق روش هیبریدیزاسیون با توالیهای RNA انجام شد، مشخص گردید که کل توالی E6 و E7 و بخشی از توالی E1 رونویسی شده است اما هیچ رونوشتی از نواحی L1 و L2 وجود ندارد که میتواند علت آن حذف این نواحی یا تغییر توالی آنها به گونهای باشد که امکان ایجاد رونوشت از آنها وجود نداشته باشد.
علت اینتگراسیون ژنوم ویروس HPV
اینتگراسیون HPV16 DNA منجر به افزایش پایداری mRNA کدکنندهی E6 و E7 میشود و طی این فرآیند توالیهای مربوطه از ناحیهی غنی از AT جداشده و به ناحیهای که غلظت کمتری از AT دارد متصل میشود و این تغییرات عامل افزایش بیان این پروتئینها در سلولهای سرطانی است.
جایگاههای موتاسیون در ژنوم HPV
یکی دیگر از نکاتی که در صورت استفاده از روش PCR در تشخیص HPV باید مدنظر قرار داد سایتهایی است که احتمال وقوع جهش در آنها بسیار زیاد است. یکی از این نواحی، L1 میباشد و این ویژگی بر کیفیت اتصال پرایمرها و نتیجهی PCR اثر مستقیم دارد.
با بررسیهایی که بر روی رونوشت E6/E7 انجام شده است، نشان داده شده است که توالی RNA دقیقاً با توالی ثبتشده در داده پایگاههای علمی نظیر NCBI همخوانی دارد، در حالی که در HPVهایی که توالی L1 وE1 در آنها حذف نشده است دارای انواع مختلف موتاسیونها اعم از موتاسیــــــــونهای Transversion،transition ,small deletion ,small insertion میباشد.
برخی از این جهشها عبارتند از:
- حذف سر `5 توالی L1 بهصورتی که DNA باقی مانده از نوکلئوتید 6334 شروع شود.
- حذف یک نوکلئوتید در ناحیه 6387
- دخول توالی CAT در موقعیت 6901
- حذف GAT در موقعیت 6949
با توجه به این اطلاعات در ارتباط با روش شناسایی HPV کدام روش را باید انتخاب کنیم؟
مزیت هدف قرار دادن توالیهایE1 و L1 توانایی آنها در شناسایی بسیاری و حتی تمام سابتایپهای HPV میباشد و عیب اصلی این نواحی این است که این توالیها به احتمال بسیار بالا طی فرآیند اینتگراسیون یا حذف میشوند یا تغییرات ساختاری اساسی پیدا میکنند، از طرف دیگر هدف قرار دادن نواحی E6 و E7 تمام نمونههایی که با سابتایپهای پرخطر آلوده شدهاند و ژنوم آنها در ژنوم میزبان اینتگره شده است را شناسایی میکند و در نتیجه استفاده از پرایمر برای این ناحیه باعث کاهش نرخ نتایج منفی کاذب میشود. استفاده از تکثیر توالی L1 در PCR با مشکلات اینتگراسیون و وقوع جهشهای مختلف در این ناحیه همراه است، در نتیجه بسیاری از خانمهای آلوده به HPV به دلیل عدم شناسایی از پروسه درمان حذف میشوند. در حالی که این اتفاق به احتمال بسیار بسیار پایینتری در روشهایی که E6/E7 را هدف قرار دادهاند، رخ خواهد داد چرا که این نواحی جزو نواحی بسیار حفاظتشده در ژنوم ویروس است و هرگونه جهش و تغییر در این توالی باعث تغییرات اساسی در فعالیت ویروس از جمله فعالیت انکوژنیک خواهد شد.
مشکل دیگر استفاده از توالی L1 به کار بردن پرایمرهای اختصاصی متعدد است.
تمام موارد ذکرشده، نشاندهندهی مزیت استفاده از پرایمر برای توالی E6/E7 نسبت به بقیهی توالیهاست. جهت تأیید بر ادعاهای ذکرشده، نتایج چند مطالعه در این ارتباط بررسی میشود.
در مطالعهای که بر روی سرطان آنال انجام شده است نشان داده شد که نرخ تشخیص عفونت HPV با استفاده از پرایمر L1، 16% و نرخ تشخیص در روش مبتنی بر استفاده از پرایمر E6 برابر 46% بود.
روش مبتنی بر استفاده از پرایمر L1 اکثر نمونههای آلوده با سابتایپهای 6 و 11 را شناسایی میکند اما نمونههای 16 و 18 را کمتر از تعداد نمونههای واقعی شناسایی میکند. در مطالعهی دیگری نشان داده شده است که از 56 نمونه در 23 نمونه حذف توالی L1 (41%) اتفاق افتاده بود.
در مطالعهای دیگر عملکرد پرایمرهای E6/E7 باMY09/11 که از قطعهی L1 است مقایسه شد و نتایج دو روش 92% با هم همخوانی داشت اما یکی از نمونهها که در روشMY09/11 منفی شده بود با روش E6/E7 مشخص شد که نمونه مثبت است.
در مطالعهای دیگر نیز نشان داده شد که در مقایسه بین روش مبتنی بر استفاده از پرایمر E7 نسبت به استفاده از پرایمرهای تغییریافتهی L1 (Nested primers (MY11/GP6q and GP5q/ GP6q حساسیت و نرخ تشخیص بالاتری دارد.
Reference:
Morris BJ. Cervical human papillomavirus screening by PCR: advantages of targeting the E6/E7 region. Clinical Chemistry and Laboratory Medicine (CCLM). 2005 Nov 1;43(11):1171-7.
Okodo M, Okayama K, Teruya K, Sasagawa T. Uniplex E6/E7 PCR method detecting E6 or E7 genes in 39 human papillomavirus types. Journal of medical virology.2018 Jan 4.
Arbyn M, Snijders PJ, Meijer CJ, Berkhof J, Cuschieri K, Kocjan BJ, Poljak M. Which high-risk HPV assays fulfil criteria for use in primary cervical cancer screening?.Clinical Microbiology and Infection. 2015 Sep 1;21(9):817-26.
Michelli E, Téllez L, Mendoza JA, Jürgensen C, Muñoz M, Pérez S, Mosqueda N, Hernández E, Noguera ME, Callejas D, Correnti M. Comparative analysis of three methods for HPV DNA detection in cervical samples. Investigacionclinica. 2011;52(4).
Shikova E, Todorova I, Ganchev G, Kouseva-Dragneva V. Detection and typing of human papillomaviruses by PCR. Biotechnology & Biotechnological Equipment. 2009 Jan 1;23(sup1):877-80.
ارتباط سرطان سر و گردن با ویروس پاپیلوما
ژنومیکس و کاربرد آن در تشخیص بیماریها
برای دانلود فایل pdf بر روی لینک زیر کلیک کنید
ورود / ثبت نام