بکارگیری تکنیک تکثير دایرهای چرخان (RCA)
برای شناسایی گونه قارچی پاتوژن
دکتر صادق خداویسی1، دکتر سهیلا محمودپور2، شهرام محمودی3، زهرا صالحی4، دکتر حمید بدلی5
1: دکتری تخصصی قارچشناسی پزشکی، گروه انگلشناسی و قارچشناسی، دانشگاه علوم پزشکی کردستان
2: دکتری علوم آزمایشگاهی، آزمایشگاه تشخیص پزشکی پاستور، سنندج
3: دانشجوی دکتری تخصصی قارچشناسی پزشکی، گروه انگلشناسی و قارچشناسی، دانشگاه علوم پزشکی تهران
4: دانشجوی دکتری تخصصی قارچشناسی پزشکی، گروه قارچشناسی پزشکی، دانشگاه تربیت مدرس
5: دانشیار، گروه انگلشناسی و قارچشناسی پزشکی، مرکز تحقیقات قارچهای تهاجمی، دانشگاه علوم پزشکی مازندران
مقدمه:
عفونتهای قارچی تهاجمی عمدتاً افراد با نقص سیستم ایمنی را مبتلا کرده و با وجود پیشرفتهای حاصل شده در زمینهی تشخیص و درمان، عامل مرگ و میر بسیاری از بیماران مستعد میباشند (1). وضعیت سیستم ایمنی فرد در پیشرفت و بروز این عفونتها نقش مهمی دارد. هرچند که بهطور قابلتوجهی وضعیت سیستم ایمنی فرد تعیینکننده روند بیماری است، اما شروع درمان ضدقارچی مؤثر، در کنترل عفونت بسیار مهم است (2). با پیشرفتهای حاصلشده در علم ژنتيک، راهکارهایی در پيش روي محققين قرار گرفته است که توسط آن توانستهاند قدمهای بسيار بلندي در امر تسريع کنترل کيفي و تشخيص بيماري بردارند. يکي از تحولات عظيم در بحث ژنتيک و بهتبع آن تشخيص طبي، معرفی روش تعیین توالی میباشد. خوشبختانه با ايجاد پايگاه دادههای ژني و پروتئيني، محققين به روشهایی دست یافتهاند که به کمک علم نوپاي بيوانفورماتيک میتوان روشهایی بسيار دقيق، سريع و اختصاصیتر از تکنیکهای متداول مولكولي كه علاوه بر گرانقیمت بودن نيازمند نیروی متخصص نيز هستند را در علوم تشخيصي ارائه نمایند؛ يكي از روشهایی كه امروزه توسط محققين بسيار موردتوجه قرار گرفته است روشهای ايزوترمال است. در اين روش بدون نياز به اعمال تناوب دمايي، تكثير DNA امکانپذیر میشود. از جمله اين تکنیکها، تکثير دایرهای چرخان (RCA) میباشد كه به دلیل اختصاصيت فوقالعاده بالای آن بسيار موردتوجه قرار گرفته است. در این تکنیک از يك پروب بسته براي شناسايي منطقه خاصي از ژنوم استفاده میشود (3). به دلیل اینکه در این تکنیک يك قالب بسته در اتصال به هدف ايجاد میشود، امكان تكثير توالي غیراختصاصی را نزديك به صفر میرساند. تکنيک مولکولي RCA بهتازگی توسط محققين جهت رفع نواقص ذکرشده براي واکنشهای زنجیرهای پليمراز ارائه شده است. تکنیک RCA از ویژگی خارقالعادهای برای شناسایی توالیهای خاص DNA یا RNA و همچنین برای نشانگرهای مولکولی غیر از DNA یا RNA برخوردار است، در نتیجه امکان تعیین چندین گونه بهصورت همزمان و نیز شناسایی جهشهای تکنوکلئوتیدی و آنتیژنهای خاص را فراهم میآورد. تکنیک RCA یک روش فوق حساس برای شناسایی است؛ بهگونهای که انواعی از فرمتهای RCA، امکان شمارش تکمولکولی DNA، RNA، اهداف پروتئینی و برخی آنالیتهای دیگر را فراهم میآورد. علاوه بر این، تشخیص براساس RCA بهعنوان روشی با تکرارپذیری خوب شناخته شده و در مقایسه با PCR، اشتباهات تکثیری در سطح پایینتری رخ میدهد (3, 4). در نتیجه چنین حساسیت بالایی امکان تعیین کمی تعداد کپیهای یک ژن و نیز شناسایی ژنهای تککپی، افتراق کمپلکسهای آنتیژن- آنتیبادی و سطح بیان mRNA در سلولهای منفرد را فراهم میآورد. در اين مقاله کاربرد تکنیک RCA جهت شناسایی سریع و دقیق گونههای قارچی در قارچهای پاتوژن مهم توضیح داده خواهد شد.
اساس روش RCA:
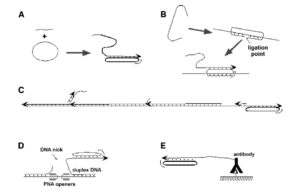
تکثیر دایرهای چرخان (RCA)، واکنشی ایزوترمال برای سنتز آنزیماتیک صدها تا میلیاردها کپی خطی از پروبهای DNA حلقوی کوچک و تکرشتهای میباشد. تکرارهای طولانی بهدستآمده از توالی DNA ممکن است بهعنوان یک تقویتکننده سیگنال برای تشخیص با حساسیت بالای اسیدهای نوکلئیک خاص و دیگر مولکولهای بیولوژیکی مهم در تشخیص ژنتیک و پروتئومیکس باشد. به دلیل کارآیی بالا و سهولت استفاده، تستهای مبنی بر RCA، در میان روشهای تشخیص مولکولی جایگاه خاصی را در مقایسه با سایر روشهای تکثیر تکدمایی به خود اختصاص دادند (3). اساس RCA بر تکثیر چرخان DNA تکرشتهای کوتاه حلقوی با DNA پلیمراز خاص در درجه حرارت ثابت است که در اواسط دهه 1990 کشف شد (5). این واکنش در ابتدا با هیبریداسیون DNA خطی تکرشتهای به DNA حلقوی کوچک خاص شروع و بهطور گسترده برای اهداف تشخیصی در تشخیص مستقیم یا غیرمستقیم DNA و RNAهای مختلف، پروتئین و سایر بیومارکرها بکار برده شد که سابق بر این بدین منظور از روشهای مختلف شناسایی بیومولکولها استفاده میشد (14). واکنش RCA شامل سیکلهای متعددی از سنتز آنزیمی همدما است که پرایمر بهوسیله DNA پلیمراز بهطور دایرهوار هیبرید میشود و این پروسه بهطور مداوم حول DNA حلقوی پروب (چند دهنوکلئوتیدی) بارها و بارها تکرار شده و ادامه مییابد (شکل 1). این یک فرایند کینتیک خطی است که بهراحتی در یک ساعت تا چند هزار توالی مکمل پشت سر هم[1] از یک DNA اصلی (الگو) حلقوی کوچک سنتز میشود. این محصولات تکثیری معمولاً دارای توزیع وسیعی از نظر طول بوده و در تصاویر الکتروفورز بر روی ژل بهصورت اسمیر پهنی از DNAهای با وزن مولکولی بالا دیده میشوند (شکل 2). بهطورکلی، تشخیص مبتنی بر RCA را میتوان به دو گروه طبقهبندی کرد؛ برخی از آنها با پروب حلقوی انجام میشوند (شکل 1، D و E)، درحالیکه دیگر روشها شامل حلقوی شدن پروبهای خطی هیبریدشده از طریق اتصال است که متعاقب RCA صورت میگیرد (شکل 1، B).
شکل 1: شکل شماتیک از فرآیندهای RCA
نوک پیکان نماد DNA پلیمراز است. DNA کوچک حلقوی با سایز کمتر از 100 نوکلئوتید و قطعات مولکول بسیار پایدار dsDNA با این سایز در RCA استفاده شده، فقط قسمتی از پروب حلقوی میتواند در هر زمان جفت شود، در نتیجه شکل هندسی کمپلکسهای حاصل شده در RCA مشابه کمان است.
(A) واکنش RCA بر روی DNA کوچک حلقوی آزاد و با استفاده از یک پرایمر انجام میشود. اگر هدف استفادهشده برای شروع واکنش RCA، مولکول DNA باشد، محصولات تکثیر بهطور ثابتی به مولکولهای هدف متصل میشوند (رجوع شود به شکل شماتیک D). برای هدفهای متصلشده به سطح، این محصولات بر روی فاز جامد ثابت میشوند.
(B) تشخیص تکثیر پروب بر اساس RCA در محل حلقوی شدن پروب الیگونوکلئوتید خطی و یک پرایمر هدف غیرمرتبط ( L-RCAیا اتصال-RCA). در برخی از موارد، ارتباط توپولوژیک بین DNA حلقوی کوچک و محل DNA هدف یا مارکر ممکن است در تکثیر حلقوی تأثیرگذار باشد. پروب حلقوی برای ادامه هیبریدیزیشن باید از DNA هدف جدا شود.
(C) مراحل اولیه double-primed RCA. در این واکنش، پرایمر دوم که مکمل محصول اصلی RCA است استفاده شده است. در اینجا، حضور DNA پلیمراز برای سنتز رشته جایگزین ضروری است.
(D). پیشرفت در واکنش RCA برای تولید dsDNA با همکاری شروعکنندههای PNA و شکاف DNA انجام میشود.
(E) در immuno-RCA، انتهای ¢5 پرایمر به یک ریپورتر آنتیبادی متصل میشود که بصورت انتخابی به یک آنالیت ثابتشده بر یک سطح جامد متصل میشود.
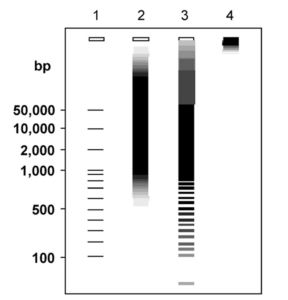
شکل 2: الگوی محصول RCA بدستآمده با کینتیک خطی یا هندسی پس از تفکیک با الکتروفورز در ژل
معمولاً تکثیر خطی با امپلیکونهای شبهاسمیر مشخص میشود که نشانگر توزیع پیوسته محصولات تکرشتهای RCA بر اساس طول است (خط 2). برخلاف آن امپلیکونهای شبهنردبان[2] معمولاً به دنبال تکثیر هندسی مشاهده میشوند و نشانگر کپیهای دو رشتهای کنکاتمریک یک الگوی حلقوی است که بهطور معمول بعد از تکثیر خطی کپیهای دو رشتهای از یک الگوی حلقوی بدست میآیند (خط 3). در برخی از موارد، اغلب محصولات RCA آنقدر بزرگ هستند که نمیتوانند روی ژل حرکت کنند (خط 4). خط 1 مربوط به سایز مارکر میباشد.
بکارگیری RCA در شناسایی گونه:
با توجه به کاهش حساسیت طیف وسیعی از پاتوژنهای قارچی به داروهای ضدقارچی، شناسایی سریع آنها در حد گونه برای مدیریت بالینی بیماران واجد اهمیت است. با وجود این، روشهای شناسایی استاندارد مبتنی بر کشت از حساسیت و سرعت پایینی برخوردارند (6)، بنابراین روشهای مولکولی با ویژگی، تکرارپذیری و حساسیت مناسب موردنیاز میباشند، لذا امروزه ابزارها و روشهای مبتنی بر PCR بهویژه با استفاده از ناحیههای ITS، ITS1 و ITS2 از کمپـــلکس ژنی ریبوزومال DNA در روشهای مختلفی مانند Multiplex PCR، Real-time PCR، آنالیزهای توالی DNA و تکنیکهای مبتنی بر پروب شامل طیفی از روشها از ساترنبلاتینگ و هیبریداسیون ریورسلاینبلات [3](RLB) نویدبخش اهدافی مناسب جهت شناسایی گونه هستند (7, 8). در میان تعداد فراوان روشهای مولکولی که برای شناسایی گونهی قارچهای پاتوژن (مخصوصاً کاندیدا و آسپرژیلوس) تعریف شده است، آنالیز توالی ناحیه ITS بهعنوان روش استاندارد طلایی مطرح است، بااینوجود تعیین توالی، هزینهبر و زمانبر بوده و برای تعداد بالای ایزولهها غیرقابل استفاده است. روشهای مبتنی بر پروب مدتهاست که بهعنوان گزینههایی جهت ردیابی طیف وسیعی از گونهها مطرح میباشند. فرمتهای جدید و حساس RLB و میکرواری امکان ردیابی چند پاتوژن را در یک تست فراهم میکند (9, 10). درحالیکه این روشها نیازمند تجهیزات بسیار پیشرفته بوده و در واقع قابلاستفاده در سطح آزمایشگاههای بالینی نیستند، RCA تکنیکی بر پایهی تکثیر چرخان حلقه DNA تکرشتهای کوتاه با DNA پلیمرازهای خاص تحت شرایط ایزوترمال است که موارد بالا را تحقق میبخشد (11). مزیتهای روش مبتنی بر RCA شامل ویژگی و انعطافپذیری آن است که پروبها قابل دستکاری بوده و میتوان آنها را برای اهداف مختلف بالینی مورد استفاده قرار داد. کاربرد این تکنیک در شناسایی پلیمورفیسم تکنوکلئوتیدی در گونههای مختلف نشاندهنده این است که روش RCA یک روش قابلاجرا بوده و با استفاده از آن میتوان پلیمورفیسمهای تکنوکلئوتیدی گونههای نزدیک به هم را افتراق داد (12). در مطالعه Zhou و همکاران، 10 عدد podlock پروب برای ناحیهی ITS2 (6 پروب اختصاصی برای هر یک از گونههای کاندیدا، 1 پروب اختصاصی برای آسپرژیلوس فومیگاتوس، 1 پروب اختصاصی برای آسپرژیلوس فلاووس، 1 پروب اختصاصی سدوسپوریوم آپیوسپرموم و 1 پروب نیز اختصاصی سدوسپوریوم پرولیفیکنس) طراحی شد (12). در این مطالعه جهت ایدهآل کردن اتصال به DNA، دمای ذوب انتهای ´5 بازوی متصلشونده به پروب، بالاتر از دمای استفادهشده برای اتصال پروب (°62 سانتیگراد) درنظر گرفته شد. بهمنظور افزایش ویژگی، بازوی متصلشونده به انتهای ´3 بهنحوی طراحی شد که دارای دمای ذوب 50 تا 57 درجه سانتیگراد (کمتر از دمای اتصال) باشد. بعلاوه ناحیهی اتصالی هر پروب طوری طراحی شد که کمترین تشابه را با سایر پاتوژنهای مشابه داشته باشد تا امکان اتصال پرایمر طی RCA فراهم شود. یافتههای این مطالعه نشان داد که پروبهای اختصاصی گونهها بهدرستی، همهی 6 گونه کاندیدای مطالعه شده شامل کاندیدا دابلینسیس و کاندیدا گیلرموندی را شناسایی کردند. علاوه بر این افتراق واضحی بین گونههای آسپرژیلوس فومیگاتوس و آسپرژیلوس فلاووس و نیز گونههای اپیوسپرموم و پرولیفیکنس از جنس سدوسپوریوم دیده شد. در کل، این مطالعه توانایی بالای این روش ساده، سریع (2 ساعت) و خیلی اختصاصی مبتنی بر RCA را برای شناسایی گونههای پاتوژن قارچی (کاندیدا، آسپرژیلوس و سدوسپوریوم) نشان داد (12). همچنین Hamzehei و همکاران در مطالعه خود از روش RCA جهت شناسایی سریع گونههای کلادوفیالوفورا که از عوامل بالقوه عفونت انسانی میباشند، استفاده کردند (13). این مطالعه نشان داد که به دلیل میزان بالای تشابه مورفولوژیک موجود میان گونههای تازه توصیفشدهی کلادوفیالوفورا، وجود مشکلاتی در شناسایی آنها امری بدیهی است. روشهای مرسوم موجود در آزمایشگاههای تشخیصی که برای شناسایی در حد گونه بکار میروند معمولاً بر مبنای اصول مورفولوژی و فیزیولوژی استوار بوده و نیازمند صرف چندین روز یا هفته زمان میباشند. همچنین این روشها اکثراً غیراختصاصی بوده، لذا در این مطالعه با استفاده از روش حساس RCA مبتنی بر پروبهای Podlock اختصاصی گونه طراحیشده برای ناحیه ITS از DNA ریبوزومال نشان دادند این پروبها بهدرستی تمام 10 گونهی کلادوفیالوفورا را شناسایی کرده و هیچگونه واکنش ناخواستهای مشاهده نشد (13). سادگی، حساسیت، پتانسیل بالا و قیمت پایین تکنیک RCA جایگاه خاصی را برای آن در میان روشهای ایزوترمال شناسایی گونهها مبتنی بر DNA مطرح میکند. همچنین در مطالعه Javaheri Tehrani و همکاران نیز پیشنهاد کردند که استفاده از روش RCA در شناسایی سریع گونههای قارچی بسیار آسان، ارزان و اختصاصی است (14)، هرچند که مطالعههای بیشتر با استفاده از طیف وسیعی از گونههای قارچی در راستای ارزیابی نقش این سیستم در شناسایی روتین عوامل قارچی میتواند کمککننده باشد.
منابع:
- Baddley JW, Stroud TP, Salzman D, Pappas PG. Invasive mold infections in allogeneic bone marrow transplant recipients. Clin Infect Dis 2001; 32(9): 1319-1324.
- Badali H, Khodavaisy S, Davoudi MM, Biranvand E, Mardani M. Antifungal Therapy for Invasive Fungal Infections. JMUMS. 2014; 24(114): 187-205.
- Demidov VV. Rolling-circle amplification (RCA). Encyclopedia of Diagnostic Genomics and Proteomics 2005: 1175-9.
- Daubendiek SL, Ryan K, Kool ET. Rolling-circle RNA synthesis: circular oligonucleotides as efficient substrates for T7 RNA polymerase. JACS 1995; 117(29): 7818-9.
- Fire A, Xu S-Q. Rolling replication of short DNA circles. PNAS 1995; 92(10): 4641-5.
- Reiss E, Obayashi T, Orle K, Yoshida M, Zancope-Oliveira R. Non-culture based diagnostic tests for mycotic infections. Med Mycol 2000; 38(1): 147-59.
- Ciardo D, Schär G, Böttger E, Altwegg M, Bosshard P. Internal transcribed spacer sequencing versus biochemical profiling for identification of medically important yeasts. J Clin Microbiol 2006; 44(1):77-84.
- Leaw SN, Chang HC, Sun HF, Barton R, Bouchara J-P, Chang TC. Identification of medically important yeast species by sequence analysis of the internal transcribed spacer regions. J Clin Microbiol 2006; 44(3): 693-9.
- Hsiao CR, Huang L, Bouchara J-P, Barton R, Li HC, Chang TC. Identification of medically important molds by an oligonucleotide array. J Clin Microbiol 2005; 43(8): 3760-8.
- Zeng X, Kong F, Halliday C, Chen S, Lau A, Playford G, et al. Reverse line blot hybridization assay for identification of medically important fungi from culture and clinical specimens. J Clin Microbiol 2007; 45(9): 2872-80.
- Nilsson M. Lock and roll: single-molecule genotyping in situ using padlock probes and rolling-circle amplification. Histochemistry Cell Biol 2006; 126(2): 159-64.
- Zhou X, Kong F, Sorrell TC, Wang H, Duan Y, Chen SC. Practical method for detection and identification of Candida, Aspergillus, and Scedosporium spp. by use of rolling-circle amplification. J Clin Microbiol 2008; 46(7): 2423-7.
- Hamzehei H, Yazdanparast SA, Davoudi MM, Khodavaisy S, Golehkheyli M, Ansari S, et al. Use of rolling circle amplification to rapidly identify species of Cladophialophora potentially causing human infection. Mycopathologia 2013; 175(5-6): 431-8.
- Javaheri Tehrani S, Aliabadian M, Fata A, Najafzadeh MJ. Rolling Circle Amplification (RCA): an approach for quick detection and identification of fungal species. Journal of Mycology Research 2014; 1(1): 55-62.
[1] Tandem Repeats
[2] – ladder
[3] Reverse Line Blot
تشخیص سریع عوامل میکروبی با بیوسنسورها
برای دانلود فایل pdf بر روی لینک زیر کلیک کنید
ورود / ثبت نام