تعیین الگوی پلاسمیدی باکتریهای گرم منفی عامل عفونتهای بیمارستانی
- زهرا رخشیدن، کارشناس ارشد ژنتیک دانشگاه علوم پزشکی اردبیل، شبکه بهداشت و درمان شهرستان مشگین شهر
- عباس حسینزاده ایواتلو، کارشناس علوم آزمایشگاهی دانشگاه علوم پزشکی اردبیل، شبکه بهداشت و درمان شهرستان مشگین شهر
- فاطمه فرجی مزرعه خلف، کارشناس علوم آزمایشگاهی دانشگاه علوم پزشکی اردبیل، شبکه بهداشت و درمان شهرستان مشگین شهر
- سپیده اصول دینی، کارشناس علوم آزمایشگاهی دانشگاه علوم پزشکی اردبیل، شبکه بهداشت و درمان شهرستان مشگین شهر
- مهناز نوبخت مللو، کارشناس علوم آزمایشگاهی دانشگاه علوم پزشکی اردبیل، شبکه بهداشت و درمان شهرستان مشگین شهر
- ناهید افقهی نجف، کارشناس علوم آزمایشگاهی دانشگاه علوم پزشکی اردبیل، شبکه بهداشت و درمان شهرستان مشگین شهر
- احمد گلمحمدی دوکش، کاردان علوم آزمایشگاهی دانشگاه علوم پزشکی اردبیل، مرکز آموزشی و درمانی امام خمینی (ره) اردبیل
مقدمه
خصوصیات فنوتیپی باکتریها نظیر الگوهای بیوشیمیایی، باکتریوفاژ تایپینگ، وجود آنتیژنهای سطحی سلول و الگوهای حساسیت به عوامل ضدمیکروبی بر اساس تغییرات در فاز رشد و موتاسیون خودبخودی، تمایل به تغییر دارند، بنابراین استفاده از این خصوصیات برای شناسایی منبع عفونت و شیوع آن برای بررسی عفونتهای ایجادشده توسط گونههای هتروژن باکتریهای گرم منفی مفید نخواهد بود، لذا استفاده از متدهای مولکولی نظیر آنالیز الگوی پلاسمیدی در مطالعات اپیدمیولوژیک ضروری است.
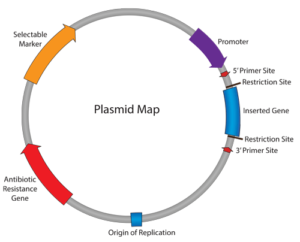
پلاسمیدها مولکولهای DNA دورشتهای مستقل خارج کروموزومی هستند که وجود آنها برای بقای سلول ضروری نیست اما ممکن است تنوع وسیعی از خصوصیات ژنتیکی را کد کنند که میزبان را برای رقابت بهتر با سایر میکروارگانیسمهای اشغالکننده همان مکان اکولوژیک مستعد بکند. یکی از خصوصیاتی که پلاسمیدها را در میکروبشناسی با اهمیت خاص جلوهگر میسازد، توانایی آنها در حمل و انتقال ژنهای کدکننده مقاومت به ترکیبات ضدمیکروبی است. در آنالیز الگوی پلاسمیدی سویههای باکتری، تعداد و اندازه پلاسميدها اساس تشخيص سويه میباشد و سویههای منشأ گرفته از يك كلون داراي تعداد مشابه از پلاسميدها بوده و اندازه آنها نيز مشابه خواهد بود (1).
شکل 1: ساختمان کلی یک پلاسمید باکتریایی
1- استخراج و خالصسازی پلاسمید از باکتریها
1- 1- مواد و محلولهای لازم برای استخراج پلاسمید از باکتریها با روش لیز قلیایی
- محیط کشت LB (Luria Bertaini) حاوی آمپیسیلین
- محلول شماره یک (بافر لیزکننده) حاوی گلوکز 50 میلیمول و 10Mm EDTA (PH=8) و 25Mm Tris-HCl
- محلول شماره 2 (محلول دناتوره کننده) حاوی 2 N NaOH و 3% SDS
- محلول شماره 3 (محلول استات پتاسیم) حاوی 120 میلیلیتر استات پتاسیم 5 مولار و 23 میلیلیتر اسید استیک گلاسیال و 57 میلیلیتر آب مقطر
- بافر TE حاوی 10Mm Tris-Hcl و 1Mm EDTA
- RNase A با غلظت 10 میلیگرم بر میلیلیتر
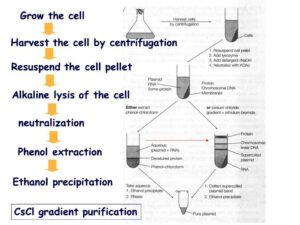
1-2- خلاصهای از مراحل استخراج و خالصسازی DNA پلاسمیدی از باکتریها
- یک کلنی منفرد و خالص از کشت جوان باکتری را به محیط کشت LB حاوی آنتیبیوتیک تلقیح کرده و به مدت 20 ساعت در 37 درجه سانتیگراد انکوبه میکنیم.
- 1/5 میلیلیتر از کشت باکتریایی را به میکروتیوب منتقل کرده و به مدت 4 دقیقه در دور 4000 سانتریفوژ میکنیم.
- 0/2 میلیلیتر از محلول شماره 1 که به مدت 1 دقیقه در فریزر 20- درجه سانتیگراد نگهداری شده بود در وضعیت فوقالعاده سرد بر روی رسوب سلولی ریخته میشود و سوسپانسیون سلول به مدت 5 دقیقه در حرارت آزمایشگاه انکوبه میشود.
- 0/4 میلیلیتر محلول شماره 2 تازه تهیهشده اضافه شده و سر لوله بسته میشود و 6 بار بهآرامی سروته شده و به مدت 5 دقیقه در آب یخ انکوبه میگردد.
- 0/3 میلیلیتر از محلول شماره 3 که به مدت 20 دقیقه در فریزر در 20- درجه سانتیگراد نگهداری شده بود در وضعیت فوقالعاده سرد اضافه گشته و 6 بار بهآرامی سروته شده و به مدت 5 دقیقه در آب یخ انکوبه میشود.
- لوله به مدت 5 دقیقه در دور 12000 در 4 درجه سانتیگراد سانتریفوژ شده و پس از سانتریفوژ کردن 750 میکرولیتر از مایع رویی به میکروتیوب جدید اضافه میگردد.
- برروی 750 میکرولیتر از مایع رویی 1/5 میکرولیتر از RNase A با غلظت 10 میلیگرم بر میلیلیتر افزوده شده و به مدت 20 دقیقه در 37 درجه انکوبه میگردد.
- بعد از اتمام انکوباسیون و خنک کردن میکروتیوبها در حرارت آزمایشگاه، 750 میکرولیتر از محلول فنل متعادل شده/ کلروفرم/ ایزوآمیل الکل (25/24/1) اضافه شده و به مدت 1 دقیقه ورتکس شده سپس به مدت 4 دقیقه در دور 12000 در 4 درجه سانتیگراد سانتریفوژ میگردد.
- مایع رویی به میکروتیوب جدید منتقل شده و بر روی آن 750 میکرولیتر اتانل 96 درجه که به مدت 10 دقیقه در فریزر 20- درجه سانتیگراد نگهداری شده بود در وضعیت فوقالعاده سرد اضافه میشود و 6 بار بهآرامی سروته شده و بعد به مدت 2 ساعت در 20- درجه سانتیگراد انکوبه میشود.
- سپس لوله به مدت 15 دقیقه با دور 12000 در 4 درجه سانتریفوژ میشود. مایع رویی دور ریخته شده و رسوب سفیدرنگ در ته لوله باقی میماند. میکروتیوب به بهطور معکوس بر روی دستمال کاغذی برای خشک شدن رسوب قرار داده میشود.
- 1 میلیلیتر اتانل 70% که به مدت 10 دقیقه در 4 درجه نگهداری شده بود در وضعیت فوقالعاده سرد اضافه شده و سر لوله بسته شده و با چند بار سروته کردن مخلوط میگردد.
- سپس لوله به مدت 1 دقیقه با دور 12000 در 4 درجه سانتریفوژ میشود. مایع رویی دور ریخته شده و میکروتیوب به بهطور معکوس بر روی دستمال کاغذی برای خشک شدن رسوب قرار داده میشود.
- به مدت 5 تا 10 دقیقه سر میکروتیوب به شکل باز در محیط آزمایشگاه قرار داده میشود تا اتانل تبخیر شود و سپس 50 میکرولیتر از بافر TE بر روی رسوب اضافه شده و رسوب در آن حل گشته و در مواقع موردنیاز برای جمع شدن TE در ته لوله از سانتریفوژ استفاده میشود.
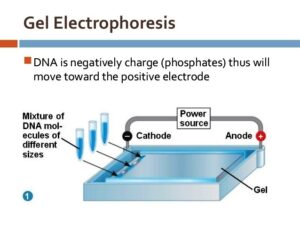
1-3- الکتروفورز DNA پلاسمیدی در روی ژل آگارز
- سینی ژل شسته شده و خشک گردیده و دو انتهای آن با چسب اتوکلاو بسته میشود.
- شانه ژل در سینی قرار داده شده بطوریکه شانه حدود 1 میلیلیتر بالاتر از سطح سینی قرار گیرد و سپس ژل بر روی میز بهصورت تراز قرار داده میشود.
- بهمنظور تهیه آگارز 0/7درصد، 140 میلیگرم پودر آگارز در ارلن مایر ریخته شده و بر روی آن 20 میلیلیتر بافر الکتروفورز 5 TBE اضافه میشود و در ارلن مایر با درپوش شیشهای پوشانده شده و سپس در میکروویو به مدت 20 ثانیه قرار داده میشود تا آگارز حل شود.
- محلول آگارز تا 60 درجه سانتیگراد خنک شده و سپس در سینی ژل ریخته میشود، طوری که هیچ حبابی مخصوصاً زیر شانهها نباشد و ضخامت ژل حدود 4 میلیلیتر باشد.
- بعد از اینکه ژل کاملاً بسته شد، بهدقت قالبهای کناری ژل و شانه برداشته شده و سینی به همراه ژل در تانک الکتروفورز قرار داده میشود، طوری که چاهکهای نمونه نزدیک قطب منفی قرار میگیرند.
- تانک الکتروفورز با 450 میلیلیتر بافر الکتروفورز 5 TBE پر میشود. مقدار بافر بهاندازهای باید باشد که سطح ژل به عمق حداقل 1 میلیمتر پوشانده شود.
- مقدار 5 میکرولیتر از محلول DNA پلاسمیدی با 1 میکرولیتر از بافر لودکنــــــــــــــــنده ژل (Gel loading buffer) حاوی 0/25 درصد بروموفنول بلو و 0/25 درصد گزیلن سیانول و 30 درصد گلیسرول در آب مخلوط شده و در چاهک موجود در ژل قرار داده میشود.
- سرپوش تانک الکتروفورز گذاشته شده و توسط سیمهای ارتباطی به Power supply متصل گردیده و جریان الکتریکی برقرار میگردد، بطوریکه ولتاژ مورداستفاده 80 ولت بوده و الکتروفورز تا مدت 1/5 ساعت ادامه خواهد یافت.
- بعد از پایان کار جریان الکتریکی قطع شده و ژل برداشته شده و به مدت 30 دقیقه در داخل آب مقطر حاوی 0/5 میکروگرم بر میلیلیتر اتیدیم بروماید جهت رنگآمیزی قرار داده میشود.
- پس از رنگآمیزی با استفاده از نور اولتراویوله حاصل از ترانس لومیناتور با طولموج 300 نانومتر موردبررسی قرار گرفته و از سایز مارکر لامبدا DNA برای بررسی اندازه قطعات استفاده میشود.
- پس از اتمام کار و تهیه عکس از ژل برای تخریب اتیدیم بروماید که سرطانزا میباشد، ژل به مدت 5 دقیقه در داخل محلول پرمنگنات پتاسیم قرار داده شده و سپس دور ریخته میشود (5-2).
شکل 2: خلاصهای از استخراج و خالصسازی پلاسمید با روش لیز قلیایی از باکتریها
4- تشریح مکانیسم خالصسازی پلاسمید با روش لیز قلیایی
- در روش لیز قلیایی برای اینکه بتوانیم مقدار زیادی از DNA را بدست آوریم بایستی باکتری را در محیط کشت حاوی آنتیبیوتیک کشت دهیم و نیز باید یک سوم لوله از محیط مایع پر شود تا مقدار کافی از اکسیژن در محیط وجود داشته باشد. همچنین محیطهای کشت به مدت 24 ساعت در 37 درجه سانتیگراد انکوبه میشوند تا مقدار کافی از سلول باکتریایی برای استخراج DNA پلاسمیدی وجود داشته باشد.
- برای آمادهسازی باکتریها بهمنظور لیز شدن، باکتریها در بافر حاوی Tris- Hcl، گلوکز و EDTA حل میشوند. Tris یا هیدروکسی متیل آمینومتان، pk اپتیمم را برای نگهداری DNA دارد و PH را بهطور ثابت حفظ میکند. EDTA با عوامل شلاته کننده نظیر +Mg2 که برای عمل آنزیم DNases موردنیاز هستند ترکیب شده و این عوامل را غیرفعال میکند. گلوکز برای بالا بردن فشار اسمزی بیرون سلول است.
- در این روش برای لیز کردن باکتریها از محلول SDS و NaOH استفاده میشود. SDS یک دترجنت است که چربیهای غشاء سلولی را حل میکند و سود عامل قلیایی است که دو رشته DNA را از هم جدا میکند.
- اضافه کردن استات پتاسیم PH محلول را به خنثی نزدیک میکند و در این مرحله DNA مجدداً دورشتهای میشود. قطعات DNA کروموزومی بهصورت تصادفی با هم جفت میشوند و بیشتر به حالت دناتوره باقی میمانند و همراه با پروتئینهای سلولی در داخل کمپلکسهای بزرگ گیر افتاده و با دودسیلسولفات پوشیده شده و از طریق سانتریفیوژ رسوب میکنند ولی DNAهای پلاسمیدی به خاطر داشتن اندازه کوچک و ساختار سوپرکویل بهصورت متصل به هم باقی میمانند و مجدداً بهصورت دورشتهای درمیآیند و ساختار اولیه را بدست میآورند و پس از سانتریفوژ کردن در مرحله رویی باقی میمانند.
شکل 3: اساس الکتروفورز در ژل آگارز
5- توصیههای مهم و احتیاطات لازم
- ورتکس کردن یا تکان دادن شدید سوسپانسیون سلولی، DNA کروموزمی را تکهتکه میکند و از سلول آزاد میکند. برای جلوگیری از این کار سلولها باید بهصورت خیلی آرام و با استفاده از سرسمپلر در بافر حل شوند.
- اگر مرحله لیز سلولی به مدت طولانی باشد و همچنین لیز سلولی بهشدت انجام گیرد باز DNA کروموزمی محصولات پلاسمیدی را آلوده خواهد کرد، لذا برای از بین بردن این مشکل زمان انکوباسیون در یخ بعد از اضافه کردن محلول لیزکننده درست به مدت 5 دقیقه باید انجام گیرد و از لیزوزیم برای لیز کردن سلول استفاده نشود.
- نگهداری SDS در حرارت کمتر از 20 درجه سانتیگراد باعث کریستالیزه شدن آن میگردد در نتیجه مقدار SDS موجود در محلول برای رسوب دادن کمپلکسهای سلولی کافی نخواهد بود.
- اگر بعد از اضافه کردن استات پتاسیم، مخلوط کردن سریعتر انجام نگیرد تودههای پتاسیم دودسیل سولفات تشکیل نخواهند شد. این تودهها همراه با کمپلکسهای DNA سلولی هستند و به شناور بودن تمایل دارند. برای رسوب دادن این تودههای شناور باید دور و زمان سانتریفوژ افزایش یافته و عمل سانتریفیوژ در 4 درجه سانتیگراد انجام گیرد (6).
6- نگاهی اجمالی به نتایج آنالیز الگوی پلاسمیدی چند باسیل گرم منفی در مطالعات انجامشده در ایران و سایر کشورها
- سودوموناس ائروجینوزا (Pseudomonas aeruginosa)
1- طبق مطالعه انجام شده در دانشگاه علوم پزشکی تهران توسط دکتر بادامی و همکاران DNA پلاسمیدی در 62 درصد از کل سویههای مورد مطالعه نشان داده شده است. تعداد پلاسمیدها نیز بین 1 تا 4 عدد و اکثراً 3 عدد بوده است (7).
2- طبق مطالعه دکتر نهایی و همکاران در دانشگاه علوم پزشکی تبریز در سال 2005، DNA پلاسمیدی در 49/6 درصد از کل سویههای مورد مطالعه نشان داده شده است. تعداد پلاسمیدها نیز از 1 تا 6 عدد و وزن مولکولی پلاسمیدها در محدوده 0/5 کیلو جفت باز تا 21 کیلوجفت باز بوده است (8).
3- طبق مطالعه Millesimo و همکاران در ایتالیا در سال 1996، DNA پلاسمیدی در 45/2 درصد از کل سویههای مورد مطالعه موجود بوده و وزن مولکولی پلاسمیدها از 1 کیلو جفت باز تا 23 کیلو جفت باز متغیر بوده و نیز تعداد پلاسمیدها بین 1 تا 3 عدد بوده است (9).
4- در یک مطالعه انجام شده در سال 2004 در ترکیه توسط AÇik و همکاران، DNA پلاسمیدی در 7/5 درصد از کل سویههای مورد مطالعه نشان داده شده و وزن مولکولی پلاسمیدها بین 1/2 تا 4/2کیلو جفت باز بوده و تعداد پلاسمیدها از 1 تا 4 عدد گزارش شده است (10).
- اشریشیا کولی (Escherichia coli)
1- در سال 1392 طبق مطالعه انجام شده توسط نورا میرمظفری ثابت و همکاران، 78/3 درصد از سویههای اشریشیا کولی دارای DNA پلاسمیدی بودهاند. تعداد پلاسمیدها از 1 تا 8 عدد متغیر بود و وزن مولکولی آنها در محدوده 5/2 کیلو جفت باز تا 10 کیلو جفت باز متغیر بوده است. (11).
2- در سال 1382 طبق مطالعه انجام شده توسط دکتر صادقی و همکاران، DNA پلاسمیدی در 90 درصد از کل سویههای مورد مطالعه نشان داده شده است. وزن مولکولی پلاسمیدها در محدوده 1 تا 21 متغیر و تعداد پلاسمیدها بین 1 تا 7 عدد گزارش شده است (12).
3- در سال 2009 طبق مطالعه انجام شده توسط Babu Uma و همکاران در هندوستان، DNA پلاسمیدی در 64 درصد از کل سویههای مورد مطالعه جداشده از نمونههای اسهالی نشان داده شده است. وزن مولکولی آنها در محدوده 1 تا 25 بوده است (13).
4- در سال 2014 طبق مطالعه انجام شده توسط O Akingbade و همکاران در نیجریه، DNA پلاسمیدی در 5/37 درصد از کل سویههای مورد مطالعه جداشده از عفونتهای ادراری نشان داده شده است (14).
- کلبسیلا پنومونیه (Klebsieela pneumonia)
1- بر طبق مطالعه انجام شده توسط Olusola A. Akingbade و همکاران در سال 2013 در نیجریه، DNA پلاسمیدی در 37/03 از کل سویهها مشاهده شده است (15).
2- در مطالعه انجام شده توسط دکتر میرداود عمرانی و همکاران در سال 2008 در ارومیه، DNA پلاسمیدی در 25/6 درصد از کل سویهها مشاهده شده است (16).
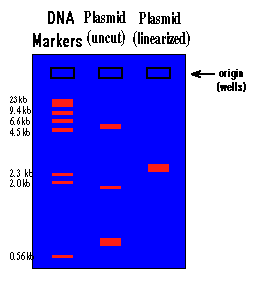
شکل 4: طرز خوانش الگوی پلاسمیدی یک باکتری
نتیجهگیری
الگوی پلاسمیدی متد مولکولی اولیه برای تعیین نوع سویههای باکتریایی است. تعداد و اندازه پلاسمیدهای موجود بهعنوان پایهای برای شناسایی سویه باکتریایی است و استفاده از الگوی پلاسمیدی در بررسی شیوعها قابل اطمینان میباشد (17). وجود الگوهای پلاسمیدی مشابه در سویههای جداشده از عفونتهای بیمارستانی در یک محدوده زمانی کوتاه نشان میدهد که احتمالاً این سویهها از یک کلون باکتریایی منشأ گرفتهاند و یا انتقال ژن در بین سویههای بیمارستانی با شیوع بالایی مطرح میباشد.
References:
- Foxman B, Rilery L: Molecular Epidemiology: Focus on infections. Am J Epidemiol, 153:1135-1141,2001.
- Nahaie MR, Goodfloow M, Harwood CR: A rapid screening procedure for Staphylococcal plasmids. J Microbial Meth, 2:73-81, 1984.
- Sambrook J, Russel DW: Molecular Cloning. A laboratory manual, volume 1, third ed.CSHL press, New York, 1.1-134& 5.1-5.17, 2001.
- Satisvan: Pasmid DNA isolation. Molecular Biology Protocols. N.d. Availabl from yahoo.com. (http://www.esb.utexas.edu, Accessed July 2003.
- Birmboim HC: A rapid alkaline extraction method for the isolation of plsmid DNA. Methods Enzymol, 100:243-255, 1983.
- Vnden HJP: General molecular biology techniques in: PCR protocols in molecular toxicology. CRC press LLC, 177-221, 1998.
- Abiri R and etal: Plasmid profiling of Pseudomonas aeruginosa isolated from burn infections in hospitalized patients in Shaheed Motahhari Burn and Accident Centre of Tehran. Burns, 29: 547-551, 2003.
- Nahaei M.R and etal: Antibiotic resistance and plasmid profiles of Pseudomonas aeruginosa strains isolated from in-patients of Sina Hospital- Tabriz. J of Ardebil University of .Medical Sciences (JAUMS) , 7(1): (23):90-98, 2007.
9.Millesimo and etal: Pseudomonas aeruginosa clinical isolates serotypes, resistance phenotypes and plasmid profiles. Euro J Epidemiol, 12:1123-9, 1996.
- AÇik L and etal: Antibiotic susceptibility, plasmidprofiles and RAPD PCR analysis of c Pseudomonas aeruginosa linical isolated in Turkey. Available from w3.. gazi. Edu.tr/leyacik/rpd.htm, 2005.
- Amir Mozaffasri Sabet N and etal: Antibiotic resistance and plasmid profiles of Escherichia coli isolated from Karaj City Patients. New Cellular and Molecular Biotechnology Journal:3(12), 2012.
- Sadeghi J and etal: Antibiotic resistance and plasmid profiles of Escherichia coli isolated from urinary tract infections from outpatient and in patient in Imam Khomeini Educational and Treatment Centre. MEDICAL JOURNAL OF TABRIZ UNIVERSITY OF MEDICAL SCIENCES, 27(2): 51-58, 2005.
13.Babu Uma and etal: Antibiotic Sensitivity and Plasmid Profiles of Escherichia coli Isolated from Pediatric Diarrhea. J Glob Infect Dis, 1(2): 107–110: 2009.
14. O Akingbade and etal: Resistant plasmid profile analysis of multidrug resistant Escherichia coli isolated from urinary tract infections in Abeokuta, Nigeria. African Health Sciences , 14 (4), 2014 .
- Olusola A. Akingbade and etal: Plasmid Profile of Isolated Klebsiella Species in a Tertiary Hospital in Abeokuta, Ogun State, Nigeria. World Applied Sciences Journal , 21 (3): 371-378, 2013.
- mir davood omrani and etal: Plasmid Profiling of Klebsiella Species and its relation with antibiotic resistance at two hospitals of Urmia(Iran). Journal of Applied Sciences, 8(15):2781-2784, 2008.
- Sulgajic v and etal: Reliability of plasmid profile analysis in the identification of epidemic strains of bacteria cousing an outbreak of intestinal infections. Vojnosanti pregl, 58:615-20, 2001.
استخراج و خالصسازی DNA پلاسمیدی از سودوموناس آئروجینوزا
نکات مهم کاربردی در میکروبشناسی بالینی (3)
جداسازی و تخلیص اسیدهای نوکلئیک
برای دانلود فایل pdf بر روی لینک زیر کلیک کنید
ورود / ثبت نام