ویژگیها و کاربردهای آپتامرها
پروین زمانی1
1.دانشجوی دکتری، گروه بیوتکنولوژی پزشکی، دانشگاه علوم پزشکی مشهد، مشهد، ایران
چکیده:
آپتامرها الیگونوکلئوتیدهای تکرشتهای یا پپتیدهای کوچکی هستند که با اختصاصیت و تمایل بالا به مولکولهای هدف متصل میشوند. تولید این لیگاندهای مصنوعی شامل تولید کتابخانههایی از نوکلئیک اسیدهای سنتتیک، جذب و تکثیر آنها با یک فرایند غنیسازی تصاعدی (SELEX) است. در سالهای اخیر آپتامرها به عنوان جایگزینی برای آنتیبادیها در نظر گرفته شدهاند؛ به این دلیل که نه تنها ویژگیهای آنتیبادیها را دارند بلکه خصوصیات دیگری مانند قدرت ایمنیزایی کمتر، پایداری دمایی بیشتر و هزینه کمتر برای ساخت دارند.
آپتامرها به دلیل ویژگیها و ساختار شیمیایی منحصر به فرد خود کاربردهای زیادی در تکنیکهای مختلف مانند الیزا، بیوسنسورها و… دارند. به طور خلاصه میتوان گفت که آپتامرها موادی هستند که در زمینههای گوناگون کاربرد دارند و میتوانند به عنوان جایگزینی برای آنتیبادیها و هم به عنوان جزء اصلی تجهیزات پزشکی در تشخیص و درمان درنظر گرفته شوند.
کلید واژه: آپتامر، آپتامرهای اسید نوکلئیکی، آپتامرهای پپتیدی، SELEX
مقدمه:
شناسایی مولکولها نقش مهمی را در طبیعت بازی میکند. انواع مختلفی از جفت مولکولهای زیستی مانند آنتیژنها و آنتیبادیها، هورمونها و گیرندههای هورمونها، پروموتر DNA و فاکتورهای نسخهبرداری، آنزیم و سوبسترا در طبیعت وجود دارد (1). اکثر این جفت مولکولهای زیستی دارای تمایل و اختصاصیت بالایی هستند و به طور وسیعی مورد مطالعه قرار گرفتهاند. اکثر این مولکولهای زیستی کاربرد زیادی از جمله در فرایندهای بیولوژیکی، درمانی و حسگرهای زیستی دارند، با این حال چالشهای زیادی در مورد استفاده از این نوع سیستمهای طبیعی در برخی شرایط آزمایشگاهی وجود دارد، به عنوان مثال بسیاری از مولکولهای زیستی (مانند آنزیمها و آنتیبادیها) ساختار عملکردی ناپایداری دارند و وقتی تحت شرایط ایدهآلی قرار میگیرند به آسانی دناتوره میشوند و عملکرد بیولوژیکی خود را از دست میدهند.
به دلیل این محدودیتها کاربرد این جفت مولکولهای زیستی در بسیاری از شرایط محدود شده است، بنابراین تلاش برای تولید لیگاندهای سنتتیک بیشتر شد، لیگاندهایی که نه تنها تمایل[1] اتصال و اختصاصیت بالایی نسبت به مولکول هدف دارند بلکه ویژگیهای خاصی مانند تحمل شرایط سخت محیط بیولوژیکی و شیمیایی را نیز دارند، بنابراین در پاسخ به این نوع نیازها آپتامرها تولید شدند (1). در این مقاله، در مورد ساختار و فرایند تولید آپتامرها و ویژگیهای آپتامرها صحبت میشود. علاوه بر این چند نوع از فرایندهای انتخاب آپتامرها به روش In vitro شرح داده میشود. همچنین تعدادی از کابردهای آپتامرها در زمینههای مختلف برای تشخیص و درمان و شناسایی مولکولها مورد بحث قرار میگیرد.
آپتامرها
آپتامرها الیگونوکلئوتیدهای ریبونوکلئیک اسید (RNA) و داکسیریبونوکلئیک اسید تکرشته (ssDNA) یا مولکولهای پپتیدی کوچکی هستند که به دلیل ساختار سهبعدی اختصاصی که دارند میتوانند با اختصاصیت و تمایل بالا به مولکول هدف خود متصل میشوند. RNA آپتامرها و DNA آپتامرها توالی و الگوی تاخوردگی متفاوتی نسبت به یکدیگر دارند اما میتوانند به یک نوع مولکول هدف متصل شوند (2).
مفهوم توانایی اتصال یک نوکلئیک اسید به پروتئین در ابتدا با مطالعاتی که در سال 1980 بر روی آدنوویروسها و ویروس HIV[2] انجام میشد، بیان شد. در این مطالعات نشان داده شد که این ویروسها تعدادی RNA ساختاری کوچک را کد میکنند که با تمایل و اختصاصیت بالا میتواند به پروتئینهای ویروسی متصل شود (3)، به عنوان مثال در مورد ویروس HIV، RNA کوچکی که TAR[3] نامیده میشود، میتواند با اتصال به پروتئین Tat ویروسی سبب افزایش نسخهبرداری شود (4). مطالعات بعدی روی آپتامر با توسعه تکنیک SELEX[4] بیشتر شد. این تکنیک اولین بار توسط گروه Gold و Szostak در سال 1990 گزارش شد و در حال حاضر پایه و اساس جداسازی آپتامرها است (5).
آپتامرهای نوکلئیک اسیدی
ویژگیهای آپتامر
به طور کلی آپتامرهای اسید نوکلئیکی طولی بین 100- 80 نوکلئوتید دارند، اما همه این نوکلئوتیدها در آپتامر نقش یکسانی را در اتصال به مولکول هدف ندارند. یک آپتامر با طول کامل معمولاً دارای سه بخش است؛ اول ناحیهای که در اتصال مستقیم به مولکول هدف نقش دارد و از 10- 15 نوکلئوتید تشکیل شده و در واقع ناحیه اتصالی است. ناحیه دیگر حاوی نوکلئوتیدهایی است که به طور مستقیم به مولکول هدف متصل نمیشود اما نقش مهمی در اتصال و برهمکنش ناحیه اتصالی با مولکول هدف دارند. نوکلئوتیدهایی که به طور مستقیم به مولکول هدف متصل میشوند یا اتصال آپتامر را به مولکول هدف تسهیل میکنند برای عملکرد آپتامرها ضروری هستند. به طور کلی تعداد این نوکلئوتیدهای ضروری بین 40-25 نوکلئوتید است (6).
سومین ناحیه حاوی نوکلئوتیدهایی است که نه به مولکول هدف متصل میشوند و نه اتصال آپتامر را به مولکول هدف تسهیل میکنند. این نوکلئوتیدها به عنوان نوکلئوتیدهای غیرضروری در نظر گرفته میشوند، بنابراین میتوان بعد از مرحله انتخاب آپتامرها این ناحیه اضافی را حذف کرد. حذف این ناحیه چندین اهمیت دارد، اول اینکه با حذف این ناحیه عملکرد اتصالی پرایمر بالا میرود. دوم، آپتامرهای کوچکتر تحرک بیشتری دارند در نتیجه امکان ساخت نانوساختارهای جدید که نیاز به وسایل حسگر بسیار حساس و کوچک دارند بیشتر میشود. سوم، سنتز آپتامرهایی با طول بیش از 60 نوکلئوتید فرایندی سخت و پرهزینه است (7).
مشخصات ساختاری آپتامرها
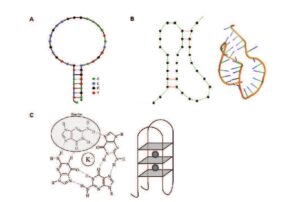
یکی از ویژگیهای متمایز آپتامرها تشکیل ساختارهای دوبعدی است. این ساختارها نتیجه ترکیبی از برهمکنش بین حلقههای آروماتیک بازها، گروههای قند و گروه فسفات است و شامل پیوندهای هیدروژنی، برهمکنشهای الکتروستاتیک و واندروالس میباشد (1). از فراوانترین ساختارهای دو بعدی که در آپتامرهای اسید نوکلئیکی دیده میشود میتوان به ساختارهای سنجاقسری[5] اشاره کرد که یکی از مشهورترین موتیفهای ساختاری است که هم در DNA آپتامرها و هم در RNA آپتامرها دیده میشود (1).
ساختارهای سودونات[6] در نتیجه برهمکنشهای مکملی بین توالیهای موجود در دنباله توالی ساختار سنجاقسری با قسمت حلقه آن تشکیل میشود. ساختارهای سودونات از مشخصههای ساختاری RNA آپتامرها هستند اما در DNA آپتامرها نیز وجود دارند (1).
ساختارهای کوآدروپلکس[7] در نتیجه اتصالات عرضی بین چهار باز گوانین موجود در توالی اسید نوکلئیکی به وجود میآید. هر باز گوانین در این ساختار با دو باز مجاور خود پیوند هیدروژنی برقرار میکند (شکل 1).
شکل 1: ساختارهای پایه آپتامرها. A: ساختار سنجاقسری، B: ساختار سودونات، C: ساختار کوادروپلکس (1)
اتصال یک آپتامر به مولکول هدف بسیار اختصاصی است. این برهمکنش آنقدر اختصاصی است که حتی تغییر کوچکی در مولکول هدف از اتصال آپتامر به آن جلوگیری میکند (8). علاوه بر این اختصاصیت، آپتامرها با تمایل بالا به مولکول هدف متصل میشوند، برای ماکرومولکولها (مانند پروتئینها) دارای ثابت تفکیکی (Kd)[8] از پیکومولار تا نانومولار هستند و برای مولکولهای کوچک ثابت تفکیک در حد نانومولار تا میلیمولار هستند (8).
طیف وسیعی از مولکولهای هدف برای آپتامرها وجود دارد که شامل یونهای فلزی، مولکولهای آلی کوچک، نوکلئوتیدها و مشتقات آنها، کوفاکتورها، نوکلئیک اسیدها، آمینواسیدها، کربوهیدراتها، آنتیبیوتیکها، پپتیدها، پروتئینها، سلول کامل و سلولهای آلوده به ویروس و باکتریها هستند (1).
مزیتها آپتامرها در مقایسه با آنتیبادیها
آپتامرها به عنوان جایگزینی برای آنتیبادیها در نظر گرفته میشوند، به این دلیل که در مقایسه با آنتیبادیهای مونوکلونال آپتامرها ویژگیهای بهتری دارند.
- پایداری بالای آپتامرها: همان طور که میدانیم پروتئینها تحت دماهای بالا به راحتی دناتوره میشوند و ساختار سه بعدی و عملکرد خود را از دست میدهند، در حالی که الیگونوکلئوتیدها پایداری دمایی بالایی دارند و در طی چرخههای تکراری دناتوراسیون و رناتوراسیون ساختار خود را حفظ میکنند، از این رو بزرگترین مزیت آپتامرهای الیگونوکلئوتیدی نسبت به آنتیبادیهایی با پایه پروتئینی در این است که در دماهای بالا پایدار هستند. آپتامرها بعد از رآنلینگ[9] دوباره ساختار طبیعی خود را به دست میآورند در حالی که آنتیبادیها متحمل دناتوراسیون برگشتناپذیر میشوند (9).
- تولید آپتامرها: تولید آنتیبادیهای مونوکلونال یک فرایند زمانبر و هزینهبر است و نیاز به جداسازی تعداد زیادی کلونی دارد. از طرف دیگر برای استفاده کلینیکی از آنتیبادیها نیاز به تولید مقدار فراوانی از آنها در کشتهای سلولهای حیوانی داریم که به نوبه خود یک فرایند زمانبر و هزینهبر است، در حالی که آپتامرها را از طریق فرایندهای شیمیایی میتوان به مقدار زیاد سنتز کرد که این فرایندهای شیمیایی نسبت به کشت سلولهای حیوانی کمهزینهتر هستند (10و 11).
- ایمونوژنیسیته پایین آپتامرها: به نظر میرسد که آپتامرها ایمونوژنیسیته و سمیت کمتری داشته باشند به این دلیل که معمولاً نوکلئیک اسیدها توسط سیستم ایمنی به عنوان عوامل خارجی شناسایی نمیشوند (12).
- تنوع مولکولهای هدف: در مورد مولکولها و توکسینهایی که نمیتوانند سیستم ایمنی بدن را تحریک کنند تولید آنتیبادیها مشکل است، اما آپتامرها را میتوان به اندازه کافی تولید کرد و در برخی از لیگاندها که توسط آنتیبادی شناسایی نمیشوند مانند یونها و مولکولهای کوچک استفاده نمود (11).
- تغییرات آپتامرها: آپتامرها را میتوان به راحتی با افزودن گروههای مختلف تغییر داد؛ به این منظور که پایداری آنها را افزایش دهیم یا برای اهداف مختلف گروههای متفاوتی مانند ترکیبات فلورسنت یا رادیوایزوتوپ را به آنها متصل نماییم (13).
- جایگاه عملکرد آپتامرها: آپتامرها میتوانند هم در داخل و هم در خارج از سلول فعالیت داشته باشند اما آنتیبادیها تنها در خارج از سلول و یا برای پروتئینهای ترشحی یا متصل به سطح خارجی سلول کاربرد دارند. (1).
- سایز آپتامرها: آپتامرها سایز کوچکتری نسبت به آنتیبادیها دارند و پاکسازی آنها از بدن راحتتر است، بنابراین ورود آنها به بافتها و سلولهای مختلف راحتتر بوده و میتوان داروهای مختلفی را به آنها متصل کرد و به جایگاه خاصی فرستاد و در فرایندهای دارورسانی هدفمند از آنها استفاده کرد (14).
انتخاب آپتامرها
از زمانی که مفهوم آپتامر بیان شد تکنولوژی SELEX برای جداسازی آپتامرهای نوکلئیک اسیدی در سال 1991 بیان شد و انواع مختلفی از آپتامرها به وجود آمد که میتوانند با اختصاصیت بالا به انواع مختلفی از مولکولهای هدف متصل شوند (15).
SELEX فرایندی است که در آن آپتامرهای اختصاصی برای مولکول هدف از یک کتابخانه ترکیبی الیگونوکلئوتیدهای سنتتیک حاوی تعداد زیادی DNA تکرشتهای با عملکرد بسیار جدا میشوند که شامل یک چرخه تکرار شونده از جذب[10]، بازیافت [11]RNA/DNA متصل شده و تکثیر[12] است (14).
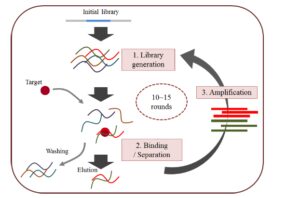
به طور کلی فرایند SELEX شامل سه مرحله است که به منظور انتخاب الیگونوکلئوتیدی که توانایی و تمایل بیشتری برای اتصال به مولکول هدف دارد تکرار میشود (16). شکل 2 نمایی کلی از فرایند SELEX را نشان میدهد (2). این سه فرایند شامل موارد زیر است:
1) تولید کتابخانه[13]، که کتابخانهای از الیگونوکلئوتید تکرشته تولید میشود. این الیگونوکلئوتید از یک ناحیه تصادفی[14] در قسمت مرکز، معمولاً بین 40-30 نوکلئوتید تشکیل شده و در دو انتهای آن جایگاه اتصال به پرایمر قرار دارد.
2) اتصال و جداسازی[15]، آپتامرها، اجزایی از کتابخانه که به مولکول هدف متصل شدهاند از اجزا متصل نشده جدا میشوند. مولکول هدف را میتوان به صورت ثابت و متصل شده به سطح مانند بیدهای مگنتیکی، ستون کروماتوگرافی و یا به صورت آزاد در محلول استفاده کرد. این مرحله یک مرحله بحرانی برای فرایند SELEX است. این مرحله با روشهای دیگری ترکیب شده و در نتیجه سبب شده است که انتخاب مولکول هدف و کتابخانه سادهتر و سریعتر شوند. 3) تکثیر، اجزایی از کتابخانه که به مولکول هدف متصل شده بودند با روش PCR تکثیر میشوند تا کتابخانه جدیدی حاصل شود و برای تکرار بعدی مورد استفاده قرار گیرد. آپتامرها از طریق این فرایند توسعه مییابند و سپس ویژگیهای آنها با روشهای بیولوژیکی متعددی شناسایی میشوند (2).
شکل 2: استراتژی کلی فرایند SELEX. با تشکیل یک کتابخانه ترکیبی (مرحله اول) آغاز میشود، با استفاده از یک فرایند تکرارشونده اتصال و جداسازی، شستشو (مرحله دوم) و تکثیر (مرحله سوم) ادامه مییابد (2)
انواع روشهای اتصال و جداسازی آپتامر
روش SELEX با استفاده از غشاهای نیتروسلولوزی[16]
غشاهای نیتروسلولوژی اغلب برای اتصال پروتئینها در روش وسترنبلات استفاده میشود. این روش برای جداسازی پروتئینها از سایر اجزا بکار میرود و برای اتصال پروتئینهایی که میتوانند با بیومولکولهای دیگری برهمکنش کنند توسعه یافت. به این دلیل که اغلب مولکولهای هدفی که در ابتدا برای SELEX استفاده میشد پروتئینی بودند استفاده از غشاهای نیتروسلولوزی در طی مراحل جداسازی مورد استفاده قرار گرفت. با این حال استفاده از این غشاها محدودیتهایی دارد؛ از جمله این که برای مولکولها و پپتیدهای کوچک قابل استفاده نیست و فرایندی طولانی مدت است و به طور کلی حداقل 12 راند نیاز دارد (17، 18).
SELEX بر اساس کروماتوگرافی تمایلی و بیدهای مگنتیکی[17]
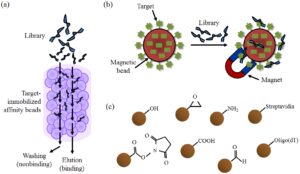
کروماتوگرافی تمایلی روشی است که برای جداسازی اجزا از یک مخلوط بیوشیمیایی مورد استفاده قرار میگیرد. عمدتاً برای خالصسازی پروتئینهای نوترکیب استفاده میشود و اساس آن بر پایه اتصال بیولوژیکی بسیار اختصاصی مانند برهمکنش بین لیگاند و رسپتور و یا یک آنتیژن و آنتیبادی است. فاز ثابت عمدتاً از بیدهای آگارزی تشکیل شده است و مولکولهای هدف روی بیدها ثابت میشوند، بیدها در داخل ستون قرار میگیرند و به جداسازی اجزاء کتابخانه که برای اتصال به مولکول هدف تمایل بالایی دارند استفاده میشوند.
در مورد تثبیت پروتئینها انواع مختلفی از تگها مانند گلوتاتیون s- ترانسفراز و his-tag مورد استفاده قرار میگیرد. در مورد مولکولهای آلی کوچک مولکول هدف از طریق پیوندهای شیمیایی به بیدها متصل میشود، بنابراین SELEX برای مولکولهای کوچک و پروتئینها را میتوان با این روش انجام داد. با این حال این روش معایبی نیز دارد؛ از جمله این که از این روش برای مولکولهایی که فاقد افینیتی تگ و گروههای شیمیایی برای اتصال به بیدها هستند نمیتوان استفاده کرد (شکل 3،a) (19).
از بیدهای مگنتیکی برای تثبیت مولکول هدف نیز استفاده میشود که از طریق یک برهمکنش یا یک واکنش شیمیایی بین افینیتی تگ و سوبسترا روی بیدها انجام میشود. استفاده از بیدهای مگنتیکی روشی سریع، آسان و قدرتمند برای جداسازی بیدهای حاوی مولکول هدف با استفاده از یک آهنربا است و به این دلیل که کتابخانههای متصل شده به مولکول هدف خیلی سریع و آسان از اجزاء متصل نشده تنها با استفاده از یک آهنربا جدا میشوند تلاشهای زیادی برای استفاده از این روش در جداسازی آپتامرها شده است (شکل 3،b) (20).
شکل 3: شماتیکی برای مرحله جداسازی از کتابخانه با استفاده از کروماتوگرافی تمایلی (a)، بیدهای مگنتیکی (b)، چندین نوع از گروههای عملکردی متصل شده به بیدها (c).
SELEX بر اساس کاپیلاری الکتروفورز[18]
این روش نسبت به روشهای دیگر مزایای زیادی از جمله سرعت، رزولوشن و توانایی بالایی دارد و حداقل مقدار نمونه رقیق شده استفاده میشود. با این روش یک آپتامر بر اساس تغییر حرکتش از بین مخلوطی از مولکولهای هدف، کتابخانه و کمپلکس مولکول هدف- کتابخانه جدا میشود. در این روش آپتامرها بر اساس بار و نیروی اصطکاکی[19] تحت یک میدان الکتریکی جدا میشوند. یکی از مزایای این روش این است که در مقایسه با روشهای دیگر تعداد راندهای کمی ( حدود 4-2 بار تکرار) نیاز دارد (21، 22).
SELEX نوری[20]
پایه روش SELEX نوری انتخاب آپتامرهایی با اختصاصیت و تمایل بالا است، با این ویژگی که آپتامر یک پیوند کوالانسی قابل القا با نور را با مولکول هدف تشکیل میدهد. این روش با جایگزینی یکی از بازهای نوکلئیک اسید کتابخانه با یک نوکلئوتید فعال شونده نوری[21] حاصل میشود. این نوکلئوتید باید قادر باشد وقتی در معرض نور قرار میگیرد یک پیوند کوالانسی را با مولکول هدف تشکیل دهد اما در عین حال باید پایدار باشد و در غیاب فعالسازی نوری[22] واکنشپذیر نباشد. تیمیدین معمولاً با 5 برمو-´2-داکسی یوریدین (BrDu)[23] جایگزین میشود، کروموفور برمویوراسیل نور UV را در محدوده 310 نانومتر جذب میکند در حالی که نوکلیئک اسید و پروتئین در این محدوده جذب ندارند (14).
با استفاده از این روش مولکول هدف، به عنوان مثال روی بیدهای آهنربایی تثبیت میشود و سپس در معرض کتابخانه الیگونوکلئوتیدی قرار میگیرد تا DNA/RNA به آن متصل شوند. در انتهای اولین مرحله انتخاب، بیدهای آهنربایی حامل کمپلکس مولکول هدف- آپتامر با استفاده از یک آهنربا از سیستم بازیافت میشوند و در معرض نور لیزر با طول موج 310 نانومتر قرار میگیرند تا فعالیت نوری را نشان دهند، در صورتی که الیگو نوکلئوتیدهای متصل نشده شسته شده و از سیستم خارج میشوند، سپس با استفاده از پروتئازها توالیهای متصل شده از مولکول هدف جدا شده و توسط واکنش PCR تکثیر میشوند و برای مرحله بعد مورد استفاده قرار میگیرند (23).
SELEX سلولی[24]
SELEX سلولی کمک میکند تا بتوان آپتامرهایی را بر علیه سلول کامل[25] پیدا کرد. در حالی که مولکولهای هدف اولیه برای روش SELEX پروتئینهای کاملاً خالص شده بودند. به عبارت دیگر مولکولهای هدف روش SELEX سلولی، پروتئینهای خارج سلولی روی سطح سلول یا ساختارهای خاصی از سلول بودند. در بیشتر موارد فرایند SELEX سلولی به این دلیل که تثبیت سلولی روی فاز جامد عملی نیست، فرایند شامل مراحل شستشو (برای سلولهای چسبنده) یا سانتریفیوژ (برای سوسپانسیون سلولی) در طی فرایند جداسازی آپتامرها است.
علاوه بر این شمارش سلولی در هر مرحله لازم است تا از انتخاب آپتامرهایی که به طور غیر اختصاصی سطوح سلولی را شناسایی کردند جلوگیری شود، بنابراین این روش تقریباً مشکلی است به این دلیل که تثبیت مولکولهای هدف غیرممکن است و نیاز به شمارش سلولی وجود دارد. با این حال آپتامرهای حاصل برای تشخیص سلولهای اختصاصی، دارورسانی هدفمند به سلولهای خاص و درمان سلولهای خاص بسیار مناسب بودهاند (1) (شکل 4(.
شکل 4: SELEX سلولی. (1) سلولهای هدف با کتابخانه اسید نوکلئیکی که شامل 1015 مولکول ssDNA است ترکیب میشوند. (2) سلولهای متصل شده به اسیدنوکلئیک از سلولهای متصل نشده جدا میشوند که میتوان از روشهای مختلفی مانند (2a) روش فلوسیتومتری (FACS)، (2b) سانتریفیوژ و (2c) روش شستشو استفاده کرد. (3) آپتامرها یا نوکلئیک اسیدهای متصل به سلول شسته میشوند و (4) توسط PCR تکثیر میشوند. قسمت انتهای ´5 پرایمر برگشت (reverse) حامل بیوتین است. علاوه بر این از یک پرایمر رفت که با فلورسنت نشانهگذاری شده است در فرایند PCR استفاده میشود و در نهایت آپتامرهایی که با اختصاصیت بالا به سطح سلول هدف متصل شدهاند به دست میآید (24)
آپتامرهای پپتیدی
آپتامرهای پپتیدی مولکولهای پروتئینی کوچکی هستند که دارای یک توالی پپتیدی متغیر میباشند که به یک پروتئین ساختاری[26] ثابت و خنثی متصل شدهاند و با تمایل بالایی به مولکول هدف متصل میشوند. این پپتیدها مولکولهای بسیار سادهای هستند که از یک کتابخانه ترکیبی بر اساس تمایل آنها به پروتئین هدف یا مولکولهای کوچک جدا میشوند و در سلولهایی باکتریایی مانند اشریشیاکولی[27] بیان میشوند. هر دو انتهایی این توالی متغیر به یک پروتئین ساختاری خنثی[28] متصل میشود، بنابراین آپتامرهای پپتیدی ساختارهای دوگانه بسته شدهای[29] هستند.
در این ساختارها ویژگیهای شناسایی مولکول هدف مشابه آنتیبادیها است اما دارای خصوصیات بهبودیافتهای هستند، به عنوان مثال سایز کوچک، پایداری بالا، حلالیت بالا، بیان بالا در باکتریها و امکان سنتز شیمیایی از ویژگیهای فولدینگ سریع میباشند. همچنین فاقد پیوندهای دیسولفیدی و سیستئین آزاد هستند که این ویژگی امکان بیان بالای آنها را در باکتریها ممکن میسازد و کاربردهای داخل سلولی آنها را بیشتر میکند (25).
شکل نهایی این ساختارهای مصنوعی هم توسط ترکیب آمینواسیدی و هم توالی پپتیدی و همچنین توسط توالی اولیه و ساختار سوم پروتئین ساختاری تعیین میشود (26).
انتخاب آپتامرهای پپتیدی
فرایند انتخاب آپتامرهای پپتیدی با فرایندهای استفاده شده برای آپتامرهای نوکلئیک اسیدی متفاوت است. اما با این حال اصول اصلی آن مشابه است که شامل استفاده از استراتژیهای متنوع برای تولید یک مجموعه از کتابخانهها، انتخاب و تکثیر است.
اولین مرحله، ساخت کتابخانهای از پپتیدها است. این کتابخانهها در مواردی مانند نوع پروتئین ساختاری، طول توالی پپتیدی، روش انتخاب و تعداد مارکرهای قابل انتخاب با هم تفاوت دارند. پروتئین ساختاری باید نسبتاً کوچک باشد، از یک زنجیره پلیپپتیدی تشکیل شده باشد و ساختار نسبتاً پایداری داشته باشد (27)، به عنوان مثال پروتئین TrxA باکتری اشریشیاکولی کاربرد گستردهای در توسعه آپتامرهای پپتیدی داشته است.
TrxA آنزیمی با جایگاه فعال کوچک است، سایز نسبتاً کوچکی دارد ( تقریباً kDa 12)، پایداری و حلالیت بالایی داشته و ساختار سه بعدی کاملاً شناخته شدهای دارد. از آن جایی که پروتئین ساختاری باید کاملاً خنثی باشد پپتیدها در داخل حلقه در مرکز جایگاه فعال قرار میگیرند، بنابراین فعالیت کاتالیتیکی آنزیم از بین میرود (25).
بعد از این که مولکول ساختاری انتخاب شد کتابخانه تولید میشود. این فرایند در سطح DNA است و به صورت تصادفی کدونها را در موقعیتهای آمینواسیدی تعیین میکند. به عبارت دیگر، در این روش کتابخانهای از ژنها ساخته میشود که میتواند به پروتئین ترجمه شود. تکنیکهای مختلفی برای تولید این کتابخانه ژنی وجود دارد و شرح آن خارج از بحث این مقاله میباشد.
روشهای مختلفی برای انتخاب آپتامرهای پپتیدی وجود دارد. به طور کلی این روشها را میتوان به سیستمهای غیردیسپلی[30]، سیستمهای دیسپلی با پایه سلولی[31] و سیستمهای دیسپلی مستقل از سلول[32] تقسیم کرد. در سیستمهای غیردیسپلی پروتئین همزمان با اجزای کتابخانه در محیط داخل سلولی بیان میشود و بنابراین انتخاب به وجود پروتئین هدف وابسته نیست اما با یک تاخوردگی صحیح در ارگانیسم میزبان تشکیل میشود. سیستم مخمر دو-هیبریدی (Y2H)[33] مثالی از این نوع تکنولوژی است (28).
فاژ دیسپلی یک تکنیک دیسپلی وابسته به سلول است. در این سیستم کتابخانه پپتید یا پروتئین به پروتئینهای پوششی ویروس متصل میشود (عمدتاً به پروتئین III) که در سطح ذره فاژی بیان میشود. سپس فاژ در تماس با مولکول هدف قرار میگیرد. بعد از انتخاب، فاژهای متصل نشده شسته میشوند و فاژهایی که به طور اختصاصی به مولکول هدف متصل شدهاند باقی میمانند. سپس این فاژها برای آلوده کردن سلولهای اشرشیاکولی جدید مورد استفاده قرار میگیرند تا کلونهای انتخاب شده تکثیر شوند (29، 30، 31). این کتابخانه فاژی جدید میتواند برای تکرار بعدی مورد استفاده قرار گیرد. ریبوزوم دیسپلی و mRNA دیسپلی مثالهایی از تکنیک دیسپلی مستقل از سلول هستند (32، 33).
کاربردهای آپتامرها
آپتامرها به عنوان مولکولهای زیستی در فرایندهای مختلفی مورد مطالعه قرار گرفتهاند، به عنوان مثال میتوان به کاربرد آنها به عنوان ابزارهای تشخیصی و درمانی، پروبهای حسگرهای زیستی، ایجاد داروهای جدید و سیستمهای دارورسانی هدفمند اشاره کرد.
درمان (داروهای جدید)
ماکوژن[34] یک آپتامر درمانی است که در واقع یک نوکلئوتید تکرشته و اختصاصی برای VEGF165 است که نقش مهمی را در رگزایی[35] و نفوذپذیری[36] در بیماری ماکولا دگرددیشن [37] وابسته به سن بازی میکند (2). این مورد در سال 2004 توسط FDA تأیید شد. این دارو یک آپتامر اختصاصی برای VEGF است. زمانی که این دارو بهVEGF متصل میشود برهمکنش VEGF با گیرندهاش مهار میشود (14)
سیستم دارورسانی
آپتامرهایی که توانایی اتصال به رسپتورهای سطح سلولی را دارند برای انتقال دارو به داخل سلول مورد بررسی قرار گرفتهاند، به عنوان مثال آنتیژنهای سطحی پروستات (PSMA)[38] یک مارکر مهم در سرطان پروستات است. یک آپتامر دوگانه حاوی آپتامر A10 و آپتامر DUP-1 ساخته شده است. آپتامر A10 برای سلولهای سرطانی PMSA+ و آپتامر DUP-1 برای سلولهای سرطانی PMSA– است. داروی دوکسوروبیسین[39] به آپتامر A10 اتصال دارد. بنابراین با اتصال آپتامر به سطح سلول دارو میتواند به داخل سلول تحویل داده شود (34).
تصویربرداری زیستی[40]
کاربرد دیگر آپتامرها به عنوان عواملی برای تصویربرداری هستند؛ به این صورت که با اتصال یک فلوروفور[41] یا مواد دیگری مانند گادولینیوم[42] برای تصویربرداری رزونانس مگنتیکی (MRI)[43] مناسب هستند. استفاد از آپتامرها به عنوان عوامل تصویربرداری مزایایی دارد؛ به عنوان مثال غیرسمی هستند به این دلیل که الیگونوکلئوتیدها سمی نیستند. علاوه بر این آپتامرها برای مولکول هدف خود بسیار اختصاصی هستند بنابراین هدفیابی صحیح و سریعی دارند و به راحتی از جریان خون عبور میکنند. با استفاده از این روش قطعیت بسیاری از نتایج به دست آمده طی تشخیص یا آنالیز کلینیکی افزایش مییابد (2).
بحث
همانطور که ذکر شد آپتامرها میتوانند به عنوان جایگزینی برای آنتیبادیها درنظر گرفته شوند، به دلیل این که آنها تحت برخی شرایط سخت پایدار هستند. چون آپتامرها بیشتر الیگونوکلئوتیدی هستند میتوان آنها را به مقدار زیاد و با خلوص بالا سنتز کرد و میتوان با مولکولهای مختلفی طی یک واکنش شیمیایی ساده آنها را تغییر داد. علاوه براین آپتامرها غیرسمی و غیر ایمونوژنیک هستند و به همین دلیل میتوان برای کاربردهای درمانی و تشخیصی از آنها استفاده کرد. با توجه به این که انواع مختلفی از مولکولهای هدف وجود دارد روشهای متنوعی از فرایند SELEX ابداع شده است تا انتخاب آپتامرها برای مولکول هدف موردنظر سریع و راحت باشد.
آپتامرها میتوانند کاربردهای زیادی در پزشکی مولکولی داشته باشند. با استفاده از آپتامرهایی که سلولها را شناسایی میکنند میتوانیم بدون اینکه اطلاعاتی از مولکولهای هدف داشته باشیم انواع مختلفی از سلولها را شناسایی کنیم. با ورود تکنولوژیهای دیگر مانند نانوتکنولوژی و بیوتکنولوژی، آپتامرها میتوانند در تشخیص پزشکی برای شناسایی سلولهای بیمار مورد استفاده قرار گیرند. علاوه بر این شناسایی مولکولهای هدف آپتامرها یک روش جدید برای کشف مارکرهای زیستی است و میتواند برای دارورسانی و درمان هدفمندشده[44] مورد استفاده قرار گیرد. بدون شک با پیشرفت سریع نانوتکنولوژی، میکروچیپها، RNA مداخلهگر (RNAi) و سایر تکنولوژیهای پیشرفته، آپتامرها میتوانند کاربرد وسیعی در این تکنولوژیها به ویژه در درمان و تشخیص بیماریها داشته باشند.
منابع:
- Zhu, Z. (2012).Development of novel analytical tools based on nucleic acid aptamers for the detection of small molecules (Doctoral dissertation, Université de Grenoble).
- Song, K. M., Lee, S., & Ban, C. (2012). Aptamers and their biological applications.Sensors, 12(1), 612-631.
- Dollins, C. M., Nair, S., & Sullenger, B. A. (2008). Aptamers in immunotherapy.Human gene therapy,19(5), 443-450.
- Sullenger, B. A., Gallardo, H. F., Ungers, G. E., & Gilboa, E. (1990). Overexpression of TAR sequences renders cells resistant to human immunodeficiency virus replication.Cell, 63(3), 601-608.
- Han, K., Liang, Z., & Zhou, N. (2010). Design strategies for aptamer-based biosensors.Sensors, 10(5), 4541-4557.
- Tang, Z., Shangguan, D., Wang, K., Shi, H., Sefah, K., Mallikratchy, P., … & Tan, W. (2007). Selection of aptamers for molecular recognition and characterization of cancer cells.Analytical chemistry, 79(13), 4900-4907.
- Maehashi, K., Katsura, T., Kerman, K., Takamura, Y., Matsumoto, K., & Tamiya, E. (2007). Label-free protein biosensor based on aptamer-modified carbon nanotube field-effect transistors.Analytical Chemistry, 79(2), 782-787.
- Jenison, R. D., Gill, S. C., Pardi, A., & Polisky, B. (1994). High-resolution molecular discrimination by RNA.Science, 263(5152), 1425-1429.
- Song, K. M., Lee, S., & Ban, C. (2012). Aptamers and their biological applications.Sensors, 12(1), 612-631.
- Ferreira, C. S., & Missailidis, S. (2007). Aptamer-based therapeutics and their potential in radiopharmaceutical design.Brazilian archives of biology and technology, 50(SPE), 63-76.
- Jayasena, S. D. (1999). Aptamers: an emerging class of molecules that rival antibodies in diagnostics.Clinical chemistry, 45(9), 1628-1650.
- Ireson, C. R., & Kelland, L. R. (2006). Discovery and development of anticancer aptamers.Molecular cancer therapeutics, 5(12), 2957-2962.
- Tope, S., Maske, S., Nagulwar, V., Sufi, J., & Welankiwar, A. (2013). Aptamers as therapeutics.Indo American Journal of Pharmaceutical Research,3(3), 2718-2743.
- Mairal, T., Özalp, V. C., Sánchez, P. L., Mir, M., Katakis, I., & O’Sullivan, C. K. (2008). Aptamers: molecular tools for analytical applications.Analytical and bioanalytical chemistry, 390(4), 989-1007.
- Syed, M. A., & Pervaiz, S. (2010). Advances in aptamers.Oligonucleotides,20(5), 215-224.
- Irvine, D., Tuerk, C., & Gold, L. (1991). SELEXION: Systematic evolution of ligands by exponential enrichment with integrated optimization by non-linear analysis.Journal of molecular biology, 222(3), 739-761.
- Stoltenburg, R., Reinemann, C., & Strehlitz, B. (2007). SELEX—a (r) evolutionary method to generate high-affinity nucleic acid ligands.Biomolecular engineering, 24(4), 381-403.
- Tombelli, S., Minunni, M., & Mascini, M. (2005). Analytical applications of aptamers.Biosensors and Bioelectronics, 20(12), 2424-2434.
- Song, K. M., Cho, M., Jo, H., Min, K., Jeon, S. H., Kim, T., … & Ban, C. (2011). Gold nanoparticle-based colorimetric detection of kanamycin using a DNA aptamer.Analytical biochemistry, 415(2), 175-181.
- Wang, C., Yang, G., Luo, Z., & Ding, H. (2009). In vitro selection of high-affinity DNA aptamers for streptavidin. Acta biochimica et biophysica Sinica, 41(4), 335-340.
- Mendonsa, S. D., & Bowser, M. T. (2005). In vitro selection of aptamers with affinity for neuropeptide Y using capillary electrophoresis.Journal of the American Chemical Society, 127(26), 9382-9383.
- Mendonsa, S. D., & Bowser, M. T. (2004). In vitro selection of high-affinity DNA ligands for human IgE using capillary electrophoresis.Analytical chemistry, 76(18), 5387-5392.
- Jensen, K. B., Atkinson, B. L., Willis, M. C., Koch, T. H., & Gold, L. (1995). Using in vitro selection to direct the covalent attachment of human immunodeficiency virus type 1 Rev protein to high-affinity RNA ligands.Proceedings of the National Academy of Sciences,92(26), 12220-12224.
- Meyer, C., Hahn, U., & Rentmeister, A. (2011). Cell-specific aptamers as emerging therapeutics.Journal of nucleic acids, 2011.
- Mascini, M., Palchetti, I., & Tombelli, S. (2012). Nucleic acid and peptide aptamers: fundamentals and bioanalytical aspects.Angewandte Chemie International Edition, 51(6), 1316-1332.
- Crawford, M., Woodman, R., & Ferrigno, P. K. (2003). Peptide aptamers: tools for biology and drug discovery.Briefings in functional genomics & proteomics,2(1), 72-79.
- Orlova, A., Magnusson, M., Eriksson, T. L., Nilsson, M., Larsson, B., Höidén-Guthenberg, I., … & Nilsson, F. Y. (2006). Tumor imaging using a picomolar affinity HER2 binding affibody molecule.Cancer research, 66(8), 4339-4348.
- Fields, S., & Song, O. K. (1989). A novel genetic system to detect protein-protein interactions.Nature, (340), 245-6.
- Georgiou, G., Stephens, D. L., Stathopoulos, C., Poetschke, H. L., Mendenhall, J., & Earhart, C. F. (1996). Display of β-lactamase on the Escherichia coli surface: outer membrane phenotypes conferred by Lpp′–OmpA′–β-lactamase fusions.Protein engineering, 9(2), 239-247.
- Samuelson, P., Gunneriusson, E., Nygren, P. Å., & Ståhl, S. (2002). Display of proteins on bacteria.Journal of Biotechnology, 96(2), 129-154.
- Hu, X., Bessette, P. H., Qian, J., Meinhart, C. D., Daugherty, P. S., & Soh, H. T. (2005). Marker-specific sorting of rare cells using dielectrophoresis.Proceedings of the National Academy of Sciences of the United States of America,102(44), 15757-15761.
- Mascini, M., Palchetti, I., & Tombelli, S. (2012). Nucleic acid and peptide aptamers: fundamentals and bioanalytical aspects.Angewandte Chemie International Edition, 51(6), 1316-1332.
- Rich, R. L., & Myszka, D. G. (2006). Survey of the year 2005 commercial optical biosensor literature.Journal of Molecular Recognition, 19(6), 478-534.
- Lupold, S. E., Hicke, B. J., Lin, Y., & Coffey, D. S. (2002). Identification and characterization of nuclease-stabilized RNA molecules that bind human prostate cancer cells via the prostate-specific membrane antigen.Cancer research, 62(14), 4029-4033.
[1] Affinity
[2] Human immunodeficiency virus (HIV)
[3] trans-activation response (TAR)
[4] Systematic Evolution of Ligands by EXponential enrichment (SELEX)
[5] Hairpin
[6] Pseudoknot
[7] Quadruplexe
[8] dissociation constants
[9] re-annealing
[10] Absorption
[11] Recovery
[12] Amplification
[13] Library generation
[14] Random sequence region
[15] binding and separation
[16] Nitrocellulose Membrane Filtration-Based SELEX
[17] Affinity Chromatography and Magnetic Bead-Based SELEX
[18] Capillary Electrophoresis-Based SELEX
[19] frictional forces
[20] photoSELEX
[21] Photoactivatable
[22] photoactivation
[23] 5-bromo-2′-deoxyuridine (BrDU)
[24] Cell-SELEX
[25] Whole cell
[26] Scaffold
[27] E. coli
[28] Inert
[29] Doubly constrained
[30] Nondisply
[31] cell-dependent display
[32] cell-free display systems
[33] yeast two-hybrid (Y2H) system
[34] Macugen
[35] Angiogenesis
[36] Permeability
[37]AGE-RELATED MACULAR DEGREDATION (AMD)
[38] prostate-specific membrane antigen (PSMA)
[39] Doxorubicin
[40] Bio-imaging
[41] Fluorophore
[42] Gadolinium
[43] magnetic resonance imaging (MRI)
[44] targeted therapy
https://pubs.acs.org/doi/10.1021/acsami.0c05750
https://medlabnews.ir/%d8%a2%d9%be%d8%aa%d8%a7%d9%85%d8%b1/
برای دانلود پی دی اف بر روی لینک زیر کلیک کنید
ورود / ثبت نام